(primary solid tumor cohort)
This pipeline computes the correlation between significantly recurrent gene mutations and selected clinical features.
Testing the association between mutation status of 156 genes and 11 clinical features across 306 patients, 5 significant findings detected with Q value < 0.25.
-
TP53 mutation correlated to 'Time to Death'.
-
POTEC mutation correlated to 'STOPPEDSMOKINGYEAR'.
-
OR2M2 mutation correlated to 'STOPPEDSMOKINGYEAR'.
-
PLSCR4 mutation correlated to 'STOPPEDSMOKINGYEAR'.
-
GRID2 mutation correlated to 'STOPPEDSMOKINGYEAR'.
Table 1. Get Full Table Overview of the association between mutation status of 156 genes and 11 clinical features. Shown in the table are P values (Q values). Thresholded by Q value < 0.25, 5 significant findings detected.
|
Clinical Features |
Time to Death |
AGE | GENDER |
PATHOLOGY T |
PATHOLOGY N |
TUMOR STAGE |
RADIATIONS RADIATION REGIMENINDICATION |
NUMBERPACKYEARSSMOKED | STOPPEDSMOKINGYEAR | TOBACCOSMOKINGHISTORYINDICATOR | YEAROFTOBACCOSMOKINGONSET | ||
| nMutated (%) | nWild-Type | logrank test | t-test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | t-test | t-test | Fisher's exact test | t-test | |
| TP53 | 207 (68%) | 99 |
7.55e-05 (0.124) |
0.983 (1.00) |
0.414 (1.00) |
0.00232 (1.00) |
0.00935 (1.00) |
0.000812 (1.00) |
0.0496 (1.00) |
0.187 (1.00) |
0.59 (1.00) |
0.318 (1.00) |
0.47 (1.00) |
| POTEC | 15 (5%) | 291 |
0.229 (1.00) |
0.0491 (1.00) |
0.133 (1.00) |
0.212 (1.00) |
0.926 (1.00) |
0.636 (1.00) |
0.544 (1.00) |
0.232 (1.00) |
3.46e-05 (0.057) |
0.00819 (1.00) |
0.872 (1.00) |
| OR2M2 | 8 (3%) | 298 |
0.633 (1.00) |
0.384 (1.00) |
1 (1.00) |
0.521 (1.00) |
0.389 (1.00) |
0.554 (1.00) |
1 (1.00) |
0.0479 (1.00) |
1.19e-07 (0.000197) |
0.389 (1.00) |
0.801 (1.00) |
| PLSCR4 | 7 (2%) | 299 |
0.675 (1.00) |
0.441 (1.00) |
0.678 (1.00) |
0.159 (1.00) |
0.682 (1.00) |
0.712 (1.00) |
1 (1.00) |
0.85 (1.00) |
8.57e-05 (0.141) |
0.64 (1.00) |
0.488 (1.00) |
| GRID2 | 13 (4%) | 293 |
0.217 (1.00) |
0.372 (1.00) |
0.526 (1.00) |
0.212 (1.00) |
0.417 (1.00) |
0.74 (1.00) |
0.527 (1.00) |
0.371 (1.00) |
3.76e-05 (0.0619) |
0.0944 (1.00) |
0.32 (1.00) |
| NOTCH1 | 57 (19%) | 249 |
0.595 (1.00) |
0.129 (1.00) |
0.323 (1.00) |
0.753 (1.00) |
0.866 (1.00) |
0.948 (1.00) |
0.501 (1.00) |
0.00872 (1.00) |
0.459 (1.00) |
0.588 (1.00) |
0.813 (1.00) |
| CASP8 | 25 (8%) | 281 |
0.913 (1.00) |
0.000572 (0.937) |
0.00181 (1.00) |
0.513 (1.00) |
0.173 (1.00) |
0.149 (1.00) |
0.636 (1.00) |
0.223 (1.00) |
0.154 (1.00) |
0.024 (1.00) |
0.203 (1.00) |
| CDKN2A | 61 (20%) | 245 |
0.563 (1.00) |
0.561 (1.00) |
0.633 (1.00) |
0.487 (1.00) |
0.978 (1.00) |
0.152 (1.00) |
0.0478 (1.00) |
0.0881 (1.00) |
0.383 (1.00) |
0.441 (1.00) |
0.595 (1.00) |
| FAT1 | 72 (24%) | 234 |
0.794 (1.00) |
0.00051 (0.837) |
0.131 (1.00) |
0.537 (1.00) |
0.76 (1.00) |
0.499 (1.00) |
0.877 (1.00) |
0.53 (1.00) |
0.12 (1.00) |
0.0245 (1.00) |
0.133 (1.00) |
| PIK3CA | 63 (21%) | 243 |
0.197 (1.00) |
0.779 (1.00) |
0.154 (1.00) |
0.723 (1.00) |
0.392 (1.00) |
0.438 (1.00) |
1 (1.00) |
0.715 (1.00) |
0.354 (1.00) |
0.665 (1.00) |
0.845 (1.00) |
| JUB | 17 (6%) | 289 |
0.13 (1.00) |
0.157 (1.00) |
1 (1.00) |
0.51 (1.00) |
0.282 (1.00) |
0.812 (1.00) |
0.775 (1.00) |
0.538 (1.00) |
0.715 (1.00) |
0.186 (1.00) |
0.0655 (1.00) |
| MLL2 | 55 (18%) | 251 |
0.922 (1.00) |
0.978 (1.00) |
1 (1.00) |
0.266 (1.00) |
0.943 (1.00) |
0.485 (1.00) |
1 (1.00) |
0.316 (1.00) |
0.0481 (1.00) |
0.147 (1.00) |
0.393 (1.00) |
| NFE2L2 | 17 (6%) | 289 |
0.038 (1.00) |
0.00698 (1.00) |
0.576 (1.00) |
0.772 (1.00) |
0.884 (1.00) |
0.502 (1.00) |
0.256 (1.00) |
0.906 (1.00) |
0.636 (1.00) |
0.426 (1.00) |
0.309 (1.00) |
| BAGE2 | 11 (4%) | 295 |
0.0589 (1.00) |
0.26 (1.00) |
0.733 (1.00) |
0.678 (1.00) |
0.807 (1.00) |
0.609 (1.00) |
0.735 (1.00) |
0.966 (1.00) |
0.369 (1.00) |
0.645 (1.00) |
0.728 (1.00) |
| NSD1 | 33 (11%) | 273 |
0.194 (1.00) |
0.0819 (1.00) |
0.301 (1.00) |
0.778 (1.00) |
0.000562 (0.92) |
0.0362 (1.00) |
0.399 (1.00) |
0.218 (1.00) |
0.843 (1.00) |
0.297 (1.00) |
0.947 (1.00) |
| HRAS | 10 (3%) | 296 |
0.0791 (1.00) |
0.227 (1.00) |
1 (1.00) |
0.241 (1.00) |
0.0538 (1.00) |
0.0885 (1.00) |
0.461 (1.00) |
0.157 (1.00) |
0.924 (1.00) |
0.157 (1.00) |
0.217 (1.00) |
| B2M | 7 (2%) | 299 |
0.233 (1.00) |
0.483 (1.00) |
1 (1.00) |
0.61 (1.00) |
0.79 (1.00) |
0.683 (1.00) |
0.011 (1.00) |
||||
| POM121L12 | 14 (5%) | 292 |
0.649 (1.00) |
0.912 (1.00) |
1 (1.00) |
0.0736 (1.00) |
0.343 (1.00) |
0.444 (1.00) |
0.759 (1.00) |
0.427 (1.00) |
0.885 (1.00) |
0.245 (1.00) |
0.773 (1.00) |
| RAC1 | 9 (3%) | 297 |
0.676 (1.00) |
0.00682 (1.00) |
0.0676 (1.00) |
0.412 (1.00) |
0.215 (1.00) |
0.297 (1.00) |
1 (1.00) |
0.721 (1.00) |
0.303 (1.00) |
0.279 (1.00) |
0.00481 (1.00) |
| ZNF804B | 21 (7%) | 285 |
0.356 (1.00) |
0.054 (1.00) |
0.805 (1.00) |
0.952 (1.00) |
0.743 (1.00) |
0.662 (1.00) |
0.117 (1.00) |
0.0266 (1.00) |
0.0221 (1.00) |
0.336 (1.00) |
0.873 (1.00) |
| OR2T12 | 11 (4%) | 295 |
0.876 (1.00) |
0.0516 (1.00) |
1 (1.00) |
0.0325 (1.00) |
0.134 (1.00) |
0.00529 (1.00) |
0.301 (1.00) |
0.0379 (1.00) |
0.112 (1.00) |
0.0598 (1.00) |
0.201 (1.00) |
| CDH10 | 23 (8%) | 283 |
0.171 (1.00) |
0.0362 (1.00) |
0.336 (1.00) |
0.113 (1.00) |
0.797 (1.00) |
0.0701 (1.00) |
0.214 (1.00) |
0.098 (1.00) |
0.0722 (1.00) |
0.828 (1.00) |
0.153 (1.00) |
| EP300 | 25 (8%) | 281 |
0.642 (1.00) |
0.536 (1.00) |
0.641 (1.00) |
0.951 (1.00) |
0.586 (1.00) |
0.549 (1.00) |
0.636 (1.00) |
0.113 (1.00) |
0.17 (1.00) |
0.374 (1.00) |
0.73 (1.00) |
| KCNT2 | 17 (6%) | 289 |
0.409 (1.00) |
0.663 (1.00) |
0.787 (1.00) |
0.238 (1.00) |
0.817 (1.00) |
0.816 (1.00) |
0.256 (1.00) |
0.17 (1.00) |
0.153 (1.00) |
0.0804 (1.00) |
0.107 (1.00) |
| LCP1 | 12 (4%) | 294 |
0.654 (1.00) |
0.847 (1.00) |
0.522 (1.00) |
0.0309 (1.00) |
1 (1.00) |
0.0394 (1.00) |
0.51 (1.00) |
0.915 (1.00) |
0.222 (1.00) |
0.457 (1.00) |
0.7 (1.00) |
| RASA1 | 14 (5%) | 292 |
0.466 (1.00) |
0.783 (1.00) |
0.364 (1.00) |
0.521 (1.00) |
0.895 (1.00) |
0.843 (1.00) |
0.204 (1.00) |
0.968 (1.00) |
0.847 (1.00) |
0.855 (1.00) |
0.437 (1.00) |
| PRAMEF11 | 10 (3%) | 296 |
0.289 (1.00) |
0.0402 (1.00) |
0.145 (1.00) |
0.71 (1.00) |
0.686 (1.00) |
1 (1.00) |
0.719 (1.00) |
0.11 (1.00) |
0.678 (1.00) |
0.872 (1.00) |
0.0976 (1.00) |
| PRSS1 | 8 (3%) | 298 |
0.287 (1.00) |
0.751 (1.00) |
0.000603 (0.987) |
0.869 (1.00) |
0.125 (1.00) |
1 (1.00) |
0.21 (1.00) |
0.11 (1.00) |
0.0487 (1.00) |
0.67 (1.00) |
|
| FBXW7 | 13 (4%) | 293 |
0.817 (1.00) |
0.18 (1.00) |
0.355 (1.00) |
0.796 (1.00) |
0.31 (1.00) |
1 (1.00) |
0.328 (1.00) |
0.474 (1.00) |
0.334 (1.00) |
0.54 (1.00) |
0.373 (1.00) |
| PEG3 | 23 (8%) | 283 |
0.563 (1.00) |
0.983 (1.00) |
0.467 (1.00) |
0.956 (1.00) |
0.642 (1.00) |
0.718 (1.00) |
0.321 (1.00) |
0.14 (1.00) |
0.0248 (1.00) |
0.076 (1.00) |
0.801 (1.00) |
| REG1A | 8 (3%) | 298 |
0.458 (1.00) |
0.573 (1.00) |
1 (1.00) |
0.636 (1.00) |
1 (1.00) |
1 (1.00) |
0.425 (1.00) |
0.136 (1.00) |
0.06 (1.00) |
0.236 (1.00) |
0.581 (1.00) |
| STEAP4 | 10 (3%) | 296 |
0.634 (1.00) |
0.981 (1.00) |
0.295 (1.00) |
0.13 (1.00) |
1 (1.00) |
0.392 (1.00) |
1 (1.00) |
0.038 (1.00) |
0.084 (1.00) |
0.808 (1.00) |
0.363 (1.00) |
| ZNF99 | 22 (7%) | 284 |
0.284 (1.00) |
0.876 (1.00) |
0.145 (1.00) |
0.124 (1.00) |
0.717 (1.00) |
0.433 (1.00) |
0.803 (1.00) |
0.327 (1.00) |
0.000708 (1.00) |
0.196 (1.00) |
0.623 (1.00) |
| FCRL4 | 13 (4%) | 293 |
0.368 (1.00) |
0.223 (1.00) |
0.526 (1.00) |
0.254 (1.00) |
0.555 (1.00) |
0.481 (1.00) |
1 (1.00) |
0.109 (1.00) |
0.151 (1.00) |
0.711 (1.00) |
0.0883 (1.00) |
| LRFN5 | 13 (4%) | 293 |
0.893 (1.00) |
0.862 (1.00) |
1 (1.00) |
0.627 (1.00) |
0.0377 (1.00) |
0.878 (1.00) |
1 (1.00) |
0.719 (1.00) |
0.437 (1.00) |
0.0655 (1.00) |
0.457 (1.00) |
| EPHA2 | 14 (5%) | 292 |
0.0195 (1.00) |
0.891 (1.00) |
0.221 (1.00) |
0.932 (1.00) |
0.184 (1.00) |
0.805 (1.00) |
1 (1.00) |
0.00747 (1.00) |
0.523 (1.00) |
0.349 (1.00) |
0.0793 (1.00) |
| POTEG | 10 (3%) | 296 |
0.432 (1.00) |
0.0789 (1.00) |
1 (1.00) |
0.477 (1.00) |
0.89 (1.00) |
0.0959 (1.00) |
0.719 (1.00) |
0.31 (1.00) |
0.446 (1.00) |
0.0187 (1.00) |
|
| CPXCR1 | 7 (2%) | 299 |
0.535 (1.00) |
0.481 (1.00) |
1 (1.00) |
0.725 (1.00) |
0.807 (1.00) |
0.671 (1.00) |
0.0728 (1.00) |
0.276 (1.00) |
|||
| FAM155A | 11 (4%) | 295 |
0.667 (1.00) |
0.497 (1.00) |
0.3 (1.00) |
0.502 (1.00) |
0.269 (1.00) |
0.202 (1.00) |
0.48 (1.00) |
0.455 (1.00) |
0.0676 (1.00) |
0.198 (1.00) |
0.454 (1.00) |
| HLA-B | 9 (3%) | 297 |
0.636 (1.00) |
0.701 (1.00) |
0.0676 (1.00) |
0.913 (1.00) |
0.0257 (1.00) |
0.311 (1.00) |
0.118 (1.00) |
0.828 (1.00) |
0.504 (1.00) |
0.0922 (1.00) |
0.0829 (1.00) |
| OR4M2 | 8 (3%) | 298 |
0.375 (1.00) |
0.608 (1.00) |
0.689 (1.00) |
0.438 (1.00) |
1 (1.00) |
0.738 (1.00) |
0.685 (1.00) |
0.383 (1.00) |
0.741 (1.00) |
0.979 (1.00) |
|
| PABPC5 | 9 (3%) | 297 |
0.723 (1.00) |
0.717 (1.00) |
1 (1.00) |
0.214 (1.00) |
0.686 (1.00) |
0.199 (1.00) |
0.698 (1.00) |
0.294 (1.00) |
0.32 (1.00) |
0.174 (1.00) |
|
| TGFBR2 | 10 (3%) | 296 |
0.668 (1.00) |
0.0292 (1.00) |
1 (1.00) |
0.71 (1.00) |
0.336 (1.00) |
0.833 (1.00) |
0.07 (1.00) |
0.287 (1.00) |
0.329 (1.00) |
0.331 (1.00) |
0.00839 (1.00) |
| AGTR1 | 8 (3%) | 298 |
0.324 (1.00) |
0.0224 (1.00) |
0.453 (1.00) |
0.898 (1.00) |
0.408 (1.00) |
0.671 (1.00) |
0.685 (1.00) |
0.0866 (1.00) |
0.0199 (1.00) |
0.389 (1.00) |
0.0329 (1.00) |
| PSG8 | 9 (3%) | 297 |
0.983 (1.00) |
0.074 (1.00) |
0.121 (1.00) |
0.956 (1.00) |
0.251 (1.00) |
0.382 (1.00) |
1 (1.00) |
0.0223 (1.00) |
0.45 (1.00) |
0.928 (1.00) |
0.909 (1.00) |
| MAGEL2 | 12 (4%) | 294 |
0.477 (1.00) |
0.342 (1.00) |
1 (1.00) |
0.24 (1.00) |
0.0997 (1.00) |
0.0533 (1.00) |
0.308 (1.00) |
0.417 (1.00) |
0.486 (1.00) |
0.225 (1.00) |
0.554 (1.00) |
| HLA-A | 9 (3%) | 297 |
0.498 (1.00) |
0.262 (1.00) |
1 (1.00) |
0.62 (1.00) |
0.336 (1.00) |
0.274 (1.00) |
0.24 (1.00) |
0.709 (1.00) |
0.172 (1.00) |
0.00554 (1.00) |
0.273 (1.00) |
| PRIM2 | 9 (3%) | 297 |
0.652 (1.00) |
0.516 (1.00) |
1 (1.00) |
0.461 (1.00) |
0.229 (1.00) |
0.342 (1.00) |
0.457 (1.00) |
0.116 (1.00) |
0.369 (1.00) |
0.0677 (1.00) |
0.907 (1.00) |
| HIST1H4E | 5 (2%) | 301 |
0.851 (1.00) |
0.549 (1.00) |
0.618 (1.00) |
0.878 (1.00) |
1 (1.00) |
0.464 (1.00) |
0.334 (1.00) |
0.725 (1.00) |
|||
| LILRB1 | 12 (4%) | 294 |
0.885 (1.00) |
0.907 (1.00) |
0.522 (1.00) |
0.292 (1.00) |
0.52 (1.00) |
0.638 (1.00) |
1 (1.00) |
0.118 (1.00) |
0.869 (1.00) |
0.602 (1.00) |
0.57 (1.00) |
| OR56A1 | 8 (3%) | 298 |
0.866 (1.00) |
0.882 (1.00) |
0.689 (1.00) |
0.13 (1.00) |
0.89 (1.00) |
0.392 (1.00) |
1 (1.00) |
0.271 (1.00) |
0.647 (1.00) |
0.274 (1.00) |
0.917 (1.00) |
| PCDH11X | 21 (7%) | 285 |
0.724 (1.00) |
0.716 (1.00) |
0.00379 (1.00) |
0.134 (1.00) |
0.797 (1.00) |
0.971 (1.00) |
0.034 (1.00) |
0.125 (1.00) |
0.399 (1.00) |
0.403 (1.00) |
0.531 (1.00) |
| ZNF750 | 12 (4%) | 294 |
0.334 (1.00) |
0.844 (1.00) |
0.19 (1.00) |
0.0125 (1.00) |
0.509 (1.00) |
0.31 (1.00) |
0.737 (1.00) |
0.9 (1.00) |
0.781 (1.00) |
0.602 (1.00) |
0.318 (1.00) |
| GABRB3 | 11 (4%) | 295 |
0.128 (1.00) |
0.621 (1.00) |
0.0389 (1.00) |
0.284 (1.00) |
0.089 (1.00) |
0.247 (1.00) |
0.735 (1.00) |
0.238 (1.00) |
0.681 (1.00) |
0.973 (1.00) |
0.000266 (0.437) |
| SEMA5A | 19 (6%) | 287 |
0.636 (1.00) |
0.386 (1.00) |
0.606 (1.00) |
0.932 (1.00) |
0.248 (1.00) |
0.737 (1.00) |
1 (1.00) |
0.037 (1.00) |
0.00813 (1.00) |
0.0318 (1.00) |
0.559 (1.00) |
| LINGO2 | 10 (3%) | 296 |
0.412 (1.00) |
0.308 (1.00) |
0.295 (1.00) |
0.263 (1.00) |
1 (1.00) |
0.554 (1.00) |
0.131 (1.00) |
0.793 (1.00) |
0.982 (1.00) |
0.63 (1.00) |
0.729 (1.00) |
| DOK6 | 8 (3%) | 298 |
0.695 (1.00) |
0.348 (1.00) |
0.00634 (1.00) |
0.835 (1.00) |
0.125 (1.00) |
0.497 (1.00) |
0.685 (1.00) |
0.0744 (1.00) |
0.0219 (1.00) |
0.822 (1.00) |
|
| SLITRK4 | 12 (4%) | 294 |
0.115 (1.00) |
0.0417 (1.00) |
0.522 (1.00) |
0.767 (1.00) |
0.378 (1.00) |
0.951 (1.00) |
0.51 (1.00) |
0.211 (1.00) |
0.645 (1.00) |
0.859 (1.00) |
0.564 (1.00) |
| OR5D13 | 7 (2%) | 299 |
0.285 (1.00) |
0.209 (1.00) |
0.0183 (1.00) |
0.651 (1.00) |
1 (1.00) |
0.877 (1.00) |
1 (1.00) |
0.306 (1.00) |
0.0353 (1.00) |
0.575 (1.00) |
0.375 (1.00) |
| PTPRT | 19 (6%) | 287 |
0.798 (1.00) |
0.509 (1.00) |
0.606 (1.00) |
0.134 (1.00) |
0.294 (1.00) |
0.251 (1.00) |
0.277 (1.00) |
0.471 (1.00) |
0.125 (1.00) |
0.604 (1.00) |
0.671 (1.00) |
| FRG2B | 6 (2%) | 300 |
0.899 (1.00) |
0.378 (1.00) |
0.0503 (1.00) |
0.762 (1.00) |
0.887 (1.00) |
0.554 (1.00) |
1 (1.00) |
0.357 (1.00) |
0.228 (1.00) |
0.249 (1.00) |
|
| CNTNAP5 | 15 (5%) | 291 |
0.173 (1.00) |
0.589 (1.00) |
0.0769 (1.00) |
0.0291 (1.00) |
0.379 (1.00) |
0.0492 (1.00) |
0.768 (1.00) |
0.139 (1.00) |
0.0128 (1.00) |
0.173 (1.00) |
0.234 (1.00) |
| HIST1H2BD | 5 (2%) | 301 |
0.33 (1.00) |
0.973 (1.00) |
0.618 (1.00) |
0.323 (1.00) |
0.362 (1.00) |
0.663 (1.00) |
0.605 (1.00) |
0.33 (1.00) |
0.213 (1.00) |
||
| CTCF | 11 (4%) | 295 |
0.239 (1.00) |
0.0444 (1.00) |
0.181 (1.00) |
0.935 (1.00) |
0.606 (1.00) |
0.533 (1.00) |
1 (1.00) |
0.15 (1.00) |
0.853 (1.00) |
0.0319 (1.00) |
0.192 (1.00) |
| EYA1 | 10 (3%) | 296 |
0.811 (1.00) |
0.215 (1.00) |
0.145 (1.00) |
0.107 (1.00) |
0.408 (1.00) |
0.927 (1.00) |
0.719 (1.00) |
0.149 (1.00) |
0.982 (1.00) |
0.97 (1.00) |
0.432 (1.00) |
| PRB3 | 9 (3%) | 297 |
0.835 (1.00) |
0.558 (1.00) |
0.453 (1.00) |
0.123 (1.00) |
0.665 (1.00) |
0.0673 (1.00) |
1 (1.00) |
0.0738 (1.00) |
0.204 (1.00) |
0.149 (1.00) |
|
| ASXL3 | 21 (7%) | 285 |
0.494 (1.00) |
0.572 (1.00) |
0.456 (1.00) |
0.727 (1.00) |
0.459 (1.00) |
0.473 (1.00) |
0.303 (1.00) |
0.669 (1.00) |
0.625 (1.00) |
0.289 (1.00) |
0.609 (1.00) |
| ANKRD30B | 9 (3%) | 297 |
0.944 (1.00) |
0.926 (1.00) |
0.71 (1.00) |
0.653 (1.00) |
0.178 (1.00) |
0.494 (1.00) |
0.457 (1.00) |
0.778 (1.00) |
0.198 (1.00) |
0.907 (1.00) |
0.812 (1.00) |
| HFM1 | 15 (5%) | 291 |
0.261 (1.00) |
0.831 (1.00) |
1 (1.00) |
0.641 (1.00) |
0.257 (1.00) |
0.638 (1.00) |
0.544 (1.00) |
0.0864 (1.00) |
0.85 (1.00) |
0.84 (1.00) |
0.0651 (1.00) |
| HIST1H1B | 7 (2%) | 299 |
0.204 (1.00) |
0.0857 (1.00) |
0.398 (1.00) |
0.898 (1.00) |
0.15 (1.00) |
0.908 (1.00) |
0.683 (1.00) |
0.0554 (1.00) |
|||
| KEAP1 | 13 (4%) | 293 |
0.989 (1.00) |
0.503 (1.00) |
1 (1.00) |
0.0736 (1.00) |
0.327 (1.00) |
0.878 (1.00) |
0.102 (1.00) |
0.415 (1.00) |
0.000541 (0.887) |
0.464 (1.00) |
0.89 (1.00) |
| OR52E2 | 6 (2%) | 300 |
0.0765 (1.00) |
0.903 (1.00) |
1 (1.00) |
0.412 (1.00) |
0.733 (1.00) |
0.738 (1.00) |
0.175 (1.00) |
0.933 (1.00) |
0.868 (1.00) |
1 (1.00) |
0.609 (1.00) |
| OR8D4 | 6 (2%) | 300 |
0.224 (1.00) |
0.132 (1.00) |
0.193 (1.00) |
0.762 (1.00) |
0.428 (1.00) |
0.2 (1.00) |
1 (1.00) |
0.804 (1.00) |
0.0764 (1.00) |
0.237 (1.00) |
0.263 (1.00) |
| POTEH | 7 (2%) | 299 |
0.502 (1.00) |
0.0775 (1.00) |
0.678 (1.00) |
0.464 (1.00) |
0.033 (1.00) |
0.877 (1.00) |
1 (1.00) |
0.81 (1.00) |
0.271 (1.00) |
0.141 (1.00) |
|
| PRB1 | 7 (2%) | 299 |
0.558 (1.00) |
0.528 (1.00) |
0.196 (1.00) |
0.618 (1.00) |
0.408 (1.00) |
0.531 (1.00) |
0.377 (1.00) |
0.26 (1.00) |
0.0148 (1.00) |
0.366 (1.00) |
0.868 (1.00) |
| SORCS1 | 15 (5%) | 291 |
0.885 (1.00) |
0.704 (1.00) |
0.133 (1.00) |
0.624 (1.00) |
0.798 (1.00) |
0.338 (1.00) |
0.0276 (1.00) |
0.0775 (1.00) |
0.414 (1.00) |
0.5 (1.00) |
0.397 (1.00) |
| TMEM195 | 8 (3%) | 298 |
0.669 (1.00) |
0.0323 (1.00) |
1 (1.00) |
0.853 (1.00) |
1 (1.00) |
0.621 (1.00) |
1 (1.00) |
0.496 (1.00) |
0.0412 (1.00) |
0.204 (1.00) |
0.28 (1.00) |
| FIGN | 12 (4%) | 294 |
0.452 (1.00) |
0.479 (1.00) |
0.742 (1.00) |
0.313 (1.00) |
0.267 (1.00) |
0.0705 (1.00) |
0.308 (1.00) |
0.204 (1.00) |
0.00673 (1.00) |
0.0898 (1.00) |
0.745 (1.00) |
| PAPPA2 | 26 (8%) | 280 |
0.949 (1.00) |
0.654 (1.00) |
0.653 (1.00) |
0.922 (1.00) |
0.0562 (1.00) |
0.536 (1.00) |
0.345 (1.00) |
0.0511 (1.00) |
0.796 (1.00) |
0.0143 (1.00) |
0.274 (1.00) |
| DPPA4 | 7 (2%) | 299 |
0.529 (1.00) |
0.692 (1.00) |
1 (1.00) |
1 (1.00) |
0.138 (1.00) |
0.738 (1.00) |
0.377 (1.00) |
0.849 (1.00) |
0.917 (1.00) |
0.911 (1.00) |
0.661 (1.00) |
| OR2T11 | 7 (2%) | 299 |
0.401 (1.00) |
0.504 (1.00) |
1 (1.00) |
0.706 (1.00) |
1 (1.00) |
0.127 (1.00) |
0.377 (1.00) |
0.381 (1.00) |
0.541 (1.00) |
0.8 (1.00) |
0.475 (1.00) |
| TARP | 5 (2%) | 301 |
0.0509 (1.00) |
0.983 (1.00) |
0.0212 (1.00) |
0.785 (1.00) |
0.433 (1.00) |
0.85 (1.00) |
1 (1.00) |
0.609 (1.00) |
1 (1.00) |
0.882 (1.00) |
|
| LIN28B | 6 (2%) | 300 |
0.466 (1.00) |
0.928 (1.00) |
0.351 (1.00) |
1 (1.00) |
0.427 (1.00) |
0.647 (1.00) |
0.585 (1.00) |
0.671 (1.00) |
0.635 (1.00) |
||
| FOSL2 | 7 (2%) | 299 |
0.00662 (1.00) |
0.761 (1.00) |
0.398 (1.00) |
0.706 (1.00) |
0.682 (1.00) |
0.877 (1.00) |
1 (1.00) |
0.412 (1.00) |
0.813 (1.00) |
0.911 (1.00) |
0.986 (1.00) |
| C9ORF135 | 6 (2%) | 300 |
0.665 (1.00) |
0.855 (1.00) |
1 (1.00) |
0.0869 (1.00) |
0.0851 (1.00) |
0.097 (1.00) |
0.647 (1.00) |
0.723 (1.00) |
0.905 (1.00) |
0.638 (1.00) |
0.726 (1.00) |
| NEUROD6 | 7 (2%) | 299 |
0.685 (1.00) |
0.326 (1.00) |
1 (1.00) |
0.636 (1.00) |
1 (1.00) |
1 (1.00) |
0.0728 (1.00) |
0.394 (1.00) |
0.895 (1.00) |
0.911 (1.00) |
0.81 (1.00) |
| OR2L13 | 7 (2%) | 299 |
0.382 (1.00) |
0.392 (1.00) |
1 (1.00) |
0.137 (1.00) |
0.328 (1.00) |
0.216 (1.00) |
0.683 (1.00) |
0.652 (1.00) |
0.554 (1.00) |
0.341 (1.00) |
|
| C6 | 11 (4%) | 295 |
0.891 (1.00) |
0.166 (1.00) |
0.501 (1.00) |
0.925 (1.00) |
0.907 (1.00) |
0.741 (1.00) |
0.48 (1.00) |
0.172 (1.00) |
0.299 (1.00) |
0.681 (1.00) |
0.21 (1.00) |
| CUL3 | 10 (3%) | 296 |
0.77 (1.00) |
0.467 (1.00) |
0.733 (1.00) |
0.0875 (1.00) |
0.554 (1.00) |
0.289 (1.00) |
0.131 (1.00) |
0.527 (1.00) |
0.251 (1.00) |
0.195 (1.00) |
0.197 (1.00) |
| ADCY2 | 16 (5%) | 290 |
0.512 (1.00) |
0.0391 (1.00) |
0.391 (1.00) |
0.343 (1.00) |
0.884 (1.00) |
0.351 (1.00) |
0.135 (1.00) |
0.0566 (1.00) |
0.969 (1.00) |
0.954 (1.00) |
0.0925 (1.00) |
| HIST1H2BK | 5 (2%) | 301 |
0.397 (1.00) |
0.694 (1.00) |
0.618 (1.00) |
0.204 (1.00) |
0.182 (1.00) |
0.588 (1.00) |
1 (1.00) |
0.0823 (1.00) |
|||
| ZNF737 | 11 (4%) | 295 |
0.512 (1.00) |
0.872 (1.00) |
0.501 (1.00) |
0.606 (1.00) |
0.647 (1.00) |
0.533 (1.00) |
0.301 (1.00) |
0.391 (1.00) |
0.027 (1.00) |
0.132 (1.00) |
0.938 (1.00) |
| OR2J2 | 6 (2%) | 300 |
0.886 (1.00) |
0.807 (1.00) |
0.668 (1.00) |
0.553 (1.00) |
0.15 (1.00) |
0.672 (1.00) |
1 (1.00) |
0.256 (1.00) |
0.941 (1.00) |
0.838 (1.00) |
|
| NPY5R | 7 (2%) | 299 |
0.0612 (1.00) |
0.0123 (1.00) |
1 (1.00) |
0.706 (1.00) |
0.125 (1.00) |
0.663 (1.00) |
0.683 (1.00) |
0.718 (1.00) |
0.128 (1.00) |
0.00922 (1.00) |
|
| CD1E | 8 (3%) | 298 |
0.572 (1.00) |
0.0468 (1.00) |
0.222 (1.00) |
0.352 (1.00) |
0.549 (1.00) |
0.364 (1.00) |
0.685 (1.00) |
0.342 (1.00) |
0.961 (1.00) |
0.377 (1.00) |
0.115 (1.00) |
| F13B | 9 (3%) | 297 |
0.639 (1.00) |
0.567 (1.00) |
0.71 (1.00) |
0.365 (1.00) |
1 (1.00) |
0.178 (1.00) |
0.0502 (1.00) |
0.38 (1.00) |
0.45 (1.00) |
0.331 (1.00) |
0.747 (1.00) |
| OR4C11 | 6 (2%) | 300 |
0.588 (1.00) |
0.121 (1.00) |
0.668 (1.00) |
0.521 (1.00) |
1 (1.00) |
0.297 (1.00) |
1 (1.00) |
0.175 (1.00) |
0.419 (1.00) |
0.565 (1.00) |
|
| LRRC4C | 18 (6%) | 288 |
0.209 (1.00) |
0.717 (1.00) |
0.788 (1.00) |
0.219 (1.00) |
0.182 (1.00) |
0.772 (1.00) |
0.785 (1.00) |
0.637 (1.00) |
0.956 (1.00) |
0.42 (1.00) |
0.676 (1.00) |
| IL1RAPL1 | 13 (4%) | 293 |
0.0905 (1.00) |
0.367 (1.00) |
1 (1.00) |
0.265 (1.00) |
0.424 (1.00) |
0.333 (1.00) |
0.328 (1.00) |
0.118 (1.00) |
0.213 (1.00) |
0.0145 (1.00) |
0.746 (1.00) |
| PEX11A | 5 (2%) | 301 |
0.274 (1.00) |
0.088 (1.00) |
0.328 (1.00) |
0.0986 (1.00) |
0.558 (1.00) |
0.0667 (1.00) |
0.107 (1.00) |
0.757 (1.00) |
0.0989 (1.00) |
0.32 (1.00) |
0.147 (1.00) |
| SULF1 | 11 (4%) | 295 |
0.809 (1.00) |
0.237 (1.00) |
0.733 (1.00) |
0.293 (1.00) |
1 (1.00) |
0.385 (1.00) |
0.301 (1.00) |
0.825 (1.00) |
0.538 (1.00) |
0.641 (1.00) |
0.804 (1.00) |
| NECAB1 | 6 (2%) | 300 |
0.793 (1.00) |
0.00999 (1.00) |
0.351 (1.00) |
0.517 (1.00) |
1 (1.00) |
0.59 (1.00) |
1 (1.00) |
0.81 (1.00) |
0.539 (1.00) |
0.58 (1.00) |
0.0423 (1.00) |
| ITGB1 | 10 (3%) | 296 |
0.133 (1.00) |
0.144 (1.00) |
0.47 (1.00) |
0.508 (1.00) |
0.451 (1.00) |
0.225 (1.00) |
0.461 (1.00) |
0.742 (1.00) |
0.194 (1.00) |
0.356 (1.00) |
0.563 (1.00) |
| TRIM58 | 7 (2%) | 299 |
0.906 (1.00) |
0.0935 (1.00) |
0.678 (1.00) |
0.197 (1.00) |
0.172 (1.00) |
0.89 (1.00) |
1 (1.00) |
0.794 (1.00) |
0.457 (1.00) |
0.261 (1.00) |
|
| TAF10 | 4 (1%) | 302 |
0.0576 (1.00) |
0.641 (1.00) |
1 (1.00) |
0.84 (1.00) |
0.182 (1.00) |
0.6 (1.00) |
0.269 (1.00) |
0.504 (1.00) |
|||
| C7ORF57 | 6 (2%) | 300 |
0.039 (1.00) |
0.237 (1.00) |
0.193 (1.00) |
0.461 (1.00) |
0.809 (1.00) |
0.342 (1.00) |
1 (1.00) |
0.79 (1.00) |
0.783 (1.00) |
0.477 (1.00) |
0.0353 (1.00) |
| REG1B | 5 (2%) | 301 |
0.514 (1.00) |
0.652 (1.00) |
1 (1.00) |
0.412 (1.00) |
1 (1.00) |
0.287 (1.00) |
0.334 (1.00) |
0.5 (1.00) |
0.929 (1.00) |
0.552 (1.00) |
|
| GRXCR1 | 6 (2%) | 300 |
0.126 (1.00) |
0.664 (1.00) |
1 (1.00) |
0.161 (1.00) |
1 (1.00) |
0.367 (1.00) |
0.647 (1.00) |
0.369 (1.00) |
0.0252 (1.00) |
0.152 (1.00) |
|
| RGS17 | 5 (2%) | 301 |
0.529 (1.00) |
0.483 (1.00) |
0.618 (1.00) |
0.84 (1.00) |
0.79 (1.00) |
0.334 (1.00) |
0.946 (1.00) |
0.644 (1.00) |
|||
| TMEM132B | 14 (5%) | 292 |
0.626 (1.00) |
0.55 (1.00) |
0.123 (1.00) |
0.734 (1.00) |
0.366 (1.00) |
0.806 (1.00) |
0.531 (1.00) |
0.12 (1.00) |
0.0647 (1.00) |
0.43 (1.00) |
0.929 (1.00) |
| FAM184A | 12 (4%) | 294 |
0.231 (1.00) |
0.39 (1.00) |
0.522 (1.00) |
0.792 (1.00) |
0.291 (1.00) |
1 (1.00) |
0.51 (1.00) |
0.856 (1.00) |
0.159 (1.00) |
0.313 (1.00) |
0.996 (1.00) |
| FAM101A | 4 (1%) | 302 |
0.806 (1.00) |
0.937 (1.00) |
1 (1.00) |
0.61 (1.00) |
1 (1.00) |
0.79 (1.00) |
1 (1.00) |
0.33 (1.00) |
|||
| OR8I2 | 6 (2%) | 300 |
0.741 (1.00) |
0.579 (1.00) |
1 (1.00) |
0.636 (1.00) |
0.269 (1.00) |
0.712 (1.00) |
1 (1.00) |
0.806 (1.00) |
0.655 (1.00) |
0.929 (1.00) |
|
| USP29 | 11 (4%) | 295 |
0.553 (1.00) |
0.76 (1.00) |
0.501 (1.00) |
0.498 (1.00) |
0.378 (1.00) |
1 (1.00) |
0.156 (1.00) |
0.733 (1.00) |
0.831 (1.00) |
0.944 (1.00) |
0.573 (1.00) |
| OR4A16 | 6 (2%) | 300 |
0.299 (1.00) |
0.675 (1.00) |
1 (1.00) |
0.514 (1.00) |
0.682 (1.00) |
0.712 (1.00) |
0.647 (1.00) |
0.415 (1.00) |
0.832 (1.00) |
0.638 (1.00) |
0.964 (1.00) |
| CASP5 | 7 (2%) | 299 |
0.12 (1.00) |
0.252 (1.00) |
1 (1.00) |
0.853 (1.00) |
0.267 (1.00) |
0.455 (1.00) |
0.377 (1.00) |
0.989 (1.00) |
0.882 (1.00) |
0.291 (1.00) |
0.272 (1.00) |
| CYP4A11 | 8 (3%) | 298 |
0.184 (1.00) |
0.978 (1.00) |
0.453 (1.00) |
0.159 (1.00) |
1 (1.00) |
0.712 (1.00) |
0.118 (1.00) |
0.899 (1.00) |
0.604 (1.00) |
0.895 (1.00) |
0.993 (1.00) |
| KIF6 | 11 (4%) | 295 |
0.269 (1.00) |
0.986 (1.00) |
0.0389 (1.00) |
0.00281 (1.00) |
0.433 (1.00) |
0.119 (1.00) |
0.48 (1.00) |
0.876 (1.00) |
0.401 (1.00) |
0.524 (1.00) |
0.6 (1.00) |
| PPAN-P2RY11 | 7 (2%) | 299 |
0.731 (1.00) |
0.754 (1.00) |
0.196 (1.00) |
1 (1.00) |
0.389 (1.00) |
0.672 (1.00) |
0.377 (1.00) |
0.821 (1.00) |
1 (1.00) |
||
| DPPA2 | 9 (3%) | 297 |
0.376 (1.00) |
0.595 (1.00) |
0.71 (1.00) |
0.47 (1.00) |
0.89 (1.00) |
1 (1.00) |
0.457 (1.00) |
0.397 (1.00) |
0.474 (1.00) |
0.8 (1.00) |
0.214 (1.00) |
| MS4A14 | 9 (3%) | 297 |
0.998 (1.00) |
0.407 (1.00) |
0.0676 (1.00) |
0.382 (1.00) |
0.498 (1.00) |
0.364 (1.00) |
0.457 (1.00) |
0.795 (1.00) |
0.793 (1.00) |
0.624 (1.00) |
0.815 (1.00) |
| BRWD3 | 16 (5%) | 290 |
0.222 (1.00) |
0.683 (1.00) |
0.000486 (0.797) |
0.731 (1.00) |
0.386 (1.00) |
0.93 (1.00) |
0.769 (1.00) |
0.838 (1.00) |
0.587 (1.00) |
0.9 (1.00) |
0.989 (1.00) |
| HIST1H3E | 5 (2%) | 301 |
0.394 (1.00) |
0.539 (1.00) |
0.618 (1.00) |
0.382 (1.00) |
0.549 (1.00) |
0.877 (1.00) |
1 (1.00) |
0.101 (1.00) |
0.535 (1.00) |
0.27 (1.00) |
|
| KRT5 | 8 (3%) | 298 |
0.811 (1.00) |
0.299 (1.00) |
0.222 (1.00) |
0.084 (1.00) |
0.269 (1.00) |
0.032 (1.00) |
1 (1.00) |
0.149 (1.00) |
0.602 (1.00) |
0.377 (1.00) |
0.0519 (1.00) |
| OR4K15 | 6 (2%) | 300 |
0.521 (1.00) |
0.647 (1.00) |
0.668 (1.00) |
0.24 (1.00) |
0.267 (1.00) |
0.0806 (1.00) |
0.344 (1.00) |
0.472 (1.00) |
0.0625 (1.00) |
0.0451 (1.00) |
0.817 (1.00) |
| OR5H6 | 6 (2%) | 300 |
0.103 (1.00) |
0.652 (1.00) |
0.668 (1.00) |
1 (1.00) |
0.269 (1.00) |
0.712 (1.00) |
0.175 (1.00) |
0.0421 (1.00) |
0.85 (1.00) |
0.226 (1.00) |
|
| PSIP1 | 7 (2%) | 299 |
0.0437 (1.00) |
0.886 (1.00) |
0.678 (1.00) |
0.0262 (1.00) |
0.362 (1.00) |
0.269 (1.00) |
0.197 (1.00) |
0.66 (1.00) |
0.0406 (1.00) |
0.186 (1.00) |
0.788 (1.00) |
| SPANXN2 | 5 (2%) | 301 |
0.215 (1.00) |
0.604 (1.00) |
0.129 (1.00) |
0.878 (1.00) |
0.773 (1.00) |
0.464 (1.00) |
0.334 (1.00) |
0.17 (1.00) |
0.651 (1.00) |
||
| ZNF98 | 6 (2%) | 300 |
0.885 (1.00) |
0.321 (1.00) |
0.668 (1.00) |
1 (1.00) |
0.433 (1.00) |
0.85 (1.00) |
1 (1.00) |
0.528 (1.00) |
|||
| RSPO2 | 5 (2%) | 301 |
0.682 (1.00) |
0.239 (1.00) |
1 (1.00) |
0.56 (1.00) |
0.602 (1.00) |
0.227 (1.00) |
0.605 (1.00) |
0.0333 (1.00) |
0.917 (1.00) |
0.396 (1.00) |
0.107 (1.00) |
| TTC33 | 5 (2%) | 301 |
0.527 (1.00) |
0.93 (1.00) |
0.328 (1.00) |
0.159 (1.00) |
0.269 (1.00) |
0.712 (1.00) |
1 (1.00) |
0.241 (1.00) |
0.242 (1.00) |
0.396 (1.00) |
0.743 (1.00) |
| DLG2 | 11 (4%) | 295 |
0.233 (1.00) |
0.472 (1.00) |
0.181 (1.00) |
0.408 (1.00) |
0.327 (1.00) |
0.249 (1.00) |
0.48 (1.00) |
0.928 (1.00) |
0.631 (1.00) |
0.757 (1.00) |
0.0195 (1.00) |
| MAGEB3 | 6 (2%) | 300 |
0.316 (1.00) |
0.349 (1.00) |
0.351 (1.00) |
0.159 (1.00) |
0.11 (1.00) |
0.712 (1.00) |
1 (1.00) |
0.504 (1.00) |
0.772 (1.00) |
0.114 (1.00) |
|
| OR2G6 | 7 (2%) | 299 |
0.666 (1.00) |
0.407 (1.00) |
1 (1.00) |
0.0372 (1.00) |
0.558 (1.00) |
0.045 (1.00) |
0.377 (1.00) |
0.122 (1.00) |
0.201 (1.00) |
0.186 (1.00) |
0.542 (1.00) |
| KPRP | 8 (3%) | 298 |
0.283 (1.00) |
0.365 (1.00) |
0.112 (1.00) |
0.898 (1.00) |
0.0404 (1.00) |
0.908 (1.00) |
0.685 (1.00) |
0.912 (1.00) |
0.09 (1.00) |
0.0571 (1.00) |
0.491 (1.00) |
| OR2M5 | 6 (2%) | 300 |
0.446 (1.00) |
0.339 (1.00) |
1 (1.00) |
0.269 (1.00) |
1 (1.00) |
0.269 (1.00) |
0.175 (1.00) |
0.28 (1.00) |
0.401 (1.00) |
0.114 (1.00) |
|
| OR2M7 | 6 (2%) | 300 |
0.776 (1.00) |
0.793 (1.00) |
0.668 (1.00) |
0.713 (1.00) |
0.255 (1.00) |
1 (1.00) |
1 (1.00) |
0.935 (1.00) |
1 (1.00) |
0.843 (1.00) |
|
| WIPF1 | 8 (3%) | 298 |
0.851 (1.00) |
0.58 (1.00) |
0.689 (1.00) |
0.293 (1.00) |
1 (1.00) |
0.765 (1.00) |
0.425 (1.00) |
0.863 (1.00) |
0.722 (1.00) |
0.809 (1.00) |
|
| PAX5 | 7 (2%) | 299 |
0.119 (1.00) |
0.089 (1.00) |
1 (1.00) |
0.0869 (1.00) |
1 (1.00) |
0.0884 (1.00) |
1 (1.00) |
0.57 (1.00) |
0.562 (1.00) |
0.18 (1.00) |
0.0748 (1.00) |
| ZNF844 | 6 (2%) | 300 |
0.482 (1.00) |
0.432 (1.00) |
0.193 (1.00) |
0.56 (1.00) |
1 (1.00) |
0.414 (1.00) |
0.647 (1.00) |
0.81 (1.00) |
|||
| CTNND2 | 23 (8%) | 283 |
0.487 (1.00) |
0.754 (1.00) |
1 (1.00) |
0.417 (1.00) |
0.0207 (1.00) |
0.463 (1.00) |
0.46 (1.00) |
0.241 (1.00) |
0.016 (1.00) |
0.36 (1.00) |
0.987 (1.00) |
| HTR1E | 7 (2%) | 299 |
0.0901 (1.00) |
0.0673 (1.00) |
0.398 (1.00) |
0.814 (1.00) |
1 (1.00) |
0.244 (1.00) |
0.377 (1.00) |
0.399 (1.00) |
0.159 (1.00) |
0.0499 (1.00) |
|
| UGT2B11 | 7 (2%) | 299 |
0.286 (1.00) |
0.337 (1.00) |
0.678 (1.00) |
0.291 (1.00) |
0.549 (1.00) |
0.121 (1.00) |
1 (1.00) |
0.879 (1.00) |
0.554 (1.00) |
0.276 (1.00) |
|
| CSNK2A1 | 6 (2%) | 300 |
0.269 (1.00) |
0.674 (1.00) |
1 (1.00) |
0.323 (1.00) |
0.558 (1.00) |
0.414 (1.00) |
1 (1.00) |
0.611 (1.00) |
0.0746 (1.00) |
0.505 (1.00) |
0.786 (1.00) |
| ELMO1 | 10 (3%) | 296 |
0.955 (1.00) |
0.203 (1.00) |
0.295 (1.00) |
0.0125 (1.00) |
0.776 (1.00) |
0.31 (1.00) |
0.461 (1.00) |
0.916 (1.00) |
0.149 (1.00) |
0.246 (1.00) |
0.26 (1.00) |
| MYCT1 | 5 (2%) | 301 |
0.438 (1.00) |
0.885 (1.00) |
0.618 (1.00) |
0.0735 (1.00) |
0.549 (1.00) |
0.11 (1.00) |
1 (1.00) |
0.237 (1.00) |
0.869 (1.00) |
0.749 (1.00) |
|
| C3ORF59 | 8 (3%) | 298 |
0.887 (1.00) |
0.831 (1.00) |
0.689 (1.00) |
0.554 (1.00) |
0.122 (1.00) |
0.723 (1.00) |
1 (1.00) |
0.379 (1.00) |
0.832 (1.00) |
0.537 (1.00) |
0.429 (1.00) |
| NTM | 7 (2%) | 299 |
0.847 (1.00) |
0.87 (1.00) |
1 (1.00) |
0.56 (1.00) |
1 (1.00) |
0.098 (1.00) |
0.197 (1.00) |
0.823 (1.00) |
0.628 (1.00) |
0.173 (1.00) |
0.721 (1.00) |
| TEX13A | 7 (2%) | 299 |
0.856 (1.00) |
0.257 (1.00) |
0.0929 (1.00) |
0.911 (1.00) |
0.31 (1.00) |
1 (1.00) |
1 (1.00) |
0.513 (1.00) |
0.834 (1.00) |
0.319 (1.00) |
|
| OR5T3 | 6 (2%) | 300 |
0.00572 (1.00) |
0.972 (1.00) |
1 (1.00) |
0.0513 (1.00) |
0.172 (1.00) |
0.314 (1.00) |
1 (1.00) |
0.38 (1.00) |
0.25 (1.00) |
0.599 (1.00) |
|
| OR8J1 | 9 (3%) | 297 |
0.706 (1.00) |
0.32 (1.00) |
0.453 (1.00) |
1 (1.00) |
0.686 (1.00) |
0.927 (1.00) |
0.24 (1.00) |
0.0166 (1.00) |
0.000428 (0.704) |
0.374 (1.00) |
0.426 (1.00) |
| ZNF334 | 8 (3%) | 298 |
0.951 (1.00) |
0.687 (1.00) |
1 (1.00) |
0.871 (1.00) |
0.745 (1.00) |
0.382 (1.00) |
0.21 (1.00) |
0.923 (1.00) |
0.839 (1.00) |
0.33 (1.00) |
0.388 (1.00) |
| KIR3DL1 | 7 (2%) | 299 |
0.538 (1.00) |
0.767 (1.00) |
0.196 (1.00) |
0.00654 (1.00) |
0.267 (1.00) |
0.00893 (1.00) |
0.683 (1.00) |
0.228 (1.00) |
0.00394 (1.00) |
0.0651 (1.00) |
0.574 (1.00) |
| HDAC9 | 11 (4%) | 295 |
0.462 (1.00) |
0.771 (1.00) |
0.733 (1.00) |
0.0875 (1.00) |
0.807 (1.00) |
0.212 (1.00) |
1 (1.00) |
0.00389 (1.00) |
0.0117 (1.00) |
0.00538 (1.00) |
0.32 (1.00) |
| PEX1 | 12 (4%) | 294 |
0.576 (1.00) |
0.575 (1.00) |
0.742 (1.00) |
0.427 (1.00) |
0.287 (1.00) |
0.0756 (1.00) |
1 (1.00) |
0.864 (1.00) |
0.404 (1.00) |
0.338 (1.00) |
0.694 (1.00) |
| KCNA4 | 11 (4%) | 295 |
0.319 (1.00) |
0.486 (1.00) |
0.181 (1.00) |
0.71 (1.00) |
0.686 (1.00) |
0.364 (1.00) |
1 (1.00) |
0.344 (1.00) |
0.141 (1.00) |
0.613 (1.00) |
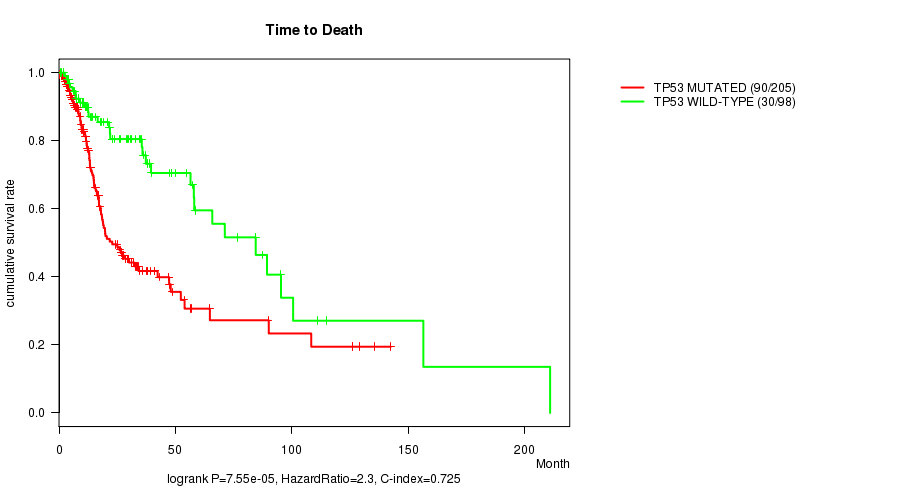
P value = 7.55e-05 (logrank test), Q value = 0.12
Table S1. Gene #2: 'TP53 MUTATION STATUS' versus Clinical Feature #1: 'Time to Death'
| nPatients | nDeath | Duration Range (Median), Month | |
|---|---|---|---|
| ALL | 303 | 120 | 0.1 - 210.9 (14.8) |
| TP53 MUTATED | 205 | 90 | 0.1 - 142.5 (13.2) |
| TP53 WILD-TYPE | 98 | 30 | 0.8 - 210.9 (21.9) |
Figure S1. Get High-res Image Gene #2: 'TP53 MUTATION STATUS' versus Clinical Feature #1: 'Time to Death'
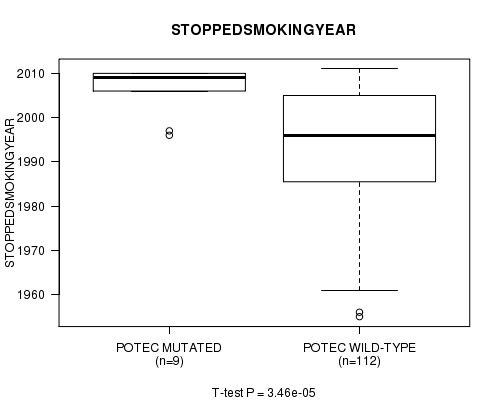
P value = 3.46e-05 (t-test), Q value = 0.057
Table S2. Gene #12: 'POTEC MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
| nPatients | Mean (Std.Dev) | |
|---|---|---|
| ALL | 121 | 1994.5 (13.8) |
| POTEC MUTATED | 9 | 2006.1 (5.6) |
| POTEC WILD-TYPE | 112 | 1993.6 (13.9) |
Figure S2. Get High-res Image Gene #12: 'POTEC MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
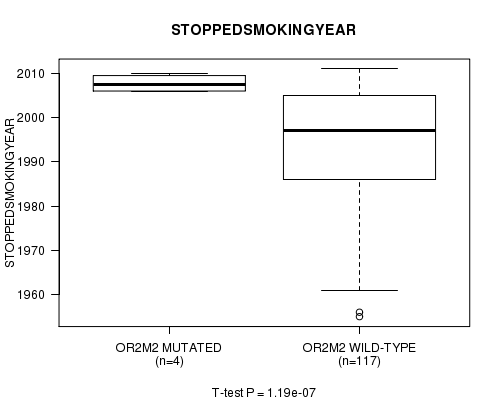
P value = 1.19e-07 (t-test), Q value = 2e-04
Table S3. Gene #38: 'OR2M2 MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
| nPatients | Mean (Std.Dev) | |
|---|---|---|
| ALL | 121 | 1994.5 (13.8) |
| OR2M2 MUTATED | 4 | 2007.8 (2.1) |
| OR2M2 WILD-TYPE | 117 | 1994.0 (13.8) |
Figure S3. Get High-res Image Gene #38: 'OR2M2 MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
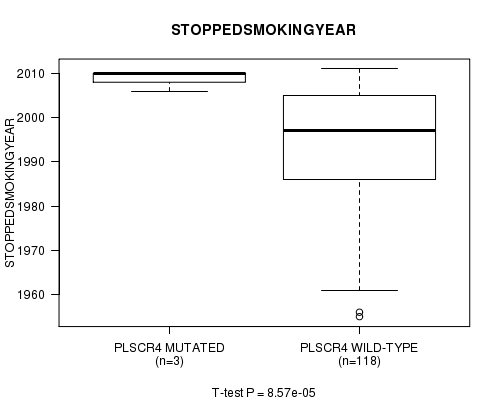
P value = 8.57e-05 (t-test), Q value = 0.14
Table S4. Gene #79: 'PLSCR4 MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
| nPatients | Mean (Std.Dev) | |
|---|---|---|
| ALL | 121 | 1994.5 (13.8) |
| PLSCR4 MUTATED | 3 | 2008.7 (2.3) |
| PLSCR4 WILD-TYPE | 118 | 1994.1 (13.8) |
Figure S4. Get High-res Image Gene #79: 'PLSCR4 MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
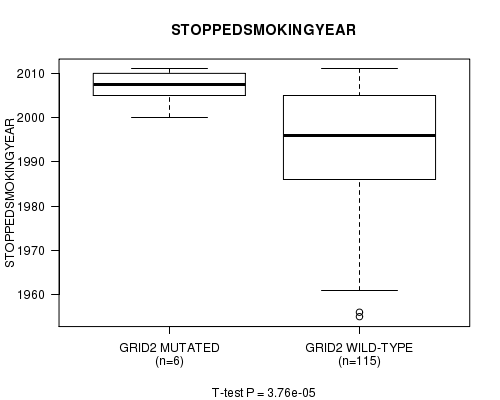
P value = 3.76e-05 (t-test), Q value = 0.062
Table S5. Gene #85: 'GRID2 MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
| nPatients | Mean (Std.Dev) | |
|---|---|---|
| ALL | 121 | 1994.5 (13.8) |
| GRID2 MUTATED | 6 | 2006.8 (4.1) |
| GRID2 WILD-TYPE | 115 | 1993.8 (13.9) |
Figure S5. Get High-res Image Gene #85: 'GRID2 MUTATION STATUS' versus Clinical Feature #9: 'STOPPEDSMOKINGYEAR'
-
Mutation data file = HNSC-TP.mutsig.cluster.txt
-
Clinical data file = HNSC-TP.clin.merged.picked.txt
-
Number of patients = 306
-
Number of significantly mutated genes = 156
-
Number of selected clinical features = 11
-
Exclude genes that fewer than K tumors have mutations, K = 3
For survival clinical features, the Kaplan-Meier survival curves of tumors with and without gene mutations were plotted and the statistical significance P values were estimated by logrank test (Bland and Altman 2004) using the 'survdiff' function in R
For continuous numerical clinical features, two-tailed Student's t test with unequal variance (Lehmann and Romano 2005) was applied to compare the clinical values between tumors with and without gene mutations using 't.test' function in R
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
This is an experimental feature. The full results of the analysis summarized in this report can be downloaded from the TCGA Data Coordination Center.