This pipeline computes the correlation between significantly recurrent gene mutations and molecular subtypes.
Testing the association between mutation status of 209 genes and 8 molecular subtypes across 224 patients, 20 significant findings detected with P value < 0.05 and Q value < 0.25.
-
FBXW7 mutation correlated to 'CN_CNMF'.
-
BRAF mutation correlated to 'MRNA_CNMF', 'MRNA_CHIERARCHICAL', and 'CN_CNMF'.
-
TP53 mutation correlated to 'CN_CNMF'.
-
FAT4 mutation correlated to 'CN_CNMF'.
-
CDH2 mutation correlated to 'MRNA_CNMF'.
-
FLG mutation correlated to 'MRNA_CNMF'.
-
DNAH5 mutation correlated to 'CN_CNMF'.
-
TMEM132D mutation correlated to 'MRNA_CNMF'.
-
ANK2 mutation correlated to 'MRNA_CHIERARCHICAL'.
-
ZC3H13 mutation correlated to 'MRNA_CHIERARCHICAL' and 'CN_CNMF'.
-
DOCK2 mutation correlated to 'MRNA_CNMF' and 'CN_CNMF'.
-
CDH10 mutation correlated to 'MRNA_CNMF' and 'CN_CNMF'.
-
SYNE1 mutation correlated to 'MRNA_CNMF', 'MRNA_CHIERARCHICAL', and 'CN_CNMF'.
Table 1. Get Full Table Overview of the association between mutation status of 209 genes and 8 molecular subtypes. Shown in the table are P values (Q values). Thresholded by P value < 0.05 and Q value < 0.25, 20 significant findings detected.
|
Clinical Features |
MRNA CNMF |
MRNA CHIERARCHICAL |
CN CNMF |
METHLYATION CNMF |
RPPA CNMF |
RPPA CHIERARCHICAL |
MIRSEQ CNMF |
MIRSEQ CHIERARCHICAL |
||
| nMutated (%) | nWild-Type | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | |
| BRAF | 22 (10%) | 202 |
1.37e-06 (0.00197) |
1.88e-07 (0.00027) |
4.17e-05 (0.0596) |
0.215 (1.00) |
0.304 (1.00) |
0.6 (1.00) |
1 (1.00) |
|
| SYNE1 | 47 (21%) | 177 |
3.76e-05 (0.0539) |
9.65e-06 (0.0138) |
1.63e-06 (0.00234) |
1 (1.00) |
0.569 (1.00) |
0.677 (1.00) |
0.367 (1.00) |
0.0326 (1.00) |
| ZC3H13 | 21 (9%) | 203 |
0.000463 (0.653) |
7.69e-05 (0.11) |
8.41e-05 (0.12) |
0.132 (1.00) |
0.0431 (1.00) |
1 (1.00) |
0.306 (1.00) |
|
| DOCK2 | 21 (9%) | 203 |
0.000159 (0.226) |
0.000416 (0.588) |
0.000138 (0.196) |
0.605 (1.00) |
0.631 (1.00) |
1 (1.00) |
0.101 (1.00) |
|
| CDH10 | 14 (6%) | 210 |
6.12e-05 (0.0874) |
0.00291 (1.00) |
0.000131 (0.186) |
0.533 (1.00) |
0.392 (1.00) |
1 (1.00) |
0.257 (1.00) |
|
| FBXW7 | 38 (17%) | 186 |
0.0217 (1.00) |
0.00968 (1.00) |
0.000122 (0.174) |
0.00881 (1.00) |
0.0362 (1.00) |
0.356 (1.00) |
0.118 (1.00) |
|
| TP53 | 120 (54%) | 104 |
0.000448 (0.633) |
0.00229 (1.00) |
7.28e-11 (1.05e-07) |
0.453 (1.00) |
0.782 (1.00) |
0.0252 (1.00) |
0.134 (1.00) |
|
| FAT4 | 39 (17%) | 185 |
0.0154 (1.00) |
0.117 (1.00) |
6.63e-05 (0.0946) |
0.701 (1.00) |
0.31 (1.00) |
0.635 (1.00) |
0.683 (1.00) |
|
| CDH2 | 16 (7%) | 208 |
3.86e-05 (0.0552) |
0.00587 (1.00) |
0.00381 (1.00) |
0.406 (1.00) |
0.322 (1.00) |
0.41 (1.00) |
0.683 (1.00) |
|
| FLG | 32 (14%) | 192 |
3.08e-06 (0.00441) |
0.00083 (1.00) |
0.00376 (1.00) |
0.233 (1.00) |
0.368 (1.00) |
1 (1.00) |
0.74 (1.00) |
|
| DNAH5 | 34 (15%) | 190 |
0.000239 (0.338) |
0.000915 (1.00) |
0.00013 (0.185) |
0.897 (1.00) |
0.391 (1.00) |
1 (1.00) |
0.238 (1.00) |
|
| TMEM132D | 19 (8%) | 205 |
3.34e-05 (0.0478) |
0.00169 (1.00) |
0.00293 (1.00) |
0.0581 (1.00) |
0.109 (1.00) |
1 (1.00) |
1 (1.00) |
|
| ANK2 | 30 (13%) | 194 |
0.000803 (1.00) |
7.61e-05 (0.108) |
0.0015 (1.00) |
0.00487 (1.00) |
0.0522 (1.00) |
1 (1.00) |
0.0416 (1.00) |
|
| NRAS | 20 (9%) | 204 |
0.0344 (1.00) |
0.241 (1.00) |
0.274 (1.00) |
0.247 (1.00) |
0.474 (1.00) |
1 (1.00) |
1 (1.00) |
|
| PIK3CA | 33 (15%) | 191 |
0.212 (1.00) |
0.783 (1.00) |
0.00048 (0.677) |
0.0876 (1.00) |
0.129 (1.00) |
0.0853 (1.00) |
1 (1.00) |
|
| APC | 160 (71%) | 64 |
0.0181 (1.00) |
0.00811 (1.00) |
0.221 (1.00) |
0.653 (1.00) |
0.452 (1.00) |
0.689 (1.00) |
0.464 (1.00) |
|
| KRAS | 96 (43%) | 128 |
0.000502 (0.706) |
0.0982 (1.00) |
0.485 (1.00) |
0.257 (1.00) |
0.634 (1.00) |
1 (1.00) |
0.0688 (1.00) |
0.735 (1.00) |
| SMAD4 | 26 (12%) | 198 |
0.0254 (1.00) |
0.334 (1.00) |
0.0355 (1.00) |
0.521 (1.00) |
0.339 (1.00) |
1 (1.00) |
0.846 (1.00) |
|
| FAM123B | 25 (11%) | 199 |
0.086 (1.00) |
0.0244 (1.00) |
0.00539 (1.00) |
0.536 (1.00) |
0.588 (1.00) |
1 (1.00) |
0.459 (1.00) |
|
| TTN | 85 (38%) | 139 |
0.00685 (1.00) |
0.0565 (1.00) |
0.00133 (1.00) |
0.657 (1.00) |
0.406 (1.00) |
0.941 (1.00) |
0.151 (1.00) |
0.927 (1.00) |
| SMAD2 | 15 (7%) | 209 |
0.123 (1.00) |
0.512 (1.00) |
0.0393 (1.00) |
0.533 (1.00) |
0.133 (1.00) |
1 (1.00) |
0.711 (1.00) |
|
| TCF7L2 | 18 (8%) | 206 |
1 (1.00) |
0.364 (1.00) |
0.417 (1.00) |
0.737 (1.00) |
0.896 (1.00) |
1 (1.00) |
0.23 (1.00) |
|
| KRTAP5-5 | 4 (2%) | 220 |
0.356 (1.00) |
1 (1.00) |
0.48 (1.00) |
0.388 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| ACVR1B | 14 (6%) | 210 |
0.119 (1.00) |
0.216 (1.00) |
0.0496 (1.00) |
0.336 (1.00) |
0.376 (1.00) |
0.435 (1.00) |
0.711 (1.00) |
|
| WBSCR17 | 19 (8%) | 205 |
0.103 (1.00) |
0.303 (1.00) |
0.141 (1.00) |
0.756 (1.00) |
0.35 (1.00) |
0.544 (1.00) |
0.618 (1.00) |
|
| PCDH9 | 22 (10%) | 202 |
0.844 (1.00) |
0.585 (1.00) |
0.0495 (1.00) |
0.262 (1.00) |
0.494 (1.00) |
0.563 (1.00) |
1 (1.00) |
|
| LRP1B | 39 (17%) | 185 |
0.00343 (1.00) |
0.00478 (1.00) |
0.00332 (1.00) |
0.161 (1.00) |
0.222 (1.00) |
1 (1.00) |
0.087 (1.00) |
|
| TNFRSF10C | 6 (3%) | 218 |
0.377 (1.00) |
1 (1.00) |
0.675 (1.00) |
0.853 (1.00) |
0.378 (1.00) |
1 (1.00) |
1 (1.00) |
|
| MAP2K4 | 11 (5%) | 213 |
0.0796 (1.00) |
0.49 (1.00) |
0.123 (1.00) |
0.688 (1.00) |
1 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| KIAA1804 | 15 (7%) | 209 |
0.237 (1.00) |
0.291 (1.00) |
0.0659 (1.00) |
0.758 (1.00) |
0.474 (1.00) |
1 (1.00) |
0.736 (1.00) |
|
| NRXN1 | 20 (9%) | 204 |
0.463 (1.00) |
0.322 (1.00) |
0.0843 (1.00) |
0.89 (1.00) |
0.524 (1.00) |
1 (1.00) |
0.13 (1.00) |
|
| KLHL4 | 16 (7%) | 208 |
0.0137 (1.00) |
0.0161 (1.00) |
0.00117 (1.00) |
0.756 (1.00) |
1 (1.00) |
0.41 (1.00) |
0.0126 (1.00) |
|
| SOX9 | 10 (4%) | 214 |
0.139 (1.00) |
0.585 (1.00) |
0.00451 (1.00) |
0.204 (1.00) |
0.694 (1.00) |
1 (1.00) |
0.584 (1.00) |
|
| PRDM9 | 16 (7%) | 208 |
0.00531 (1.00) |
0.87 (1.00) |
0.0701 (1.00) |
0.703 (1.00) |
1 (1.00) |
1 (1.00) |
0.255 (1.00) |
|
| PCBP1 | 6 (3%) | 218 |
0.187 (1.00) |
0.222 (1.00) |
0.396 (1.00) |
0.423 (1.00) |
0.482 (1.00) |
1 (1.00) |
1 (1.00) |
|
| DMD | 33 (15%) | 191 |
0.0202 (1.00) |
0.0205 (1.00) |
0.0052 (1.00) |
0.263 (1.00) |
0.309 (1.00) |
1 (1.00) |
0.156 (1.00) |
|
| CSMD1 | 26 (12%) | 198 |
0.16 (1.00) |
0.124 (1.00) |
0.0523 (1.00) |
1 (1.00) |
0.00982 (1.00) |
0.78 (1.00) |
0.0581 (1.00) |
0.142 (1.00) |
| LIFR | 18 (8%) | 206 |
0.119 (1.00) |
0.635 (1.00) |
0.219 (1.00) |
0.875 (1.00) |
0.699 (1.00) |
1 (1.00) |
0.064 (1.00) |
|
| OR6N1 | 10 (4%) | 214 |
0.00204 (1.00) |
0.127 (1.00) |
0.013 (1.00) |
1 (1.00) |
0.792 (1.00) |
0.304 (1.00) |
1 (1.00) |
|
| CCDC160 | 7 (3%) | 217 |
0.0688 (1.00) |
0.477 (1.00) |
0.108 (1.00) |
0.204 (1.00) |
0.694 (1.00) |
1 (1.00) |
0.0575 (1.00) |
|
| CNTN6 | 17 (8%) | 207 |
0.162 (1.00) |
0.341 (1.00) |
0.0782 (1.00) |
0.602 (1.00) |
0.35 (1.00) |
1 (1.00) |
0.322 (1.00) |
|
| CPXCR1 | 9 (4%) | 215 |
0.00296 (1.00) |
0.153 (1.00) |
0.191 (1.00) |
0.0909 (1.00) |
0.697 (1.00) |
0.304 (1.00) |
0.0285 (1.00) |
|
| SLITRK1 | 15 (7%) | 209 |
0.517 (1.00) |
0.879 (1.00) |
0.159 (1.00) |
0.554 (1.00) |
0.88 (1.00) |
1 (1.00) |
0.181 (1.00) |
|
| CNBD1 | 7 (3%) | 217 |
0.768 (1.00) |
0.688 (1.00) |
0.293 (1.00) |
1 (1.00) |
0.406 (1.00) |
|||
| CTNNB1 | 11 (5%) | 213 |
0.672 (1.00) |
0.75 (1.00) |
0.048 (1.00) |
0.296 (1.00) |
0.282 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| DKK2 | 6 (3%) | 218 |
0.0358 (1.00) |
0.0109 (1.00) |
0.628 (1.00) |
0.158 (1.00) |
0.876 (1.00) |
1 (1.00) |
1 (1.00) |
|
| ERBB4 | 18 (8%) | 206 |
0.121 (1.00) |
0.553 (1.00) |
0.00631 (1.00) |
0.0969 (1.00) |
0.211 (1.00) |
0.481 (1.00) |
0.76 (1.00) |
|
| ARID1A | 21 (9%) | 203 |
0.0239 (1.00) |
0.102 (1.00) |
0.0136 (1.00) |
0.857 (1.00) |
0.702 (1.00) |
1 (1.00) |
0.064 (1.00) |
|
| KIAA1486 | 13 (6%) | 211 |
0.361 (1.00) |
0.915 (1.00) |
0.306 (1.00) |
0.235 (1.00) |
0.439 (1.00) |
1 (1.00) |
0.0638 (1.00) |
|
| ROBO1 | 18 (8%) | 206 |
0.0053 (1.00) |
0.00861 (1.00) |
0.0221 (1.00) |
0.0261 (1.00) |
0.264 (1.00) |
0.502 (1.00) |
0.281 (1.00) |
|
| ZFHX4 | 24 (11%) | 200 |
0.72 (1.00) |
0.332 (1.00) |
0.0166 (1.00) |
0.448 (1.00) |
0.265 (1.00) |
1 (1.00) |
0.072 (1.00) |
|
| EPHA3 | 16 (7%) | 208 |
0.000482 (0.678) |
0.00405 (1.00) |
0.214 (1.00) |
0.581 (1.00) |
0.711 (1.00) |
1 (1.00) |
0.23 (1.00) |
|
| PRIM2 | 8 (4%) | 216 |
0.277 (1.00) |
0.779 (1.00) |
0.889 (1.00) |
0.564 (1.00) |
0.37 (1.00) |
1 (1.00) |
0.406 (1.00) |
|
| UBE2NL | 7 (3%) | 217 |
0.108 (1.00) |
0.446 (1.00) |
0.402 (1.00) |
0.448 (1.00) |
0.694 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| ACVR2A | 9 (4%) | 215 |
0.00858 (1.00) |
0.443 (1.00) |
0.0325 (1.00) |
0.429 (1.00) |
0.469 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| ADAM29 | 11 (5%) | 213 |
0.451 (1.00) |
1 (1.00) |
0.447 (1.00) |
0.0901 (1.00) |
0.436 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| ATP10A | 19 (8%) | 205 |
0.0197 (1.00) |
0.101 (1.00) |
0.141 (1.00) |
0.00172 (1.00) |
0.0188 (1.00) |
1 (1.00) |
0.386 (1.00) |
|
| GABRA5 | 9 (4%) | 215 |
0.487 (1.00) |
0.342 (1.00) |
0.756 (1.00) |
0.0425 (1.00) |
0.375 (1.00) |
0.244 (1.00) |
0.456 (1.00) |
|
| NALCN | 20 (9%) | 204 |
0.0419 (1.00) |
0.101 (1.00) |
0.141 (1.00) |
0.567 (1.00) |
0.881 (1.00) |
1 (1.00) |
0.053 (1.00) |
|
| OTOL1 | 8 (4%) | 216 |
0.132 (1.00) |
1 (1.00) |
0.943 (1.00) |
0.241 (1.00) |
0.876 (1.00) |
0.274 (1.00) |
0.502 (1.00) |
|
| PTEN | 7 (3%) | 217 |
0.0222 (1.00) |
0.218 (1.00) |
0.0429 (1.00) |
0.612 (1.00) |
0.436 (1.00) |
0.244 (1.00) |
0.0575 (1.00) |
|
| SDK1 | 27 (12%) | 197 |
0.0106 (1.00) |
0.000738 (1.00) |
0.24 (1.00) |
0.961 (1.00) |
0.425 (1.00) |
0.6 (1.00) |
1 (1.00) |
|
| TLL1 | 15 (7%) | 209 |
0.0358 (1.00) |
0.631 (1.00) |
0.02 (1.00) |
0.184 (1.00) |
0.189 (1.00) |
0.359 (1.00) |
0.00952 (1.00) |
|
| TRPC4 | 17 (8%) | 207 |
0.0053 (1.00) |
0.0217 (1.00) |
0.0139 (1.00) |
0.575 (1.00) |
0.149 (1.00) |
0.481 (1.00) |
0.171 (1.00) |
|
| ATM | 25 (11%) | 199 |
0.00324 (1.00) |
0.0867 (1.00) |
0.000415 (0.587) |
0.293 (1.00) |
0.459 (1.00) |
0.6 (1.00) |
0.162 (1.00) |
|
| GPC6 | 10 (4%) | 214 |
0.569 (1.00) |
0.316 (1.00) |
0.509 (1.00) |
0.524 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| CDH8 | 14 (6%) | 210 |
0.00127 (1.00) |
0.000535 (0.753) |
0.124 (1.00) |
0.0768 (1.00) |
0.089 (1.00) |
1 (1.00) |
1 (1.00) |
|
| MGC26647 | 7 (3%) | 217 |
0.287 (1.00) |
0.872 (1.00) |
0.0498 (1.00) |
1 (1.00) |
0.518 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| DKK4 | 7 (3%) | 217 |
0.299 (1.00) |
0.125 (1.00) |
0.402 (1.00) |
0.226 (1.00) |
0.876 (1.00) |
1 (1.00) |
1 (1.00) |
|
| PTPRM | 20 (9%) | 204 |
0.0483 (1.00) |
0.00861 (1.00) |
0.00293 (1.00) |
0.611 (1.00) |
0.254 (1.00) |
0.523 (1.00) |
0.0381 (1.00) |
|
| AFF2 | 17 (8%) | 207 |
0.0512 (1.00) |
0.0541 (1.00) |
0.201 (1.00) |
0.672 (1.00) |
0.116 (1.00) |
0.481 (1.00) |
0.255 (1.00) |
|
| NCAM2 | 12 (5%) | 212 |
0.0115 (1.00) |
0.313 (1.00) |
0.0617 (1.00) |
0.18 (1.00) |
0.123 (1.00) |
1 (1.00) |
0.0387 (1.00) |
|
| OR2M4 | 8 (4%) | 216 |
0.0102 (1.00) |
0.254 (1.00) |
0.0382 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.502 (1.00) |
|
| RYR2 | 29 (13%) | 195 |
0.041 (1.00) |
0.0914 (1.00) |
0.0195 (1.00) |
0.036 (1.00) |
0.149 (1.00) |
0.6 (1.00) |
0.0755 (1.00) |
|
| CDH9 | 15 (7%) | 209 |
0.135 (1.00) |
0.137 (1.00) |
0.0226 (1.00) |
0.0912 (1.00) |
0.035 (1.00) |
1 (1.00) |
0.158 (1.00) |
|
| GRIK3 | 17 (8%) | 207 |
0.00835 (1.00) |
0.00963 (1.00) |
0.196 (1.00) |
0.256 (1.00) |
0.195 (1.00) |
1 (1.00) |
0.711 (1.00) |
|
| ELF3 | 6 (3%) | 218 |
0.328 (1.00) |
0.125 (1.00) |
0.0514 (1.00) |
0.612 (1.00) |
0.436 (1.00) |
1 (1.00) |
0.406 (1.00) |
|
| TRPS1 | 17 (8%) | 207 |
0.01 (1.00) |
0.0792 (1.00) |
0.00133 (1.00) |
0.878 (1.00) |
0.474 (1.00) |
0.458 (1.00) |
0.354 (1.00) |
|
| FAM133A | 7 (3%) | 217 |
0.333 (1.00) |
0.872 (1.00) |
0.0183 (1.00) |
0.341 (1.00) |
0.599 (1.00) |
1 (1.00) |
0.456 (1.00) |
|
| IL1RAPL2 | 10 (4%) | 214 |
0.356 (1.00) |
0.75 (1.00) |
0.0473 (1.00) |
0.07 (1.00) |
0.234 (1.00) |
0.0412 (1.00) |
1 (1.00) |
|
| CCBP2 | 10 (4%) | 214 |
0.154 (1.00) |
0.316 (1.00) |
0.623 (1.00) |
0.593 (1.00) |
0.439 (1.00) |
1 (1.00) |
0.162 (1.00) |
|
| OPCML | 10 (4%) | 214 |
0.335 (1.00) |
0.347 (1.00) |
0.543 (1.00) |
0.821 (1.00) |
0.109 (1.00) |
1 (1.00) |
0.0935 (1.00) |
|
| CDH18 | 13 (6%) | 211 |
0.00184 (1.00) |
0.313 (1.00) |
0.0984 (1.00) |
0.363 (1.00) |
0.376 (1.00) |
1 (1.00) |
0.62 (1.00) |
|
| OR2L13 | 10 (4%) | 214 |
0.165 (1.00) |
0.443 (1.00) |
0.0275 (1.00) |
0.245 (1.00) |
0.821 (1.00) |
1 (1.00) |
1 (1.00) |
|
| LRRN3 | 10 (4%) | 214 |
0.00253 (1.00) |
0.0952 (1.00) |
0.446 (1.00) |
0.4 (1.00) |
0.735 (1.00) |
0.274 (1.00) |
0.502 (1.00) |
|
| BAI3 | 18 (8%) | 206 |
0.00782 (1.00) |
0.206 (1.00) |
0.423 (1.00) |
0.629 (1.00) |
0.548 (1.00) |
1 (1.00) |
0.255 (1.00) |
|
| MGC42105 | 11 (5%) | 213 |
0.00161 (1.00) |
0.197 (1.00) |
0.147 (1.00) |
0.564 (1.00) |
0.0345 (1.00) |
1 (1.00) |
1 (1.00) |
|
| TXNDC3 | 11 (5%) | 213 |
0.00292 (1.00) |
0.0485 (1.00) |
0.0125 (1.00) |
0.351 (1.00) |
0.0693 (1.00) |
1 (1.00) |
0.00311 (1.00) |
|
| LPPR4 | 13 (6%) | 211 |
0.0835 (1.00) |
0.00634 (1.00) |
0.0342 (1.00) |
0.221 (1.00) |
0.469 (1.00) |
1 (1.00) |
0.653 (1.00) |
|
| UNC13C | 17 (8%) | 207 |
0.00812 (1.00) |
0.137 (1.00) |
0.0257 (1.00) |
0.297 (1.00) |
0.896 (1.00) |
1 (1.00) |
0.255 (1.00) |
|
| SCN7A | 15 (7%) | 209 |
0.000715 (1.00) |
0.0293 (1.00) |
0.0492 (1.00) |
0.156 (1.00) |
0.88 (1.00) |
1 (1.00) |
0.205 (1.00) |
|
| ACOT4 | 3 (1%) | 221 |
0.298 (1.00) |
0.288 (1.00) |
0.829 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| GGT1 | 3 (1%) | 221 |
0.254 (1.00) |
1 (1.00) |
1 (1.00) |
|||||
| GRIA1 | 15 (7%) | 209 |
0.0584 (1.00) |
0.31 (1.00) |
0.0104 (1.00) |
0.104 (1.00) |
0.392 (1.00) |
1 (1.00) |
0.0226 (1.00) |
|
| ING1 | 6 (3%) | 218 |
0.294 (1.00) |
0.71 (1.00) |
0.628 (1.00) |
1 (1.00) |
0.694 (1.00) |
1 (1.00) |
0.406 (1.00) |
|
| PCDHA10 | 10 (4%) | 214 |
0.0819 (1.00) |
0.258 (1.00) |
0.108 (1.00) |
0.106 (1.00) |
0.0355 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| ZNF208 | 11 (5%) | 213 |
0.186 (1.00) |
0.915 (1.00) |
0.249 (1.00) |
0.204 (1.00) |
0.274 (1.00) |
1 (1.00) |
0.114 (1.00) |
|
| CRTC1 | 6 (3%) | 218 |
0.123 (1.00) |
0.0422 (1.00) |
0.837 (1.00) |
0.209 (1.00) |
0.482 (1.00) |
1 (1.00) |
0.406 (1.00) |
|
| FAM5C | 13 (6%) | 211 |
0.459 (1.00) |
0.712 (1.00) |
0.0186 (1.00) |
0.0151 (1.00) |
0.0123 (1.00) |
0.0711 (1.00) |
0.653 (1.00) |
|
| GUCY1A3 | 12 (5%) | 212 |
0.395 (1.00) |
0.197 (1.00) |
0.0311 (1.00) |
0.564 (1.00) |
0.469 (1.00) |
1 (1.00) |
0.584 (1.00) |
|
| FAM22F | 9 (4%) | 215 |
0.691 (1.00) |
0.731 (1.00) |
1 (1.00) |
0.342 (1.00) |
0.37 (1.00) |
1 (1.00) |
1 (1.00) |
|
| CHRDL1 | 11 (5%) | 213 |
0.168 (1.00) |
0.648 (1.00) |
0.472 (1.00) |
0.255 (1.00) |
0.439 (1.00) |
1 (1.00) |
0.584 (1.00) |
|
| EDNRB | 10 (4%) | 214 |
0.0668 (1.00) |
0.912 (1.00) |
0.757 (1.00) |
0.0226 (1.00) |
0.539 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| IFT80 | 10 (4%) | 214 |
0.0592 (1.00) |
0.153 (1.00) |
0.254 (1.00) |
0.419 (1.00) |
0.375 (1.00) |
0.304 (1.00) |
0.162 (1.00) |
|
| CASP14 | 8 (4%) | 216 |
1 (1.00) |
0.333 (1.00) |
0.889 (1.00) |
1 (1.00) |
1 (1.00) |
0.274 (1.00) |
1 (1.00) |
|
| LASS3 | 9 (4%) | 215 |
0.00364 (1.00) |
0.182 (1.00) |
0.104 (1.00) |
0.18 (1.00) |
0.792 (1.00) |
1 (1.00) |
0.0747 (1.00) |
|
| P2RY10 | 7 (3%) | 217 |
0.661 (1.00) |
0.382 (1.00) |
0.402 (1.00) |
0.204 (1.00) |
0.694 (1.00) |
1 (1.00) |
1 (1.00) |
|
| CNTN1 | 14 (6%) | 210 |
0.0177 (1.00) |
0.0609 (1.00) |
0.00405 (1.00) |
0.358 (1.00) |
0.865 (1.00) |
1 (1.00) |
0.181 (1.00) |
|
| ISL1 | 8 (4%) | 216 |
0.304 (1.00) |
0.443 (1.00) |
0.435 (1.00) |
1 (1.00) |
0.647 (1.00) |
1 (1.00) |
0.502 (1.00) |
|
| ARID2 | 15 (7%) | 209 |
0.161 (1.00) |
0.132 (1.00) |
0.0254 (1.00) |
0.16 (1.00) |
0.3 (1.00) |
1 (1.00) |
0.0638 (1.00) |
|
| EIF4A2 | 5 (2%) | 219 |
0.548 (1.00) |
0.512 (1.00) |
0.73 (1.00) |
0.439 (1.00) |
0.599 (1.00) |
1 (1.00) |
0.141 (1.00) |
|
| OR51V1 | 9 (4%) | 215 |
0.00273 (1.00) |
0.00886 (1.00) |
0.0016 (1.00) |
0.414 (1.00) |
0.458 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| CDH11 | 14 (6%) | 210 |
0.0683 (1.00) |
0.247 (1.00) |
0.0175 (1.00) |
0.296 (1.00) |
0.201 (1.00) |
0.385 (1.00) |
1 (1.00) |
|
| CHST9 | 8 (4%) | 216 |
0.0121 (1.00) |
0.443 (1.00) |
0.0706 (1.00) |
0.218 (1.00) |
0.019 (1.00) |
1 (1.00) |
0.456 (1.00) |
|
| HTR3B | 6 (3%) | 218 |
0.131 (1.00) |
0.736 (1.00) |
0.628 (1.00) |
0.0191 (1.00) |
0.149 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| LRRIQ3 | 10 (4%) | 214 |
0.176 (1.00) |
0.231 (1.00) |
0.157 (1.00) |
0.419 (1.00) |
0.375 (1.00) |
1 (1.00) |
0.114 (1.00) |
|
| HCN1 | 10 (4%) | 214 |
0.287 (1.00) |
0.382 (1.00) |
0.0201 (1.00) |
0.699 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| CHRM2 | 10 (4%) | 214 |
0.0935 (1.00) |
0.443 (1.00) |
0.115 (1.00) |
0.641 (1.00) |
0.539 (1.00) |
1 (1.00) |
0.545 (1.00) |
|
| CSMD3 | 28 (12%) | 196 |
0.0072 (1.00) |
0.00467 (1.00) |
0.00457 (1.00) |
0.106 (1.00) |
0.0552 (1.00) |
0.6 (1.00) |
0.0587 (1.00) |
|
| DCAF4L2 | 11 (5%) | 213 |
0.00364 (1.00) |
0.00609 (1.00) |
0.027 (1.00) |
0.419 (1.00) |
0.234 (1.00) |
0.304 (1.00) |
0.545 (1.00) |
|
| DACH2 | 12 (5%) | 212 |
0.134 (1.00) |
0.29 (1.00) |
0.256 (1.00) |
0.448 (1.00) |
0.149 (1.00) |
1 (1.00) |
0.114 (1.00) |
|
| PCDH11X | 14 (6%) | 210 |
0.0194 (1.00) |
0.512 (1.00) |
0.107 (1.00) |
0.07 (1.00) |
0.375 (1.00) |
1 (1.00) |
0.181 (1.00) |
|
| KCNA4 | 12 (5%) | 212 |
0.113 (1.00) |
0.0186 (1.00) |
0.00254 (1.00) |
0.0198 (1.00) |
0.0585 (1.00) |
1 (1.00) |
0.584 (1.00) |
|
| DPP10 | 13 (6%) | 211 |
0.00138 (1.00) |
0.49 (1.00) |
0.0895 (1.00) |
0.317 (1.00) |
0.133 (1.00) |
0.385 (1.00) |
0.158 (1.00) |
|
| EVC2 | 17 (8%) | 207 |
0.00556 (1.00) |
0.00861 (1.00) |
0.0383 (1.00) |
0.0541 (1.00) |
0.0678 (1.00) |
1 (1.00) |
0.711 (1.00) |
|
| C15ORF2 | 12 (5%) | 212 |
0.0187 (1.00) |
0.443 (1.00) |
0.166 (1.00) |
0.296 (1.00) |
0.0859 (1.00) |
0.304 (1.00) |
0.0492 (1.00) |
|
| NLRP5 | 12 (5%) | 212 |
0.172 (1.00) |
1 (1.00) |
0.0649 (1.00) |
0.34 (1.00) |
0.0466 (1.00) |
1 (1.00) |
0.584 (1.00) |
|
| BTNL8 | 3 (1%) | 221 |
0.298 (1.00) |
0.288 (1.00) |
0.254 (1.00) |
0.719 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| KLK2 | 3 (1%) | 221 |
0.471 (1.00) |
0.288 (1.00) |
0.829 (1.00) |
1 (1.00) |
||||
| LUZP2 | 6 (3%) | 218 |
0.0794 (1.00) |
0.183 (1.00) |
0.175 (1.00) |
0.0633 (1.00) |
0.0814 (1.00) |
1 (1.00) |
1 (1.00) |
|
| NXF5 | 7 (3%) | 217 |
0.257 (1.00) |
0.861 (1.00) |
0.425 (1.00) |
0.702 (1.00) |
1 (1.00) |
1 (1.00) |
0.0421 (1.00) |
|
| PCDHB5 | 14 (6%) | 210 |
0.264 (1.00) |
0.496 (1.00) |
0.0582 (1.00) |
0.0752 (1.00) |
0.0227 (1.00) |
0.385 (1.00) |
0.653 (1.00) |
|
| LOXL2 | 9 (4%) | 215 |
0.674 (1.00) |
0.698 (1.00) |
0.191 (1.00) |
0.297 (1.00) |
0.274 (1.00) |
1 (1.00) |
0.32 (1.00) |
|
| UBQLNL | 5 (2%) | 219 |
0.121 (1.00) |
0.208 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| MYO1B | 13 (6%) | 211 |
0.00981 (1.00) |
0.00115 (1.00) |
0.00752 (1.00) |
0.013 (1.00) |
0.469 (1.00) |
1 (1.00) |
0.653 (1.00) |
|
| SPATA17 | 9 (4%) | 215 |
0.111 (1.00) |
0.479 (1.00) |
0.0519 (1.00) |
0.342 (1.00) |
0.188 (1.00) |
1 (1.00) |
1 (1.00) |
|
| RIMS1 | 15 (7%) | 209 |
0.254 (1.00) |
0.408 (1.00) |
0.00135 (1.00) |
0.554 (1.00) |
0.88 (1.00) |
0.458 (1.00) |
0.736 (1.00) |
|
| COL12A1 | 21 (9%) | 203 |
0.0106 (1.00) |
0.398 (1.00) |
0.0451 (1.00) |
0.136 (1.00) |
0.0938 (1.00) |
0.523 (1.00) |
0.0753 (1.00) |
|
| JAKMIP2 | 13 (6%) | 211 |
0.0168 (1.00) |
0.247 (1.00) |
0.0855 (1.00) |
0.533 (1.00) |
0.189 (1.00) |
0.385 (1.00) |
0.158 (1.00) |
|
| EPHA5 | 13 (6%) | 211 |
0.0675 (1.00) |
0.132 (1.00) |
0.0666 (1.00) |
0.634 (1.00) |
0.244 (1.00) |
1 (1.00) |
0.653 (1.00) |
|
| ODZ1 | 22 (10%) | 202 |
0.396 (1.00) |
0.272 (1.00) |
0.0474 (1.00) |
0.783 (1.00) |
0.963 (1.00) |
0.204 (1.00) |
0.669 (1.00) |
|
| POSTN | 11 (5%) | 213 |
0.0215 (1.00) |
0.0882 (1.00) |
0.272 (1.00) |
0.853 (1.00) |
0.177 (1.00) |
1 (1.00) |
1 (1.00) |
|
| ZCWPW2 | 7 (3%) | 217 |
0.36 (1.00) |
0.516 (1.00) |
1 (1.00) |
0.766 (1.00) |
0.375 (1.00) |
1 (1.00) |
0.104 (1.00) |
|
| ZEB2 | 15 (7%) | 209 |
0.0434 (1.00) |
0.87 (1.00) |
0.164 (1.00) |
0.803 (1.00) |
0.88 (1.00) |
0.385 (1.00) |
0.653 (1.00) |
|
| TUSC3 | 7 (3%) | 217 |
0.00678 (1.00) |
0.153 (1.00) |
0.04 (1.00) |
0.38 (1.00) |
0.694 (1.00) |
1 (1.00) |
0.00375 (1.00) |
|
| CACNG7 | 6 (3%) | 218 |
0.649 (1.00) |
0.23 (1.00) |
0.363 (1.00) |
0.0551 (1.00) |
0.0779 (1.00) |
1 (1.00) |
0.213 (1.00) |
|
| RWDD2B | 8 (4%) | 216 |
0.0527 (1.00) |
0.443 (1.00) |
0.182 (1.00) |
0.897 (1.00) |
0.599 (1.00) |
1 (1.00) |
0.00375 (1.00) |
|
| SLC25A13 | 7 (3%) | 217 |
0.148 (1.00) |
0.597 (1.00) |
0.425 (1.00) |
0.162 (1.00) |
0.123 (1.00) |
1 (1.00) |
0.0421 (1.00) |
|
| DPF2 | 4 (2%) | 220 |
0.471 (1.00) |
0.288 (1.00) |
0.381 (1.00) |
0.116 (1.00) |
0.0925 (1.00) |
1 (1.00) |
0.141 (1.00) |
|
| PCDH10 | 17 (8%) | 207 |
0.0996 (1.00) |
0.0505 (1.00) |
0.0139 (1.00) |
0.42 (1.00) |
0.896 (1.00) |
1 (1.00) |
0.23 (1.00) |
|
| CDH12 | 13 (6%) | 211 |
0.00538 (1.00) |
0.132 (1.00) |
0.0874 (1.00) |
0.014 (1.00) |
0.0181 (1.00) |
1 (1.00) |
0.00946 (1.00) |
|
| MCF2 | 12 (5%) | 212 |
0.0885 (1.00) |
0.056 (1.00) |
0.263 (1.00) |
0.235 (1.00) |
0.692 (1.00) |
1 (1.00) |
0.135 (1.00) |
|
| NCKAP5 | 15 (7%) | 209 |
0.0261 (1.00) |
0.018 (1.00) |
0.131 (1.00) |
0.0538 (1.00) |
0.277 (1.00) |
1 (1.00) |
0.711 (1.00) |
|
| RIMS2 | 13 (6%) | 211 |
0.00189 (1.00) |
0.368 (1.00) |
0.000306 (0.433) |
0.0534 (1.00) |
0.469 (1.00) |
1 (1.00) |
0.653 (1.00) |
|
| ATP7A | 6 (3%) | 218 |
0.704 (1.00) |
0.819 (1.00) |
0.917 (1.00) |
0.32 (1.00) |
0.375 (1.00) |
1 (1.00) |
1 (1.00) |
|
| TPTE | 12 (5%) | 212 |
0.0249 (1.00) |
0.231 (1.00) |
0.000369 (0.522) |
0.146 (1.00) |
0.469 (1.00) |
1 (1.00) |
0.62 (1.00) |
|
| OR8B4 | 6 (3%) | 218 |
0.0688 (1.00) |
0.405 (1.00) |
0.206 (1.00) |
0.448 (1.00) |
0.694 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| SFMBT2 | 13 (6%) | 211 |
0.552 (1.00) |
0.247 (1.00) |
0.387 (1.00) |
0.479 (1.00) |
0.647 (1.00) |
1 (1.00) |
0.683 (1.00) |
|
| FLRT2 | 13 (6%) | 211 |
0.149 (1.00) |
0.74 (1.00) |
0.019 (1.00) |
0.165 (1.00) |
0.539 (1.00) |
1 (1.00) |
0.135 (1.00) |
|
| BCHE | 9 (4%) | 215 |
0.057 (1.00) |
0.259 (1.00) |
0.314 (1.00) |
0.766 (1.00) |
0.876 (1.00) |
1 (1.00) |
1 (1.00) |
|
| C8B | 10 (4%) | 214 |
0.0748 (1.00) |
0.259 (1.00) |
0.757 (1.00) |
0.699 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| FREM2 | 23 (10%) | 201 |
0.179 (1.00) |
0.413 (1.00) |
0.299 (1.00) |
0.659 (1.00) |
0.744 (1.00) |
0.6 (1.00) |
0.409 (1.00) |
|
| ZNF167 | 7 (3%) | 217 |
0.287 (1.00) |
0.688 (1.00) |
0.0498 (1.00) |
0.448 (1.00) |
0.274 (1.00) |
1 (1.00) |
0.406 (1.00) |
|
| GABRG1 | 8 (4%) | 216 |
0.562 (1.00) |
0.892 (1.00) |
0.459 (1.00) |
0.583 (1.00) |
0.876 (1.00) |
1 (1.00) |
0.0747 (1.00) |
|
| SAT1 | 4 (2%) | 220 |
0.00282 (1.00) |
0.0612 (1.00) |
0.0742 (1.00) |
0.827 (1.00) |
0.222 (1.00) |
1 (1.00) |
1 (1.00) |
|
| CHD4 | 17 (8%) | 207 |
0.0106 (1.00) |
0.058 (1.00) |
0.000862 (1.00) |
0.168 (1.00) |
0.242 (1.00) |
1 (1.00) |
0.0101 (1.00) |
|
| PCDHB2 | 14 (6%) | 210 |
0.012 (1.00) |
0.00956 (1.00) |
0.0175 (1.00) |
0.185 (1.00) |
0.539 (1.00) |
1 (1.00) |
0.653 (1.00) |
|
| TMTC1 | 13 (6%) | 211 |
0.181 (1.00) |
0.056 (1.00) |
0.0147 (1.00) |
0.0624 (1.00) |
0.102 (1.00) |
0.385 (1.00) |
0.158 (1.00) |
|
| SH3GL3 | 8 (4%) | 216 |
0.108 (1.00) |
1 (1.00) |
0.596 (1.00) |
0.106 (1.00) |
0.876 (1.00) |
1 (1.00) |
0.133 (1.00) |
|
| ZFPM2 | 6 (3%) | 218 |
0.024 (1.00) |
0.736 (1.00) |
0.541 (1.00) |
0.106 (1.00) |
0.0355 (1.00) |
1 (1.00) |
0.0549 (1.00) |
|
| CACNG3 | 8 (4%) | 216 |
0.0187 (1.00) |
0.0952 (1.00) |
0.00923 (1.00) |
0.0646 (1.00) |
0.234 (1.00) |
0.274 (1.00) |
0.0747 (1.00) |
|
| POTEB | 8 (4%) | 216 |
0.309 (1.00) |
1 (1.00) |
0.287 (1.00) |
0.827 (1.00) |
0.482 (1.00) |
1 (1.00) |
1 (1.00) |
|
| GRIN2A | 19 (8%) | 205 |
0.413 (1.00) |
0.462 (1.00) |
0.0439 (1.00) |
0.159 (1.00) |
0.123 (1.00) |
0.481 (1.00) |
0.76 (1.00) |
|
| NRXN3 | 17 (8%) | 207 |
0.000473 (0.667) |
0.0656 (1.00) |
0.00293 (1.00) |
0.737 (1.00) |
0.089 (1.00) |
1 (1.00) |
0.736 (1.00) |
|
| OR7C1 | 6 (3%) | 218 |
0.131 (1.00) |
0.0109 (1.00) |
0.541 (1.00) |
1 (1.00) |
0.406 (1.00) |
|||
| SPATA8 | 3 (1%) | 221 |
0.312 (1.00) |
1 (1.00) |
0.104 (1.00) |
|||||
| OR2M2 | 6 (3%) | 218 |
0.808 (1.00) |
1 (1.00) |
0.917 (1.00) |
0.423 (1.00) |
0.482 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| SORCS1 | 15 (7%) | 209 |
0.243 (1.00) |
0.217 (1.00) |
0.131 (1.00) |
0.967 (1.00) |
0.533 (1.00) |
1 (1.00) |
0.205 (1.00) |
|
| IL18R1 | 10 (4%) | 214 |
0.0398 (1.00) |
0.101 (1.00) |
0.254 (1.00) |
0.625 (1.00) |
1 (1.00) |
1 (1.00) |
0.114 (1.00) |
|
| DPY19L1 | 5 (2%) | 219 |
0.0102 (1.00) |
0.324 (1.00) |
0.194 (1.00) |
0.244 (1.00) |
0.274 (1.00) |
1 (1.00) |
0.292 (1.00) |
|
| C16ORF45 | 3 (1%) | 221 |
0.139 (1.00) |
0.588 (1.00) |
0.108 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| INHBA | 9 (4%) | 215 |
0.005 (1.00) |
0.0952 (1.00) |
0.0706 (1.00) |
0.509 (1.00) |
0.234 (1.00) |
0.025 (1.00) |
0.0575 (1.00) |
|
| MIER3 | 11 (5%) | 213 |
0.000183 (0.26) |
0.0174 (1.00) |
0.00207 (1.00) |
0.67 (1.00) |
0.821 (1.00) |
1 (1.00) |
0.62 (1.00) |
|
| ZNF585A | 10 (4%) | 214 |
0.00278 (1.00) |
0.0485 (1.00) |
0.00451 (1.00) |
0.0897 (1.00) |
0.0172 (1.00) |
1 (1.00) |
0.0935 (1.00) |
|
| TMPRSS13 | 4 (2%) | 220 |
0.388 (1.00) |
0.403 (1.00) |
0.11 (1.00) |
1 (1.00) |
0.292 (1.00) |
|||
| FSTL5 | 11 (5%) | 213 |
0.0586 (1.00) |
0.197 (1.00) |
0.0316 (1.00) |
0.34 (1.00) |
0.282 (1.00) |
1 (1.00) |
0.193 (1.00) |
|
| DOK5 | 6 (3%) | 218 |
0.894 (1.00) |
0.552 (1.00) |
0.628 (1.00) |
0.514 (1.00) |
1 (1.00) |
1 (1.00) |
0.213 (1.00) |
|
| ZNF99 | 11 (5%) | 213 |
0.052 (1.00) |
0.231 (1.00) |
0.0153 (1.00) |
0.07 (1.00) |
0.0355 (1.00) |
1 (1.00) |
0.0387 (1.00) |
|
| GML | 6 (3%) | 218 |
0.453 (1.00) |
0.153 (1.00) |
0.628 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| B2M | 5 (2%) | 219 |
0.0423 (1.00) |
0.11 (1.00) |
0.0452 (1.00) |
0.702 (1.00) |
1 (1.00) |
1 (1.00) |
0.0288 (1.00) |
|
| PPP2R2B | 3 (1%) | 221 |
0.471 (1.00) |
0.288 (1.00) |
0.445 (1.00) |
1 (1.00) |
0.228 (1.00) |
|||
| LRTM1 | 7 (3%) | 217 |
0.0934 (1.00) |
0.218 (1.00) |
0.402 (1.00) |
0.0191 (1.00) |
0.0502 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| OR10G9 | 6 (3%) | 218 |
0.241 (1.00) |
0.324 (1.00) |
0.206 (1.00) |
0.612 (1.00) |
0.436 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| ERBB3 | 14 (6%) | 210 |
0.147 (1.00) |
0.09 (1.00) |
0.124 (1.00) |
0.629 (1.00) |
0.524 (1.00) |
1 (1.00) |
0.158 (1.00) |
|
| PSG8 | 8 (4%) | 216 |
0.894 (1.00) |
0.477 (1.00) |
0.107 (1.00) |
0.439 (1.00) |
0.599 (1.00) |
0.244 (1.00) |
0.249 (1.00) |
|
| SYT16 | 9 (4%) | 215 |
0.0778 (1.00) |
0.042 (1.00) |
0.182 (1.00) |
0.158 (1.00) |
0.234 (1.00) |
0.304 (1.00) |
0.545 (1.00) |
|
| LPHN3 | 13 (6%) | 211 |
0.00327 (1.00) |
0.0792 (1.00) |
0.387 (1.00) |
0.00382 (1.00) |
0.201 (1.00) |
1 (1.00) |
0.135 (1.00) |
|
| LRP2 | 35 (16%) | 189 |
0.00219 (1.00) |
0.0155 (1.00) |
0.0014 (1.00) |
0.468 (1.00) |
0.639 (1.00) |
0.342 (1.00) |
0.277 (1.00) |
|
| SOHLH2 | 9 (4%) | 215 |
0.86 (1.00) |
0.042 (1.00) |
0.327 (1.00) |
0.0422 (1.00) |
0.274 (1.00) |
1 (1.00) |
0.502 (1.00) |
|
| ZIM3 | 9 (4%) | 215 |
0.0665 (1.00) |
0.892 (1.00) |
0.119 (1.00) |
0.0131 (1.00) |
0.759 (1.00) |
1 (1.00) |
0.0285 (1.00) |
|
| DSEL | 14 (6%) | 210 |
0.0139 (1.00) |
0.217 (1.00) |
0.00117 (1.00) |
0.0766 (1.00) |
0.244 (1.00) |
1 (1.00) |
0.158 (1.00) |
|
| RNASE11 | 5 (2%) | 219 |
0.814 (1.00) |
0.71 (1.00) |
0.148 (1.00) |
0.337 (1.00) |
0.668 (1.00) |
1 (1.00) |
0.0177 (1.00) |
|
| MDGA2 | 10 (4%) | 214 |
0.0217 (1.00) |
0.03 (1.00) |
0.0201 (1.00) |
0.106 (1.00) |
0.109 (1.00) |
0.304 (1.00) |
1 (1.00) |
|
| RBMS3 | 6 (3%) | 218 |
0.294 (1.00) |
0.841 (1.00) |
1 (1.00) |
0.296 (1.00) |
1 (1.00) |
1 (1.00) |
0.406 (1.00) |
|
| COL6A6 | 16 (7%) | 208 |
0.0612 (1.00) |
0.496 (1.00) |
0.00426 (1.00) |
0.555 (1.00) |
0.826 (1.00) |
0.458 (1.00) |
0.736 (1.00) |
|
| TMBIM4 | 4 (2%) | 220 |
1 (1.00) |
1 (1.00) |
0.65 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| FAM135B | 15 (7%) | 209 |
0.0233 (1.00) |
0.0471 (1.00) |
0.116 (1.00) |
0.928 (1.00) |
0.711 (1.00) |
0.107 (1.00) |
0.354 (1.00) |
|
| CYP7B1 | 7 (3%) | 217 |
0.0102 (1.00) |
0.0593 (1.00) |
0.108 (1.00) |
0.162 (1.00) |
0.792 (1.00) |
1 (1.00) |
0.351 (1.00) |
|
| SLC16A7 | 9 (4%) | 215 |
0.0291 (1.00) |
0.101 (1.00) |
0.119 (1.00) |
0.716 (1.00) |
0.274 (1.00) |
1 (1.00) |
0.0747 (1.00) |
P value = 0.000122 (Fisher's exact test), Q value = 0.17
Table S1. Gene #1: 'FBXW7 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| FBXW7 MUTATED | 4 | 24 | 8 | 2 |
| FBXW7 WILD-TYPE | 82 | 59 | 28 | 14 |
Figure S1. Get High-res Image Gene #1: 'FBXW7 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
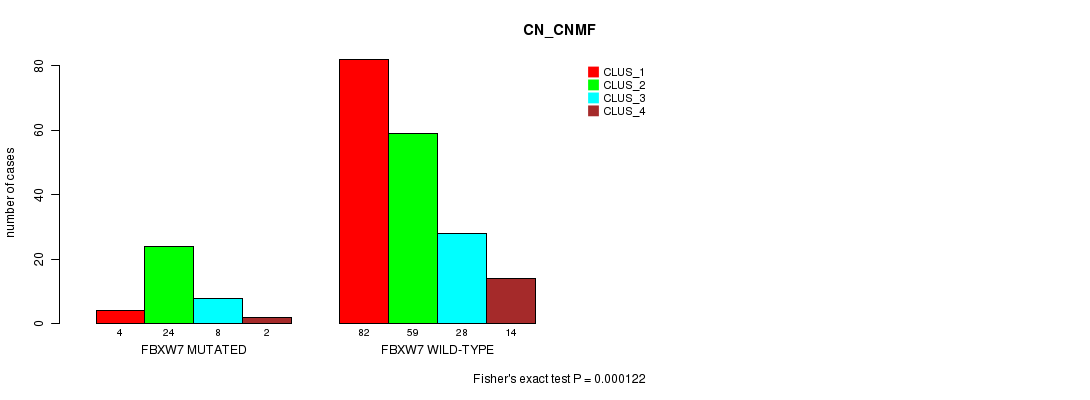
P value = 1.37e-06 (Fisher's exact test), Q value = 0.002
Table S2. Gene #5: 'BRAF MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| BRAF MUTATED | 13 | 1 | 1 | 5 |
| BRAF WILD-TYPE | 31 | 56 | 66 | 32 |
Figure S2. Get High-res Image Gene #5: 'BRAF MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
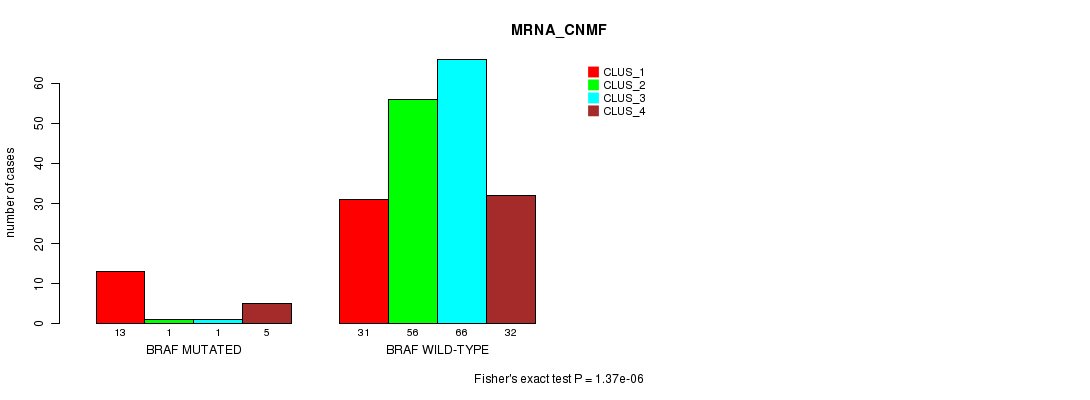
P value = 1.88e-07 (Fisher's exact test), Q value = 0.00027
Table S3. Gene #5: 'BRAF MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 |
|---|---|---|---|
| ALL | 59 | 108 | 38 |
| BRAF MUTATED | 17 | 2 | 1 |
| BRAF WILD-TYPE | 42 | 106 | 37 |
Figure S3. Get High-res Image Gene #5: 'BRAF MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
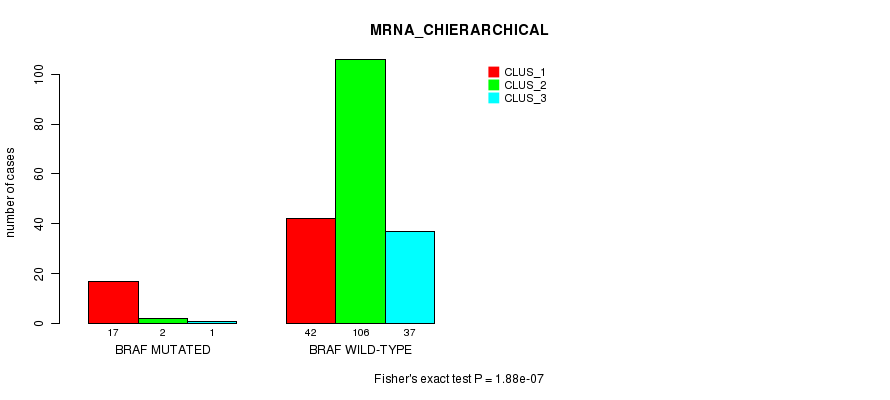
P value = 4.17e-05 (Fisher's exact test), Q value = 0.06
Table S4. Gene #5: 'BRAF MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| BRAF MUTATED | 1 | 18 | 3 | 0 |
| BRAF WILD-TYPE | 85 | 65 | 33 | 16 |
Figure S4. Get High-res Image Gene #5: 'BRAF MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
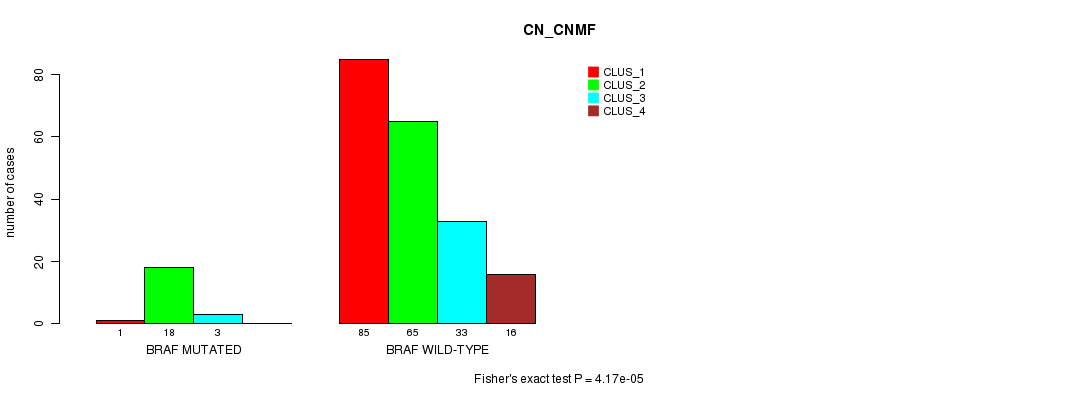
P value = 7.28e-11 (Fisher's exact test), Q value = 1e-07
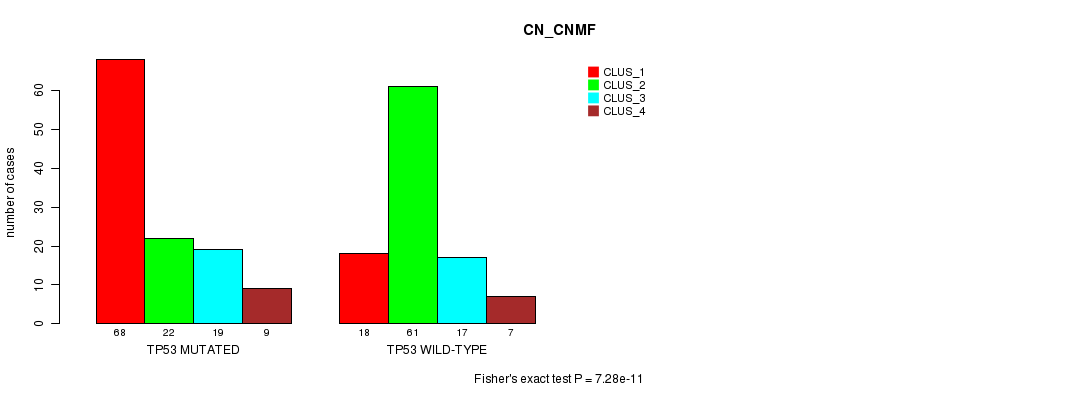
Table S5. Gene #8: 'TP53 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| TP53 MUTATED | 68 | 22 | 19 | 9 |
| TP53 WILD-TYPE | 18 | 61 | 17 | 7 |
Figure S5. Get High-res Image Gene #8: 'TP53 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
P value = 6.63e-05 (Fisher's exact test), Q value = 0.095
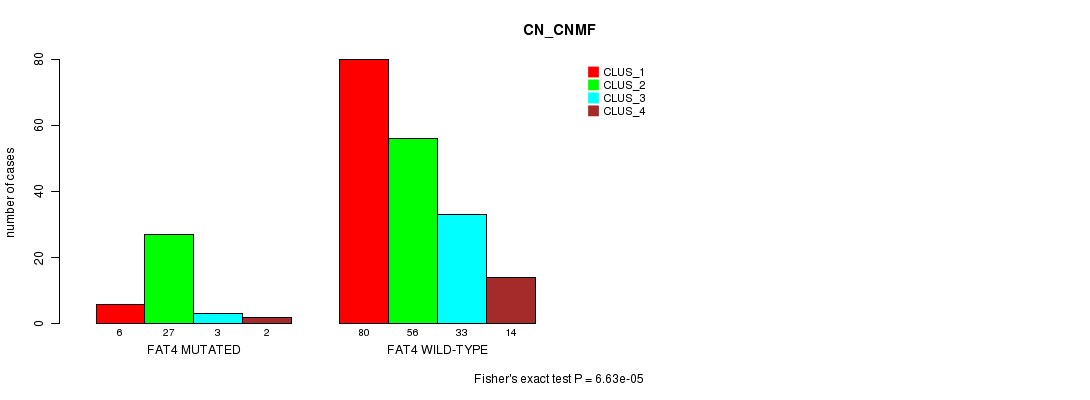
Table S6. Gene #13: 'FAT4 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| FAT4 MUTATED | 6 | 27 | 3 | 2 |
| FAT4 WILD-TYPE | 80 | 56 | 33 | 14 |
Figure S6. Get High-res Image Gene #13: 'FAT4 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
P value = 3.86e-05 (Fisher's exact test), Q value = 0.055
Table S7. Gene #35: 'CDH2 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| CDH2 MUTATED | 7 | 0 | 1 | 7 |
| CDH2 WILD-TYPE | 37 | 57 | 66 | 30 |
Figure S7. Get High-res Image Gene #35: 'CDH2 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'

P value = 3.08e-06 (Fisher's exact test), Q value = 0.0044
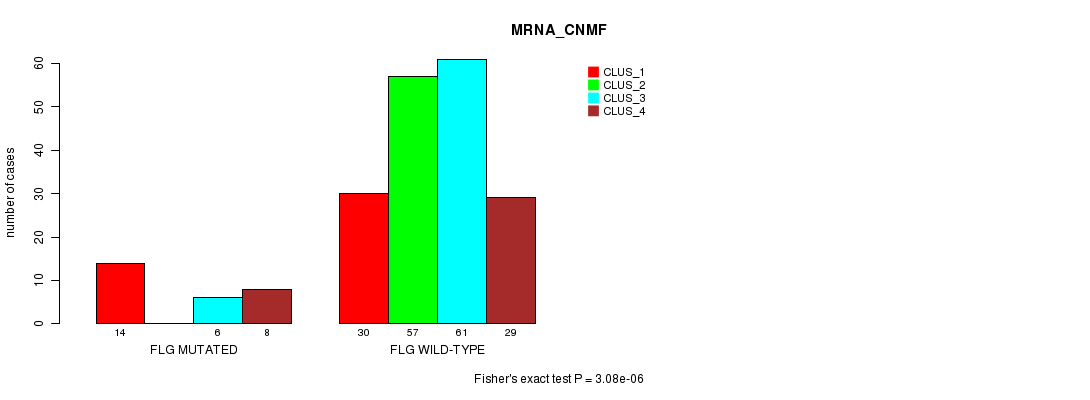
Table S8. Gene #40: 'FLG MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| FLG MUTATED | 14 | 0 | 6 | 8 |
| FLG WILD-TYPE | 30 | 57 | 61 | 29 |
Figure S8. Get High-res Image Gene #40: 'FLG MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
P value = 0.00013 (Fisher's exact test), Q value = 0.18
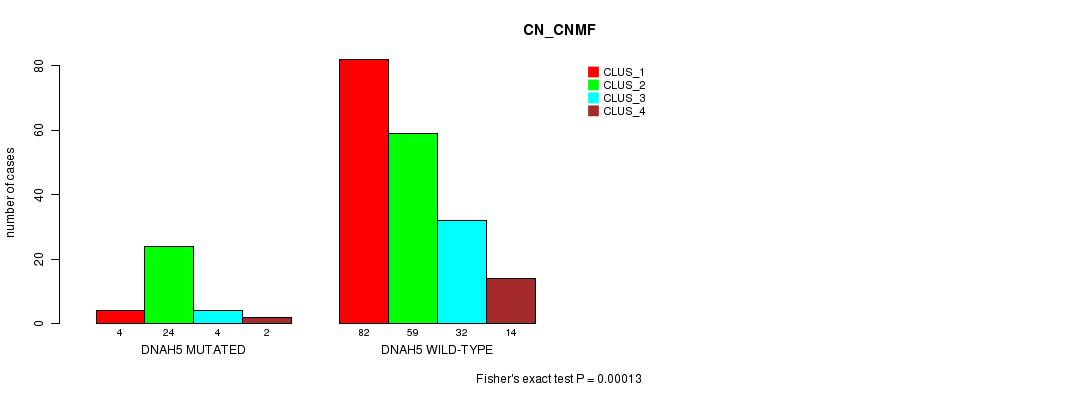
Table S9. Gene #51: 'DNAH5 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| DNAH5 MUTATED | 4 | 24 | 4 | 2 |
| DNAH5 WILD-TYPE | 82 | 59 | 32 | 14 |
Figure S9. Get High-res Image Gene #51: 'DNAH5 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
P value = 3.34e-05 (Fisher's exact test), Q value = 0.048
Table S10. Gene #58: 'TMEM132D MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| TMEM132D MUTATED | 11 | 1 | 1 | 3 |
| TMEM132D WILD-TYPE | 33 | 56 | 66 | 34 |
Figure S10. Get High-res Image Gene #58: 'TMEM132D MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
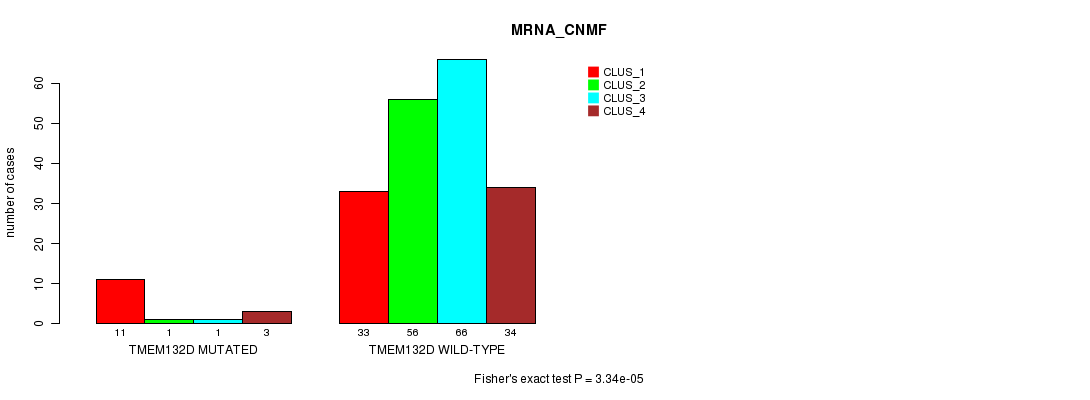
P value = 7.61e-05 (Fisher's exact test), Q value = 0.11
Table S11. Gene #100: 'ANK2 MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 |
|---|---|---|---|
| ALL | 59 | 108 | 38 |
| ANK2 MUTATED | 16 | 9 | 0 |
| ANK2 WILD-TYPE | 43 | 99 | 38 |
Figure S11. Get High-res Image Gene #100: 'ANK2 MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
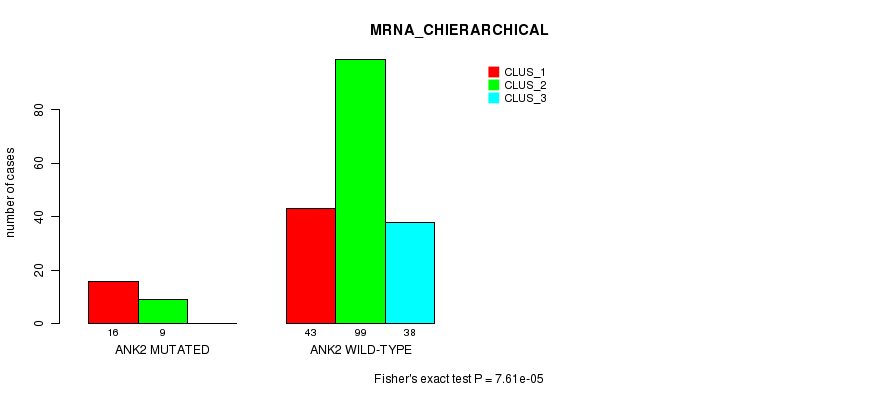
P value = 7.69e-05 (Fisher's exact test), Q value = 0.11
Table S12. Gene #109: 'ZC3H13 MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 |
|---|---|---|---|
| ALL | 59 | 108 | 38 |
| ZC3H13 MUTATED | 14 | 4 | 1 |
| ZC3H13 WILD-TYPE | 45 | 104 | 37 |
Figure S12. Get High-res Image Gene #109: 'ZC3H13 MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
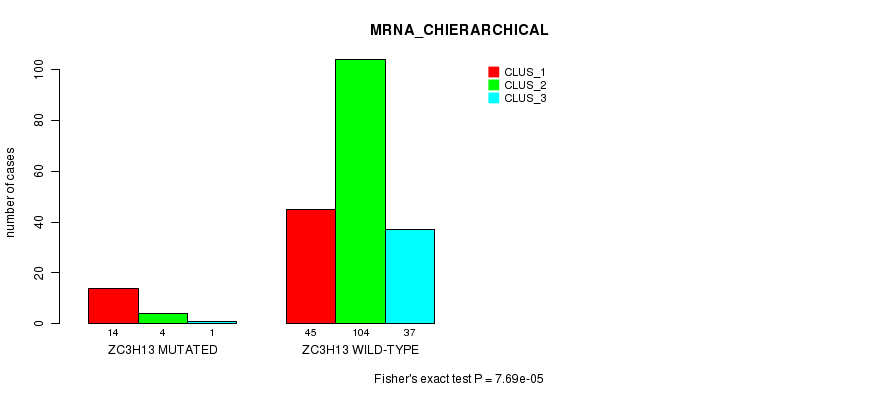
P value = 8.41e-05 (Fisher's exact test), Q value = 0.12
Table S13. Gene #109: 'ZC3H13 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| ZC3H13 MUTATED | 2 | 18 | 1 | 0 |
| ZC3H13 WILD-TYPE | 84 | 65 | 35 | 16 |
Figure S13. Get High-res Image Gene #109: 'ZC3H13 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
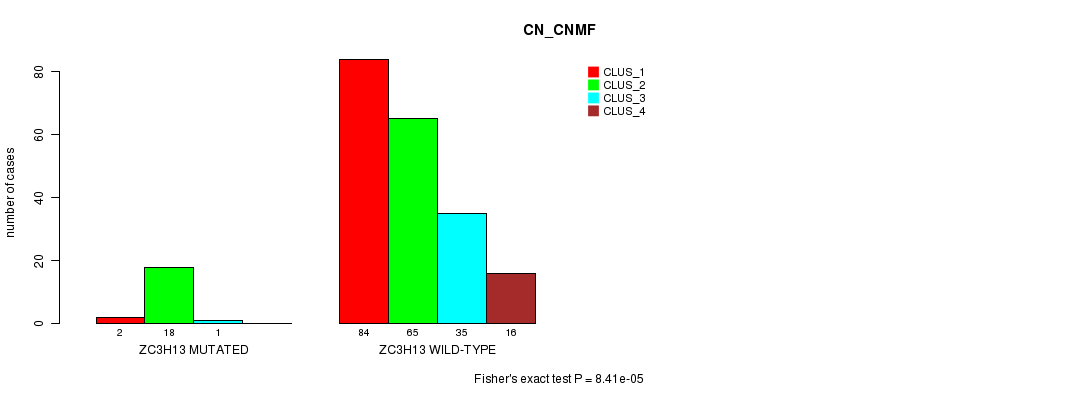
P value = 0.000159 (Fisher's exact test), Q value = 0.23
Table S14. Gene #119: 'DOCK2 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| DOCK2 MUTATED | 8 | 1 | 1 | 7 |
| DOCK2 WILD-TYPE | 36 | 56 | 66 | 30 |
Figure S14. Get High-res Image Gene #119: 'DOCK2 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
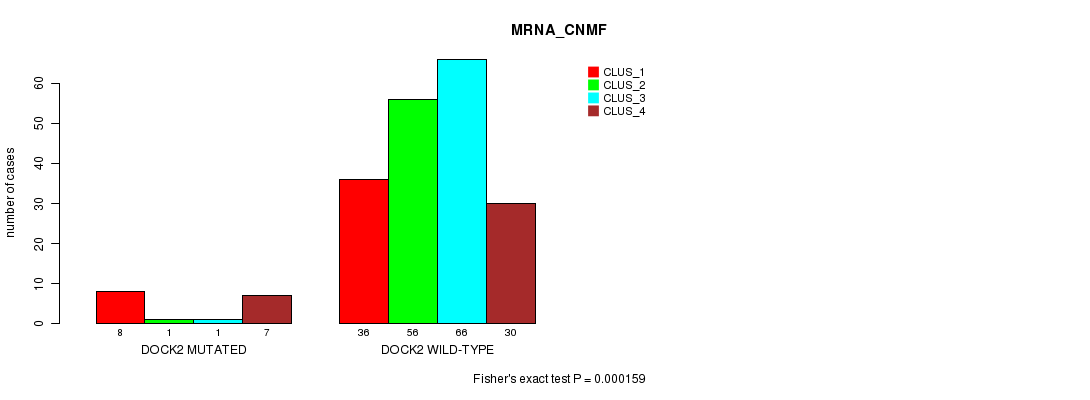
P value = 0.000138 (Fisher's exact test), Q value = 0.2
Table S15. Gene #119: 'DOCK2 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| DOCK2 MUTATED | 1 | 17 | 2 | 1 |
| DOCK2 WILD-TYPE | 85 | 66 | 34 | 15 |
Figure S15. Get High-res Image Gene #119: 'DOCK2 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'

P value = 6.12e-05 (Fisher's exact test), Q value = 0.087
Table S16. Gene #138: 'CDH10 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| CDH10 MUTATED | 4 | 0 | 1 | 8 |
| CDH10 WILD-TYPE | 40 | 57 | 66 | 29 |
Figure S16. Get High-res Image Gene #138: 'CDH10 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
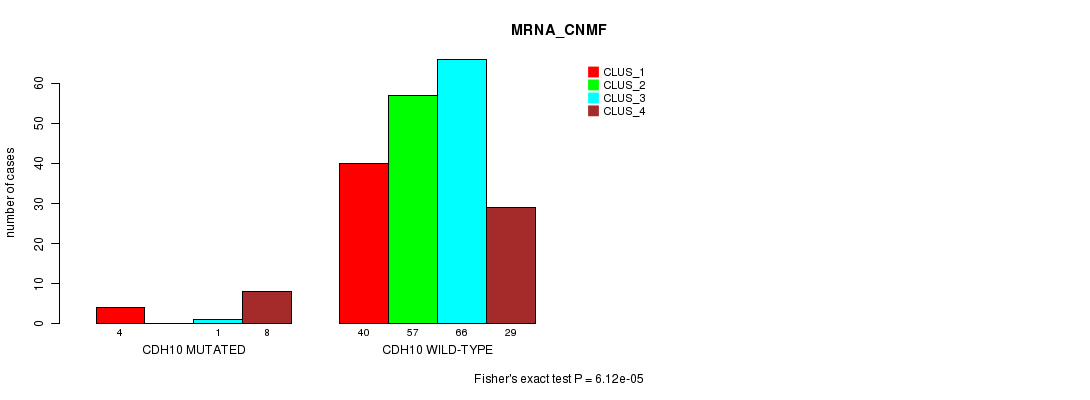
P value = 0.000131 (Fisher's exact test), Q value = 0.19
Table S17. Gene #138: 'CDH10 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| CDH10 MUTATED | 0 | 13 | 1 | 0 |
| CDH10 WILD-TYPE | 86 | 70 | 35 | 16 |
Figure S17. Get High-res Image Gene #138: 'CDH10 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
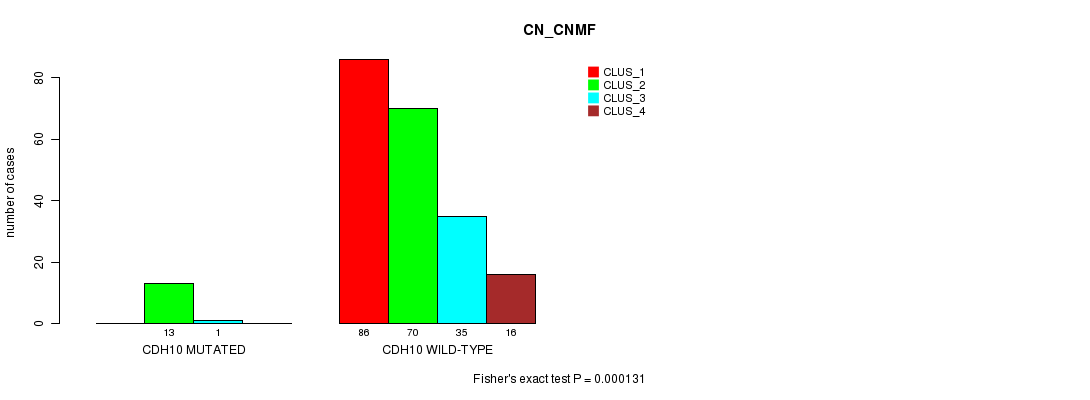
P value = 3.76e-05 (Fisher's exact test), Q value = 0.054
Table S18. Gene #142: 'SYNE1 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 44 | 57 | 67 | 37 |
| SYNE1 MUTATED | 14 | 8 | 6 | 17 |
| SYNE1 WILD-TYPE | 30 | 49 | 61 | 20 |
Figure S18. Get High-res Image Gene #142: 'SYNE1 MUTATION STATUS' versus Clinical Feature #1: 'MRNA_CNMF'
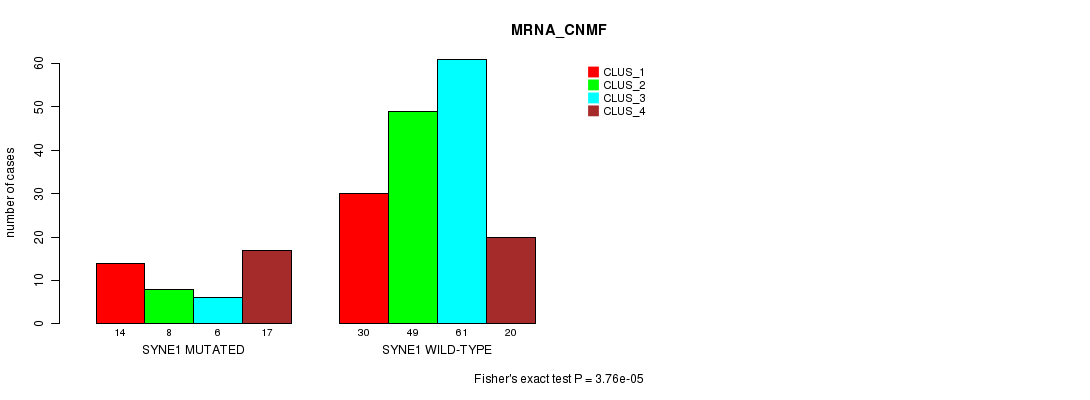
P value = 9.65e-06 (Fisher's exact test), Q value = 0.014
Table S19. Gene #142: 'SYNE1 MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 |
|---|---|---|---|
| ALL | 59 | 108 | 38 |
| SYNE1 MUTATED | 26 | 16 | 3 |
| SYNE1 WILD-TYPE | 33 | 92 | 35 |
Figure S19. Get High-res Image Gene #142: 'SYNE1 MUTATION STATUS' versus Clinical Feature #2: 'MRNA_CHIERARCHICAL'
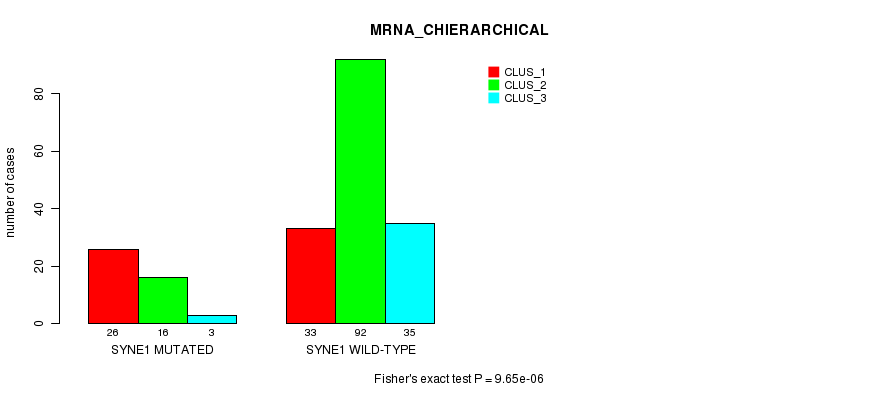
P value = 1.63e-06 (Fisher's exact test), Q value = 0.0023
Table S20. Gene #142: 'SYNE1 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 |
|---|---|---|---|---|
| ALL | 86 | 83 | 36 | 16 |
| SYNE1 MUTATED | 7 | 32 | 6 | 0 |
| SYNE1 WILD-TYPE | 79 | 51 | 30 | 16 |
Figure S20. Get High-res Image Gene #142: 'SYNE1 MUTATION STATUS' versus Clinical Feature #3: 'CN_CNMF'
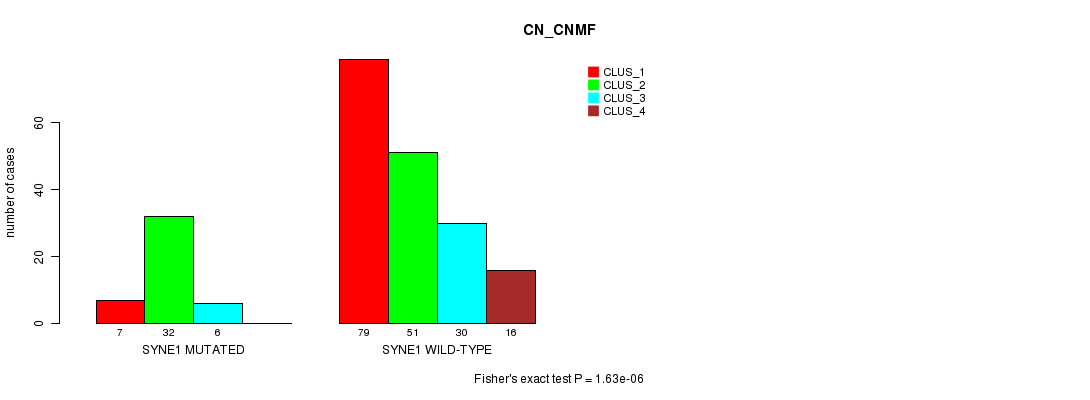
-
Mutation data file = COADREAD-TP.mutsig.cluster.txt
-
Molecular subtypes file = COADREAD-TP.transferedmergedcluster.txt
-
Number of patients = 224
-
Number of significantly mutated genes = 209
-
Number of Molecular subtypes = 8
-
Exclude genes that fewer than K tumors have mutations, K = 3
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
This is an experimental feature. The full results of the analysis summarized in this report can be downloaded from the TCGA Data Coordination Center.