This pipeline computes the correlation between significantly recurrent gene mutations and selected clinical features.
Testing the association between mutation status of 179 genes and 13 clinical features across 172 patients, 6 significant findings detected with Q value < 0.25.
-
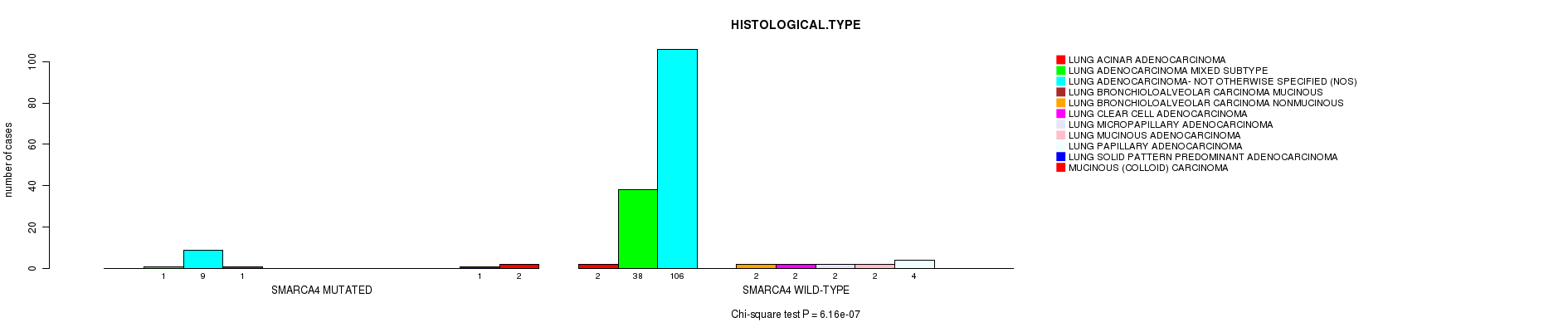
SMARCA4 mutation correlated to 'HISTOLOGICAL.TYPE'.
-
U2AF1 mutation correlated to 'HISTOLOGICAL.TYPE'.
-
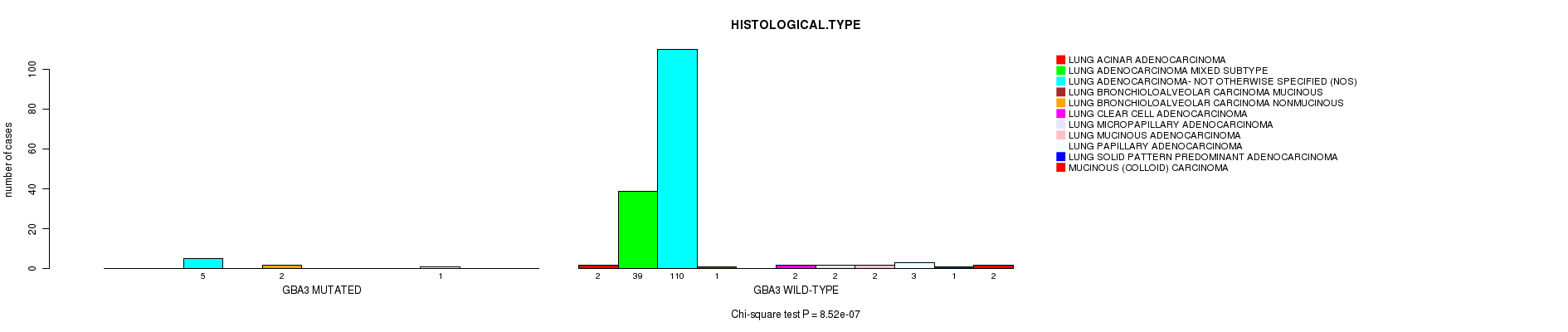
GBA3 mutation correlated to 'HISTOLOGICAL.TYPE'.
-
OR13G1 mutation correlated to 'HISTOLOGICAL.TYPE'.
-
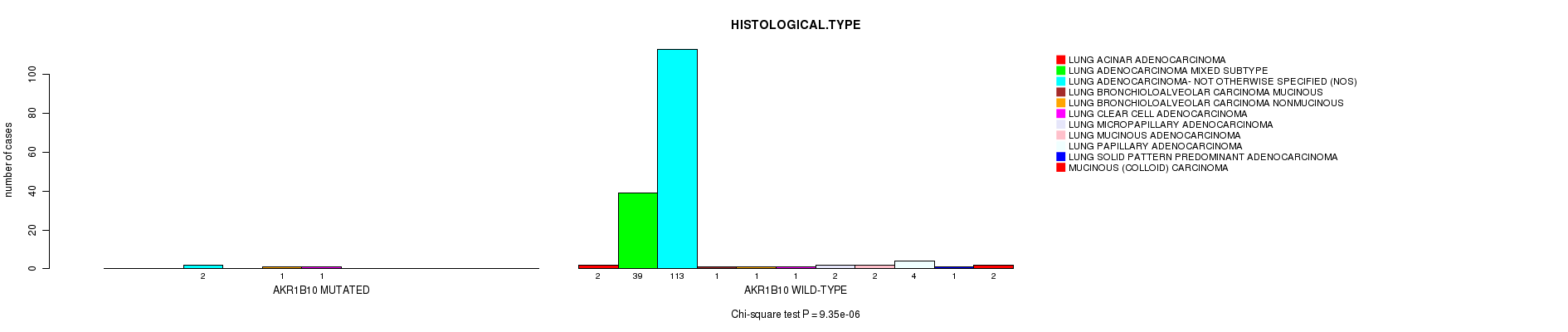
AKR1B10 mutation correlated to 'HISTOLOGICAL.TYPE'.
-
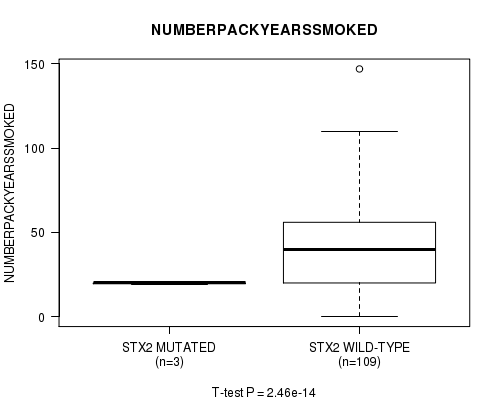
STX2 mutation correlated to 'NUMBERPACKYEARSSMOKED'.
Table 1. Get Full Table Overview of the association between mutation status of 179 genes and 13 clinical features. Shown in the table are P values (Q values). Thresholded by Q value < 0.25, 6 significant findings detected.
|
Clinical Features |
Time to Death |
AGE |
NEOPLASM DISEASESTAGE |
PATHOLOGY T STAGE |
PATHOLOGY N STAGE |
PATHOLOGY M STAGE |
GENDER |
KARNOFSKY PERFORMANCE SCORE |
HISTOLOGICAL TYPE |
RADIATIONS RADIATION REGIMENINDICATION |
NUMBERPACKYEARSSMOKED | YEAROFTOBACCOSMOKINGONSET |
COMPLETENESS OF RESECTION |
||
| nMutated (%) | nWild-Type | logrank test | t-test | Chi-square test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | t-test | Chi-square test | Fisher's exact test | t-test | t-test | Fisher's exact test | |
| SMARCA4 | 14 (8%) | 158 |
0.609 (1.00) |
0.769 (1.00) |
0.42 (1.00) |
0.868 (1.00) |
0.694 (1.00) |
0.707 (1.00) |
0.171 (1.00) |
6.16e-07 (0.0013) |
1 (1.00) |
0.657 (1.00) |
0.188 (1.00) |
1 (1.00) |
|
| U2AF1 | 6 (3%) | 166 |
0.4 (1.00) |
0.89 (1.00) |
0.484 (1.00) |
0.855 (1.00) |
0.0572 (1.00) |
0.0713 (1.00) |
0.22 (1.00) |
1.19e-08 (2.51e-05) |
0.427 (1.00) |
0.762 (1.00) |
0.265 (1.00) |
1 (1.00) |
|
| GBA3 | 8 (5%) | 164 |
0.405 (1.00) |
0.0279 (1.00) |
0.258 (1.00) |
1 (1.00) |
0.0409 (1.00) |
1 (1.00) |
0.728 (1.00) |
8.52e-07 (0.0018) |
1 (1.00) |
0.0895 (1.00) |
0.379 (1.00) |
1 (1.00) |
|
| OR13G1 | 9 (5%) | 163 |
0.12 (1.00) |
0.215 (1.00) |
0.446 (1.00) |
1 (1.00) |
0.14 (1.00) |
1 (1.00) |
1 (1.00) |
7.17e-06 (0.0152) |
1 (1.00) |
0.273 (1.00) |
0.879 (1.00) |
1 (1.00) |
|
| AKR1B10 | 4 (2%) | 168 |
0.446 (1.00) |
0.00728 (1.00) |
0.581 (1.00) |
1 (1.00) |
0.783 (1.00) |
0.00711 (1.00) |
1 (1.00) |
9.35e-06 (0.0198) |
0.308 (1.00) |
0.353 (1.00) |
|||
| STX2 | 4 (2%) | 168 |
0.891 (1.00) |
0.601 (1.00) |
0.36 (1.00) |
0.11 (1.00) |
1 (1.00) |
1 (1.00) |
0.334 (1.00) |
0.997 (1.00) |
0.308 (1.00) |
2.46e-14 (5.22e-11) |
0.759 (1.00) |
1 (1.00) |
|
| CDKN2A | 8 (5%) | 164 |
0.422 (1.00) |
0.118 (1.00) |
0.953 (1.00) |
1 (1.00) |
0.764 (1.00) |
1 (1.00) |
0.728 (1.00) |
1 (1.00) |
1 (1.00) |
0.471 (1.00) |
0.0411 (1.00) |
1 (1.00) |
|
| KRAS | 47 (27%) | 125 |
0.796 (1.00) |
0.953 (1.00) |
0.59 (1.00) |
0.0847 (1.00) |
0.405 (1.00) |
1 (1.00) |
0.493 (1.00) |
0.428 (1.00) |
0.913 (1.00) |
0.362 (1.00) |
0.575 (1.00) |
0.779 (1.00) |
0.3 (1.00) |
| TP53 | 87 (51%) | 85 |
0.7 (1.00) |
0.0614 (1.00) |
0.892 (1.00) |
0.645 (1.00) |
0.84 (1.00) |
0.542 (1.00) |
0.225 (1.00) |
0.74 (1.00) |
0.233 (1.00) |
1 (1.00) |
0.363 (1.00) |
0.396 (1.00) |
0.914 (1.00) |
| EGFR | 22 (13%) | 150 |
0.0192 (1.00) |
0.142 (1.00) |
0.613 (1.00) |
0.701 (1.00) |
0.468 (1.00) |
0.132 (1.00) |
0.176 (1.00) |
0.723 (1.00) |
0.222 (1.00) |
0.485 (1.00) |
0.168 (1.00) |
0.565 (1.00) |
|
| STK11 | 20 (12%) | 152 |
0.145 (1.00) |
0.589 (1.00) |
0.415 (1.00) |
0.601 (1.00) |
0.851 (1.00) |
1 (1.00) |
0.0941 (1.00) |
0.983 (1.00) |
0.688 (1.00) |
0.447 (1.00) |
0.403 (1.00) |
0.621 (1.00) |
|
| KEAP1 | 32 (19%) | 140 |
0.632 (1.00) |
0.792 (1.00) |
0.721 (1.00) |
0.513 (1.00) |
1 (1.00) |
0.177 (1.00) |
1 (1.00) |
0.128 (1.00) |
0.239 (1.00) |
1 (1.00) |
0.179 (1.00) |
0.216 (1.00) |
0.945 (1.00) |
| RBM10 | 12 (7%) | 160 |
0.696 (1.00) |
0.179 (1.00) |
0.701 (1.00) |
0.00478 (1.00) |
0.12 (1.00) |
0.535 (1.00) |
0.0676 (1.00) |
0.996 (1.00) |
0.603 (1.00) |
0.378 (1.00) |
0.139 (1.00) |
0.702 (1.00) |
|
| NF1 | 21 (12%) | 151 |
0.517 (1.00) |
0.627 (1.00) |
0.434 (1.00) |
0.474 (1.00) |
0.399 (1.00) |
0.0206 (1.00) |
0.819 (1.00) |
0.786 (1.00) |
0.4 (1.00) |
0.65 (1.00) |
0.902 (1.00) |
0.529 (1.00) |
|
| GPR112 | 35 (20%) | 137 |
0.522 (1.00) |
0.955 (1.00) |
0.315 (1.00) |
0.0486 (1.00) |
0.115 (1.00) |
1 (1.00) |
0.569 (1.00) |
0.645 (1.00) |
0.353 (1.00) |
0.51 (1.00) |
0.256 (1.00) |
0.982 (1.00) |
0.522 (1.00) |
| FLG | 42 (24%) | 130 |
0.0431 (1.00) |
0.282 (1.00) |
0.386 (1.00) |
0.528 (1.00) |
0.49 (1.00) |
0.758 (1.00) |
0.723 (1.00) |
0.807 (1.00) |
0.204 (1.00) |
0.731 (1.00) |
0.1 (1.00) |
0.345 (1.00) |
|
| HRNR | 24 (14%) | 148 |
0.392 (1.00) |
0.374 (1.00) |
0.0106 (1.00) |
0.0606 (1.00) |
0.373 (1.00) |
0.216 (1.00) |
0.389 (1.00) |
0.0632 (1.00) |
0.652 (1.00) |
0.444 (1.00) |
0.648 (1.00) |
0.515 (1.00) |
0.736 (1.00) |
| MUC7 | 14 (8%) | 158 |
0.258 (1.00) |
0.308 (1.00) |
0.749 (1.00) |
0.619 (1.00) |
0.805 (1.00) |
1 (1.00) |
0.0539 (1.00) |
0.681 (1.00) |
1 (1.00) |
0.507 (1.00) |
0.345 (1.00) |
0.639 (1.00) |
|
| BRAF | 15 (9%) | 157 |
0.391 (1.00) |
0.495 (1.00) |
0.231 (1.00) |
0.168 (1.00) |
0.659 (1.00) |
0.353 (1.00) |
0.287 (1.00) |
0.997 (1.00) |
0.129 (1.00) |
0.81 (1.00) |
0.491 (1.00) |
0.796 (1.00) |
|
| COL11A1 | 34 (20%) | 138 |
0.767 (1.00) |
0.829 (1.00) |
0.671 (1.00) |
0.88 (1.00) |
0.582 (1.00) |
0.513 (1.00) |
1 (1.00) |
0.0327 (1.00) |
0.217 (1.00) |
0.5 (1.00) |
0.623 (1.00) |
0.383 (1.00) |
0.784 (1.00) |
| PCK1 | 10 (6%) | 162 |
0.3 (1.00) |
0.321 (1.00) |
0.112 (1.00) |
0.146 (1.00) |
0.613 (1.00) |
0.0886 (1.00) |
1 (1.00) |
0.999 (1.00) |
1 (1.00) |
0.517 (1.00) |
0.00122 (1.00) |
0.126 (1.00) |
|
| RIMS2 | 32 (19%) | 140 |
0.865 (1.00) |
0.708 (1.00) |
0.431 (1.00) |
0.555 (1.00) |
0.871 (1.00) |
0.92 (1.00) |
0.239 (1.00) |
0.953 (1.00) |
0.719 (1.00) |
1 (1.00) |
0.319 (1.00) |
0.0367 (1.00) |
0.457 (1.00) |
| CSMD3 | 78 (45%) | 94 |
0.0637 (1.00) |
0.41 (1.00) |
0.0805 (1.00) |
0.596 (1.00) |
0.0709 (1.00) |
0.543 (1.00) |
0.645 (1.00) |
0.756 (1.00) |
0.767 (1.00) |
0.789 (1.00) |
0.33 (1.00) |
0.165 (1.00) |
0.842 (1.00) |
| RB1 | 7 (4%) | 165 |
0.204 (1.00) |
0.292 (1.00) |
0.735 (1.00) |
0.478 (1.00) |
0.764 (1.00) |
0.203 (1.00) |
0.25 (1.00) |
0.306 (1.00) |
1 (1.00) |
0.888 (1.00) |
0.961 (1.00) |
0.666 (1.00) |
|
| FTSJD1 | 6 (3%) | 166 |
0.629 (1.00) |
0.839 (1.00) |
0.204 (1.00) |
0.416 (1.00) |
0.622 (1.00) |
1 (1.00) |
0.688 (1.00) |
1 (1.00) |
1 (1.00) |
0.587 (1.00) |
0.234 (1.00) |
1 (1.00) |
|
| RIT1 | 8 (5%) | 164 |
0.664 (1.00) |
0.301 (1.00) |
0.474 (1.00) |
0.38 (1.00) |
0.161 (1.00) |
0.142 (1.00) |
1 (1.00) |
0.924 (1.00) |
0.526 (1.00) |
0.351 (1.00) |
0.287 (1.00) |
0.15 (1.00) |
|
| LRP1B | 60 (35%) | 112 |
0.357 (1.00) |
0.698 (1.00) |
0.376 (1.00) |
0.296 (1.00) |
0.361 (1.00) |
0.49 (1.00) |
0.336 (1.00) |
0.726 (1.00) |
0.833 (1.00) |
1 (1.00) |
0.333 (1.00) |
0.934 (1.00) |
0.255 (1.00) |
| OR10J3 | 9 (5%) | 163 |
0.919 (1.00) |
0.941 (1.00) |
0.332 (1.00) |
0.471 (1.00) |
0.396 (1.00) |
0.757 (1.00) |
0.734 (1.00) |
0.977 (1.00) |
0.569 (1.00) |
0.8 (1.00) |
0.908 (1.00) |
1 (1.00) |
|
| LTBP1 | 20 (12%) | 152 |
0.147 (1.00) |
0.415 (1.00) |
0.234 (1.00) |
0.751 (1.00) |
0.0307 (1.00) |
0.0777 (1.00) |
0.638 (1.00) |
0.887 (1.00) |
1 (1.00) |
0.0046 (1.00) |
0.297 (1.00) |
0.786 (1.00) |
|
| SETD2 | 15 (9%) | 157 |
0.215 (1.00) |
0.514 (1.00) |
0.547 (1.00) |
0.341 (1.00) |
0.762 (1.00) |
0.232 (1.00) |
0.175 (1.00) |
0.986 (1.00) |
0.129 (1.00) |
0.282 (1.00) |
0.417 (1.00) |
0.307 (1.00) |
|
| ZCCHC5 | 10 (6%) | 162 |
0.296 (1.00) |
0.62 (1.00) |
0.25 (1.00) |
0.109 (1.00) |
0.827 (1.00) |
0.774 (1.00) |
0.346 (1.00) |
0.887 (1.00) |
0.603 (1.00) |
0.598 (1.00) |
0.0232 (1.00) |
1 (1.00) |
|
| ADAMTS5 | 14 (8%) | 158 |
0.00361 (1.00) |
0.396 (1.00) |
0.0251 (1.00) |
0.272 (1.00) |
0.478 (1.00) |
0.39 (1.00) |
1 (1.00) |
0.987 (1.00) |
0.35 (1.00) |
0.899 (1.00) |
0.463 (1.00) |
1 (1.00) |
|
| MYL10 | 5 (3%) | 167 |
0.512 (1.00) |
0.421 (1.00) |
0.177 (1.00) |
0.0222 (1.00) |
0.172 (1.00) |
1 (1.00) |
1 (1.00) |
0.935 (1.00) |
0.37 (1.00) |
0.221 (1.00) |
0.23 (1.00) |
||
| SMAD4 | 7 (4%) | 165 |
0.763 (1.00) |
0.462 (1.00) |
0.529 (1.00) |
0.0838 (1.00) |
0.764 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.541 (1.00) |
0.0108 (1.00) |
1 (1.00) |
|
| ZNF268 | 5 (3%) | 167 |
0.871 (1.00) |
0.503 (1.00) |
0.566 (1.00) |
1 (1.00) |
1 (1.00) |
0.314 (1.00) |
0.181 (1.00) |
1 (1.00) |
1 (1.00) |
0.213 (1.00) |
0.437 (1.00) |
0.462 (1.00) |
|
| OR4Q3 | 11 (6%) | 161 |
0.232 (1.00) |
0.631 (1.00) |
0.271 (1.00) |
0.0388 (1.00) |
0.262 (1.00) |
0.38 (1.00) |
0.115 (1.00) |
0.697 (1.00) |
0.247 (1.00) |
0.174 (1.00) |
0.527 (1.00) |
||
| ARID1A | 11 (6%) | 161 |
0.376 (1.00) |
0.0116 (1.00) |
0.923 (1.00) |
0.875 (1.00) |
0.827 (1.00) |
0.652 (1.00) |
0.551 (1.00) |
0.994 (1.00) |
0.602 (1.00) |
0.175 (1.00) |
0.0382 (1.00) |
0.576 (1.00) |
|
| CCDC73 | 11 (6%) | 161 |
0.645 (1.00) |
0.597 (1.00) |
0.414 (1.00) |
0.258 (1.00) |
0.754 (1.00) |
0.512 (1.00) |
0.551 (1.00) |
0.0819 (1.00) |
1 (1.00) |
0.211 (1.00) |
0.656 (1.00) |
0.345 (1.00) |
|
| MGA | 16 (9%) | 156 |
0.451 (1.00) |
0.0209 (1.00) |
0.274 (1.00) |
0.742 (1.00) |
0.0709 (1.00) |
0.511 (1.00) |
0.437 (1.00) |
0.0441 (1.00) |
1 (1.00) |
0.742 (1.00) |
0.0879 (1.00) |
0.86 (1.00) |
|
| FAM48B1 | 10 (6%) | 162 |
0.925 (1.00) |
0.806 (1.00) |
0.712 (1.00) |
0.928 (1.00) |
0.396 (1.00) |
0.342 (1.00) |
0.111 (1.00) |
0.987 (1.00) |
1 (1.00) |
0.00191 (1.00) |
0.0797 (1.00) |
1 (1.00) |
|
| IFNA7 | 5 (3%) | 167 |
0.889 (1.00) |
0.78 (1.00) |
0.0155 (1.00) |
0.0561 (1.00) |
0.0727 (1.00) |
1 (1.00) |
0.662 (1.00) |
0.935 (1.00) |
0.37 (1.00) |
0.765 (1.00) |
0.38 (1.00) |
0.289 (1.00) |
|
| MLL3 | 34 (20%) | 138 |
0.563 (1.00) |
0.56 (1.00) |
0.941 (1.00) |
0.0856 (1.00) |
0.381 (1.00) |
0.854 (1.00) |
0.57 (1.00) |
0.548 (1.00) |
0.685 (1.00) |
0.5 (1.00) |
0.455 (1.00) |
0.834 (1.00) |
0.183 (1.00) |
| ASTN2 | 14 (8%) | 158 |
0.793 (1.00) |
0.76 (1.00) |
0.388 (1.00) |
0.457 (1.00) |
0.928 (1.00) |
0.25 (1.00) |
1 (1.00) |
0.000229 (0.484) |
1 (1.00) |
0.894 (1.00) |
0.888 (1.00) |
0.67 (1.00) |
|
| MBD1 | 6 (3%) | 166 |
0.536 (1.00) |
0.221 (1.00) |
0.0253 (1.00) |
0.142 (1.00) |
0.622 (1.00) |
1 (1.00) |
1 (1.00) |
0.979 (1.00) |
1 (1.00) |
0.173 (1.00) |
0.96 (1.00) |
1 (1.00) |
|
| FLI1 | 6 (3%) | 166 |
0.193 (1.00) |
0.566 (1.00) |
0.205 (1.00) |
0.212 (1.00) |
0.622 (1.00) |
1 (1.00) |
0.688 (1.00) |
1 (1.00) |
0.427 (1.00) |
0.888 (1.00) |
0.765 (1.00) |
0.354 (1.00) |
|
| LAX1 | 6 (3%) | 166 |
0.101 (1.00) |
0.468 (1.00) |
0.584 (1.00) |
0.0772 (1.00) |
0.622 (1.00) |
0.652 (1.00) |
0.688 (1.00) |
1 (1.00) |
1 (1.00) |
0.438 (1.00) |
1 (1.00) |
||
| SVOP | 7 (4%) | 165 |
0.771 (1.00) |
0.465 (1.00) |
0.584 (1.00) |
0.317 (1.00) |
0.135 (1.00) |
0.709 (1.00) |
0.704 (1.00) |
0.876 (1.00) |
1 (1.00) |
0.68 (1.00) |
0.338 (1.00) |
0.608 (1.00) |
|
| MTTP | 6 (3%) | 166 |
0.921 (1.00) |
0.145 (1.00) |
0.278 (1.00) |
0.416 (1.00) |
1 (1.00) |
0.379 (1.00) |
0.415 (1.00) |
0.982 (1.00) |
0.0869 (1.00) |
0.544 (1.00) |
0.145 (1.00) |
0.107 (1.00) |
|
| COL19A1 | 14 (8%) | 158 |
0.754 (1.00) |
0.276 (1.00) |
0.167 (1.00) |
0.342 (1.00) |
1 (1.00) |
0.707 (1.00) |
0.263 (1.00) |
0.0716 (1.00) |
0.00027 (0.571) |
0.615 (1.00) |
0.952 (1.00) |
0.676 (1.00) |
1 (1.00) |
| OR10G9 | 10 (6%) | 162 |
0.834 (1.00) |
0.0965 (1.00) |
0.335 (1.00) |
0.0946 (1.00) |
0.359 (1.00) |
0.605 (1.00) |
0.516 (1.00) |
0.989 (1.00) |
0.603 (1.00) |
0.489 (1.00) |
0.553 (1.00) |
0.527 (1.00) |
|
| LRRC32 | 11 (6%) | 161 |
0.85 (1.00) |
0.397 (1.00) |
0.926 (1.00) |
0.0247 (1.00) |
1 (1.00) |
0.652 (1.00) |
0.551 (1.00) |
0.552 (1.00) |
0.247 (1.00) |
0.819 (1.00) |
0.685 (1.00) |
0.455 (1.00) |
|
| TEX15 | 24 (14%) | 148 |
0.803 (1.00) |
0.351 (1.00) |
0.244 (1.00) |
0.154 (1.00) |
0.328 (1.00) |
1 (1.00) |
0.269 (1.00) |
0.797 (1.00) |
0.444 (1.00) |
0.396 (1.00) |
0.407 (1.00) |
0.369 (1.00) |
|
| CNGA2 | 11 (6%) | 161 |
0.532 (1.00) |
0.341 (1.00) |
0.479 (1.00) |
0.645 (1.00) |
0.0678 (1.00) |
0.792 (1.00) |
0.349 (1.00) |
0.158 (1.00) |
1 (1.00) |
0.85 (1.00) |
0.966 (1.00) |
0.796 (1.00) |
|
| ANP32C | 8 (5%) | 164 |
0.776 (1.00) |
0.473 (1.00) |
0.0197 (1.00) |
0.0384 (1.00) |
0.475 (1.00) |
1 (1.00) |
0.145 (1.00) |
0.34 (1.00) |
0.526 (1.00) |
0.146 (1.00) |
0.38 (1.00) |
1 (1.00) |
|
| KIAA1755 | 12 (7%) | 160 |
0.568 (1.00) |
0.385 (1.00) |
0.701 (1.00) |
0.248 (1.00) |
0.833 (1.00) |
1 (1.00) |
0.148 (1.00) |
0.245 (1.00) |
1 (1.00) |
0.42 (1.00) |
0.715 (1.00) |
0.419 (1.00) |
|
| OVCH1 | 14 (8%) | 158 |
0.8 (1.00) |
0.722 (1.00) |
0.138 (1.00) |
0.282 (1.00) |
0.928 (1.00) |
0.47 (1.00) |
0.171 (1.00) |
0.697 (1.00) |
1 (1.00) |
0.271 (1.00) |
0.327 (1.00) |
0.416 (1.00) |
|
| PJA1 | 8 (5%) | 164 |
0.101 (1.00) |
0.368 (1.00) |
0.579 (1.00) |
0.448 (1.00) |
0.693 (1.00) |
0.743 (1.00) |
0.728 (1.00) |
0.174 (1.00) |
1 (1.00) |
0.704 (1.00) |
0.808 (1.00) |
1 (1.00) |
|
| SUN1 | 8 (5%) | 164 |
0.25 (1.00) |
0.0693 (1.00) |
0.104 (1.00) |
0.489 (1.00) |
0.0969 (1.00) |
0.5 (1.00) |
0.728 (1.00) |
1 (1.00) |
1 (1.00) |
0.24 (1.00) |
0.885 (1.00) |
1 (1.00) |
|
| PABPC3 | 9 (5%) | 163 |
0.0864 (1.00) |
0.41 (1.00) |
0.197 (1.00) |
0.838 (1.00) |
0.585 (1.00) |
0.0663 (1.00) |
0.51 (1.00) |
0.977 (1.00) |
0.569 (1.00) |
0.548 (1.00) |
0.354 (1.00) |
0.244 (1.00) |
|
| STXBP5L | 12 (7%) | 160 |
0.146 (1.00) |
0.167 (1.00) |
0.105 (1.00) |
0.548 (1.00) |
0.264 (1.00) |
0.234 (1.00) |
0.148 (1.00) |
0.999 (1.00) |
0.603 (1.00) |
0.715 (1.00) |
0.286 (1.00) |
0.759 (1.00) |
|
| ELTD1 | 15 (9%) | 157 |
0.0283 (1.00) |
0.0397 (1.00) |
0.0643 (1.00) |
0.0324 (1.00) |
0.127 (1.00) |
0.42 (1.00) |
0.788 (1.00) |
0.146 (1.00) |
1 (1.00) |
0.811 (1.00) |
0.648 (1.00) |
0.419 (1.00) |
|
| KRT28 | 7 (4%) | 165 |
0.645 (1.00) |
0.925 (1.00) |
0.019 (1.00) |
0.0684 (1.00) |
0.0312 (1.00) |
0.203 (1.00) |
0.455 (1.00) |
0.000201 (0.424) |
0.116 (1.00) |
0.071 (1.00) |
0.499 (1.00) |
1 (1.00) |
|
| EPHA6 | 22 (13%) | 150 |
0.607 (1.00) |
0.673 (1.00) |
0.734 (1.00) |
0.299 (1.00) |
0.596 (1.00) |
0.492 (1.00) |
0.108 (1.00) |
0.953 (1.00) |
0.345 (1.00) |
1 (1.00) |
0.198 (1.00) |
0.714 (1.00) |
0.479 (1.00) |
| TLR4 | 23 (13%) | 149 |
0.96 (1.00) |
0.844 (1.00) |
0.117 (1.00) |
0.055 (1.00) |
0.453 (1.00) |
0.452 (1.00) |
1 (1.00) |
0.664 (1.00) |
0.428 (1.00) |
0.719 (1.00) |
0.855 (1.00) |
0.378 (1.00) |
|
| OR10R2 | 10 (6%) | 162 |
0.785 (1.00) |
0.623 (1.00) |
0.518 (1.00) |
0.427 (1.00) |
0.397 (1.00) |
0.774 (1.00) |
0.111 (1.00) |
0.443 (1.00) |
0.603 (1.00) |
0.308 (1.00) |
0.389 (1.00) |
1 (1.00) |
|
| OR6F1 | 10 (6%) | 162 |
0.416 (1.00) |
0.926 (1.00) |
0.497 (1.00) |
0.343 (1.00) |
0.236 (1.00) |
0.342 (1.00) |
1 (1.00) |
0.999 (1.00) |
0.603 (1.00) |
0.878 (1.00) |
0.957 (1.00) |
0.419 (1.00) |
|
| DACT1 | 10 (6%) | 162 |
0.151 (1.00) |
0.375 (1.00) |
0.221 (1.00) |
0.862 (1.00) |
0.00798 (1.00) |
1 (1.00) |
1 (1.00) |
0.585 (1.00) |
0.212 (1.00) |
0.225 (1.00) |
0.0671 (1.00) |
1 (1.00) |
|
| APC | 10 (6%) | 162 |
0.0154 (1.00) |
0.895 (1.00) |
0.472 (1.00) |
0.734 (1.00) |
0.721 (1.00) |
0.121 (1.00) |
1 (1.00) |
0.999 (1.00) |
0.212 (1.00) |
0.454 (1.00) |
0.0604 (1.00) |
0.345 (1.00) |
|
| SCG2 | 8 (5%) | 164 |
0.533 (1.00) |
0.622 (1.00) |
0.44 (1.00) |
0.182 (1.00) |
0.161 (1.00) |
1 (1.00) |
0.145 (1.00) |
0.0332 (1.00) |
0.146 (1.00) |
0.441 (1.00) |
0.978 (1.00) |
1 (1.00) |
|
| THEMIS | 10 (6%) | 162 |
0.0222 (1.00) |
0.991 (1.00) |
0.47 (1.00) |
0.398 (1.00) |
0.441 (1.00) |
0.342 (1.00) |
0.516 (1.00) |
0.999 (1.00) |
0.603 (1.00) |
0.768 (1.00) |
0.753 (1.00) |
0.759 (1.00) |
|
| BRE | 4 (2%) | 168 |
0.714 (1.00) |
0.568 (1.00) |
0.292 (1.00) |
0.021 (1.00) |
0.394 (1.00) |
1 (1.00) |
0.334 (1.00) |
1 (1.00) |
1 (1.00) |
0.61 (1.00) |
0.919 (1.00) |
1 (1.00) |
|
| SERPINB13 | 9 (5%) | 163 |
0.169 (1.00) |
0.69 (1.00) |
0.658 (1.00) |
0.471 (1.00) |
0.239 (1.00) |
1 (1.00) |
0.181 (1.00) |
0.0717 (1.00) |
0.179 (1.00) |
0.967 (1.00) |
0.296 (1.00) |
1 (1.00) |
|
| GATA3 | 6 (3%) | 166 |
0.0552 (1.00) |
0.0656 (1.00) |
0.364 (1.00) |
0.546 (1.00) |
0.467 (1.00) |
0.243 (1.00) |
0.415 (1.00) |
0.725 (1.00) |
0.427 (1.00) |
0.662 (1.00) |
0.892 (1.00) |
1 (1.00) |
|
| GNG2 | 4 (2%) | 168 |
0.857 (1.00) |
0.13 (1.00) |
0.756 (1.00) |
1 (1.00) |
0.815 (1.00) |
1 (1.00) |
1 (1.00) |
0.479 (1.00) |
1 (1.00) |
0.449 (1.00) |
1 (1.00) |
||
| TSHZ3 | 25 (15%) | 147 |
0.0168 (1.00) |
0.0263 (1.00) |
0.407 (1.00) |
0.947 (1.00) |
0.946 (1.00) |
0.482 (1.00) |
0.136 (1.00) |
0.279 (1.00) |
0.654 (1.00) |
1 (1.00) |
0.476 (1.00) |
0.0796 (1.00) |
0.397 (1.00) |
| FGB | 10 (6%) | 162 |
0.0867 (1.00) |
0.247 (1.00) |
0.641 (1.00) |
0.172 (1.00) |
1 (1.00) |
1 (1.00) |
0.755 (1.00) |
0.917 (1.00) |
1 (1.00) |
0.826 (1.00) |
0.459 (1.00) |
0.00971 (1.00) |
|
| PDGFA | 5 (3%) | 167 |
0.765 (1.00) |
0.907 (1.00) |
0.486 (1.00) |
0.395 (1.00) |
1 (1.00) |
1 (1.00) |
0.0191 (1.00) |
0.0208 (1.00) |
1 (1.00) |
0.793 (1.00) |
0.938 (1.00) |
0.37 (1.00) |
|
| F8 | 15 (9%) | 157 |
0.854 (1.00) |
0.0212 (1.00) |
0.524 (1.00) |
0.915 (1.00) |
0.385 (1.00) |
0.602 (1.00) |
0.418 (1.00) |
0.269 (1.00) |
1 (1.00) |
0.105 (1.00) |
0.785 (1.00) |
0.796 (1.00) |
|
| EPHB6 | 19 (11%) | 153 |
0.234 (1.00) |
0.194 (1.00) |
0.43 (1.00) |
0.277 (1.00) |
0.837 (1.00) |
0.0642 (1.00) |
0.81 (1.00) |
0.811 (1.00) |
0.673 (1.00) |
0.413 (1.00) |
0.56 (1.00) |
0.827 (1.00) |
|
| CTRC | 4 (2%) | 168 |
0.735 (1.00) |
0.99 (1.00) |
0.183 (1.00) |
0.281 (1.00) |
0.394 (1.00) |
0.503 (1.00) |
0.626 (1.00) |
0.997 (1.00) |
0.308 (1.00) |
1 (1.00) |
|||
| C12ORF74 | 3 (2%) | 169 |
0.966 (1.00) |
0.0114 (1.00) |
0.783 (1.00) |
1 (1.00) |
1 (1.00) |
0.194 (1.00) |
1 (1.00) |
0.119 (1.00) |
|||||
| DENND2A | 9 (5%) | 163 |
0.044 (1.00) |
0.463 (1.00) |
0.0564 (1.00) |
0.115 (1.00) |
0.721 (1.00) |
0.426 (1.00) |
0.0819 (1.00) |
0.977 (1.00) |
1 (1.00) |
0.694 (1.00) |
0.668 (1.00) |
1 (1.00) |
|
| POF1B | 9 (5%) | 163 |
0.132 (1.00) |
0.987 (1.00) |
0.714 (1.00) |
1 (1.00) |
0.254 (1.00) |
0.426 (1.00) |
0.181 (1.00) |
0.913 (1.00) |
1 (1.00) |
0.283 (1.00) |
0.521 (1.00) |
0.196 (1.00) |
|
| TMTC1 | 22 (13%) | 150 |
0.154 (1.00) |
0.293 (1.00) |
0.124 (1.00) |
0.153 (1.00) |
0.784 (1.00) |
0.438 (1.00) |
0.0695 (1.00) |
0.118 (1.00) |
0.413 (1.00) |
0.646 (1.00) |
0.355 (1.00) |
0.146 (1.00) |
|
| OR2F2 | 6 (3%) | 166 |
0.0809 (1.00) |
0.414 (1.00) |
0.474 (1.00) |
0.855 (1.00) |
0.177 (1.00) |
0.652 (1.00) |
1 (1.00) |
1 (1.00) |
0.427 (1.00) |
0.000666 (1.00) |
0.0115 (1.00) |
1 (1.00) |
|
| SPATA18 | 8 (5%) | 164 |
0.44 (1.00) |
0.507 (1.00) |
0.0255 (1.00) |
0.489 (1.00) |
0.0071 (1.00) |
1 (1.00) |
0.728 (1.00) |
0.34 (1.00) |
1 (1.00) |
0.44 (1.00) |
0.596 (1.00) |
1 (1.00) |
|
| FSCB | 15 (9%) | 157 |
0.321 (1.00) |
0.352 (1.00) |
0.854 (1.00) |
0.915 (1.00) |
0.805 (1.00) |
0.728 (1.00) |
0.596 (1.00) |
0.0884 (1.00) |
0.00453 (1.00) |
0.314 (1.00) |
0.311 (1.00) |
1 (1.00) |
|
| CDH6 | 17 (10%) | 155 |
0.302 (1.00) |
0.722 (1.00) |
0.822 (1.00) |
0.608 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.482 (1.00) |
0.368 (1.00) |
0.669 (1.00) |
0.0457 (1.00) |
0.0838 (1.00) |
|
| TTC18 | 5 (3%) | 167 |
0.818 (1.00) |
0.863 (1.00) |
0.0386 (1.00) |
0.28 (1.00) |
0.651 (1.00) |
0.00899 (1.00) |
0.063 (1.00) |
0.0928 (1.00) |
0.37 (1.00) |
0.497 (1.00) |
0.173 (1.00) |
||
| NALCN | 24 (14%) | 148 |
0.273 (1.00) |
0.0118 (1.00) |
0.501 (1.00) |
0.662 (1.00) |
0.518 (1.00) |
0.894 (1.00) |
0.667 (1.00) |
0.707 (1.00) |
1 (1.00) |
0.882 (1.00) |
0.946 (1.00) |
0.189 (1.00) |
|
| ABCB5 | 19 (11%) | 153 |
0.534 (1.00) |
0.946 (1.00) |
0.36 (1.00) |
0.0194 (1.00) |
0.508 (1.00) |
0.434 (1.00) |
0.332 (1.00) |
0.0391 (1.00) |
1 (1.00) |
0.0586 (1.00) |
0.462 (1.00) |
0.529 (1.00) |
|
| OR5I1 | 11 (6%) | 161 |
0.576 (1.00) |
0.898 (1.00) |
0.121 (1.00) |
0.0115 (1.00) |
0.262 (1.00) |
0.792 (1.00) |
0.115 (1.00) |
0.362 (1.00) |
1 (1.00) |
0.0815 (1.00) |
0.93 (1.00) |
0.455 (1.00) |
|
| COL5A1 | 13 (8%) | 159 |
0.925 (1.00) |
0.114 (1.00) |
0.408 (1.00) |
0.152 (1.00) |
0.49 (1.00) |
0.455 (1.00) |
0.262 (1.00) |
0.635 (1.00) |
1 (1.00) |
0.514 (1.00) |
0.546 (1.00) |
0.39 (1.00) |
|
| ITGAX | 17 (10%) | 155 |
0.709 (1.00) |
0.431 (1.00) |
0.0285 (1.00) |
0.0252 (1.00) |
0.554 (1.00) |
1 (1.00) |
0.8 (1.00) |
0.237 (1.00) |
1 (1.00) |
0.278 (1.00) |
0.254 (1.00) |
0.0655 (1.00) |
|
| SVEP1 | 21 (12%) | 151 |
0.236 (1.00) |
0.849 (1.00) |
0.0941 (1.00) |
0.177 (1.00) |
0.612 (1.00) |
1 (1.00) |
0.642 (1.00) |
0.609 (1.00) |
0.221 (1.00) |
0.404 (1.00) |
0.294 (1.00) |
0.17 (1.00) |
|
| FBXL7 | 17 (10%) | 155 |
0.682 (1.00) |
0.331 (1.00) |
0.086 (1.00) |
0.175 (1.00) |
0.83 (1.00) |
0.643 (1.00) |
0.127 (1.00) |
0.254 (1.00) |
1 (1.00) |
0.688 (1.00) |
0.518 (1.00) |
0.786 (1.00) |
|
| RP1L1 | 32 (19%) | 140 |
0.216 (1.00) |
0.458 (1.00) |
0.114 (1.00) |
0.163 (1.00) |
0.381 (1.00) |
0.771 (1.00) |
1 (1.00) |
0.65 (1.00) |
0.16 (1.00) |
0.998 (1.00) |
0.0539 (1.00) |
0.935 (1.00) |
|
| DEFB112 | 4 (2%) | 168 |
0.576 (1.00) |
0.0827 (1.00) |
0.656 (1.00) |
1 (1.00) |
0.651 (1.00) |
0.247 (1.00) |
0.334 (1.00) |
0.0187 (1.00) |
0.0386 (1.00) |
0.185 (1.00) |
0.264 (1.00) |
1 (1.00) |
|
| ARID2 | 12 (7%) | 160 |
0.158 (1.00) |
0.0417 (1.00) |
0.57 (1.00) |
0.397 (1.00) |
0.393 (1.00) |
0.419 (1.00) |
1 (1.00) |
0.999 (1.00) |
1 (1.00) |
0.622 (1.00) |
0.155 (1.00) |
0.827 (1.00) |
|
| ANKRD44 | 9 (5%) | 163 |
0.278 (1.00) |
0.573 (1.00) |
0.656 (1.00) |
0.508 (1.00) |
1 (1.00) |
0.426 (1.00) |
0.734 (1.00) |
0.913 (1.00) |
1 (1.00) |
0.488 (1.00) |
0.196 (1.00) |
||
| OR4C16 | 14 (8%) | 158 |
0.999 (1.00) |
0.525 (1.00) |
0.395 (1.00) |
0.906 (1.00) |
0.385 (1.00) |
0.707 (1.00) |
0.578 (1.00) |
0.00369 (1.00) |
1 (1.00) |
0.443 (1.00) |
0.885 (1.00) |
0.666 (1.00) |
|
| EPS8L3 | 4 (2%) | 168 |
0.281 (1.00) |
0.977 (1.00) |
0.0302 (1.00) |
0.11 (1.00) |
0.651 (1.00) |
1 (1.00) |
0.334 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| GPR158 | 17 (10%) | 155 |
0.908 (1.00) |
0.907 (1.00) |
0.354 (1.00) |
0.239 (1.00) |
0.938 (1.00) |
0.878 (1.00) |
0.446 (1.00) |
0.992 (1.00) |
1 (1.00) |
0.197 (1.00) |
0.802 (1.00) |
1 (1.00) |
|
| SPEG | 16 (9%) | 156 |
0.125 (1.00) |
0.98 (1.00) |
0.319 (1.00) |
0.96 (1.00) |
1 (1.00) |
0.511 (1.00) |
0.193 (1.00) |
0.33 (1.00) |
1 (1.00) |
0.926 (1.00) |
0.781 (1.00) |
0.505 (1.00) |
|
| TARS2 | 8 (5%) | 164 |
0.493 (1.00) |
0.522 (1.00) |
0.732 (1.00) |
0.265 (1.00) |
0.693 (1.00) |
0.254 (1.00) |
0.728 (1.00) |
1 (1.00) |
1 (1.00) |
0.314 (1.00) |
0.835 (1.00) |
0.15 (1.00) |
|
| OR2T27 | 8 (5%) | 164 |
0.233 (1.00) |
0.669 (1.00) |
0.866 (1.00) |
0.814 (1.00) |
0.693 (1.00) |
1 (1.00) |
0.728 (1.00) |
0.998 (1.00) |
1 (1.00) |
0.387 (1.00) |
0.092 (1.00) |
1 (1.00) |
|
| GPR174 | 6 (3%) | 166 |
0.516 (1.00) |
0.0537 (1.00) |
0.385 (1.00) |
1 (1.00) |
0.467 (1.00) |
0.155 (1.00) |
0.22 (1.00) |
0.00304 (1.00) |
1 (1.00) |
0.154 (1.00) |
0.137 (1.00) |
1 (1.00) |
|
| KIF9 | 7 (4%) | 165 |
0.532 (1.00) |
0.169 (1.00) |
0.00213 (1.00) |
0.183 (1.00) |
0.881 (1.00) |
0.709 (1.00) |
0.455 (1.00) |
0.306 (1.00) |
1 (1.00) |
0.174 (1.00) |
0.73 (1.00) |
1 (1.00) |
|
| C1ORF173 | 25 (15%) | 147 |
0.993 (1.00) |
0.15 (1.00) |
0.0573 (1.00) |
0.0335 (1.00) |
0.921 (1.00) |
1 (1.00) |
0.287 (1.00) |
0.864 (1.00) |
0.7 (1.00) |
0.57 (1.00) |
0.201 (1.00) |
1 (1.00) |
|
| CYP1A1 | 4 (2%) | 168 |
0.95 (1.00) |
0.747 (1.00) |
0.756 (1.00) |
1 (1.00) |
0.815 (1.00) |
1 (1.00) |
0.626 (1.00) |
0.0242 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| ENAH | 4 (2%) | 168 |
0.482 (1.00) |
0.371 (1.00) |
0.103 (1.00) |
0.226 (1.00) |
0.394 (1.00) |
1 (1.00) |
0.334 (1.00) |
0.997 (1.00) |
1 (1.00) |
0.557 (1.00) |
0.712 (1.00) |
1 (1.00) |
|
| SH2D2A | 6 (3%) | 166 |
0.246 (1.00) |
0.811 (1.00) |
0.411 (1.00) |
0.124 (1.00) |
0.497 (1.00) |
1 (1.00) |
0.688 (1.00) |
0.807 (1.00) |
1 (1.00) |
0.191 (1.00) |
0.23 (1.00) |
||
| CD5L | 12 (7%) | 160 |
0.751 (1.00) |
0.922 (1.00) |
0.095 (1.00) |
0.123 (1.00) |
0.922 (1.00) |
1 (1.00) |
0.229 (1.00) |
0.659 (1.00) |
0.281 (1.00) |
0.781 (1.00) |
0.914 (1.00) |
0.716 (1.00) |
|
| ANKRD56 | 11 (6%) | 161 |
0.318 (1.00) |
0.578 (1.00) |
0.118 (1.00) |
0.148 (1.00) |
0.433 (1.00) |
1 (1.00) |
0.756 (1.00) |
0.0201 (1.00) |
1 (1.00) |
0.458 (1.00) |
0.585 (1.00) |
0.419 (1.00) |
|
| HOXA5 | 6 (3%) | 166 |
0.148 (1.00) |
0.238 (1.00) |
0.604 (1.00) |
0.212 (1.00) |
0.622 (1.00) |
0.652 (1.00) |
0.0952 (1.00) |
0.979 (1.00) |
1 (1.00) |
0.0675 (1.00) |
0.851 (1.00) |
0.608 (1.00) |
|
| FERD3L | 7 (4%) | 165 |
0.0436 (1.00) |
0.164 (1.00) |
0.309 (1.00) |
0.23 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.116 (1.00) |
0.645 (1.00) |
0.881 (1.00) |
0.289 (1.00) |
|
| SIRPB1 | 6 (3%) | 166 |
0.11 (1.00) |
0.911 (1.00) |
0.192 (1.00) |
0.311 (1.00) |
0.0572 (1.00) |
0.155 (1.00) |
0.688 (1.00) |
0.0856 (1.00) |
1 (1.00) |
0.222 (1.00) |
0.514 (1.00) |
1 (1.00) |
|
| COL3A1 | 22 (13%) | 150 |
0.142 (1.00) |
0.449 (1.00) |
0.728 (1.00) |
0.767 (1.00) |
1 (1.00) |
0.709 (1.00) |
0.00561 (1.00) |
0.985 (1.00) |
1 (1.00) |
0.649 (1.00) |
0.0705 (1.00) |
0.709 (1.00) |
|
| GTF2I | 3 (2%) | 169 |
0.0418 (1.00) |
0.177 (1.00) |
0.235 (1.00) |
1 (1.00) |
0.594 (1.00) |
0.00172 (1.00) |
1 (1.00) |
1 (1.00) |
|||||
| RUFY2 | 6 (3%) | 166 |
0.0928 (1.00) |
0.531 (1.00) |
0.247 (1.00) |
0.416 (1.00) |
0.824 (1.00) |
0.0713 (1.00) |
1 (1.00) |
1 (1.00) |
0.427 (1.00) |
0.756 (1.00) |
0.909 (1.00) |
1 (1.00) |
|
| VPS13C | 19 (11%) | 153 |
0.465 (1.00) |
0.465 (1.00) |
0.911 (1.00) |
1 (1.00) |
0.944 (1.00) |
0.771 (1.00) |
0.144 (1.00) |
0.428 (1.00) |
0.379 (1.00) |
0.786 (1.00) |
0.446 (1.00) |
0.373 (1.00) |
|
| TBX15 | 11 (6%) | 161 |
0.346 (1.00) |
0.693 (1.00) |
0.00181 (1.00) |
0.0273 (1.00) |
0.699 (1.00) |
0.275 (1.00) |
0.756 (1.00) |
0.05 (1.00) |
1 (1.00) |
0.639 (1.00) |
0.871 (1.00) |
0.664 (1.00) |
|
| XIRP2 | 47 (27%) | 125 |
0.387 (1.00) |
0.622 (1.00) |
0.377 (1.00) |
0.489 (1.00) |
0.472 (1.00) |
0.805 (1.00) |
0.493 (1.00) |
0.392 (1.00) |
0.557 (1.00) |
0.153 (1.00) |
0.644 (1.00) |
0.735 (1.00) |
|
| HOXA3 | 6 (3%) | 166 |
0.723 (1.00) |
0.888 (1.00) |
0.234 (1.00) |
0.0104 (1.00) |
0.85 (1.00) |
0.155 (1.00) |
0.415 (1.00) |
0.00212 (1.00) |
1 (1.00) |
0.491 (1.00) |
0.571 (1.00) |
1 (1.00) |
|
| KDR | 20 (12%) | 152 |
0.234 (1.00) |
0.483 (1.00) |
0.765 (1.00) |
0.398 (1.00) |
0.238 (1.00) |
0.88 (1.00) |
0.638 (1.00) |
0.955 (1.00) |
0.479 (1.00) |
0.688 (1.00) |
0.256 (1.00) |
0.835 (1.00) |
0.894 (1.00) |
| OR56A4 | 6 (3%) | 166 |
0.422 (1.00) |
0.72 (1.00) |
0.181 (1.00) |
0.546 (1.00) |
0.105 (1.00) |
0.652 (1.00) |
0.415 (1.00) |
1 (1.00) |
1 (1.00) |
0.911 (1.00) |
0.646 (1.00) |
1 (1.00) |
|
| HCK | 6 (3%) | 166 |
0.177 (1.00) |
0.815 (1.00) |
0.284 (1.00) |
0.0925 (1.00) |
0.733 (1.00) |
1 (1.00) |
0.688 (1.00) |
1 (1.00) |
1 (1.00) |
0.435 (1.00) |
0.462 (1.00) |
||
| SPTA1 | 48 (28%) | 124 |
0.134 (1.00) |
0.281 (1.00) |
0.609 (1.00) |
0.881 (1.00) |
0.0477 (1.00) |
0.764 (1.00) |
0.501 (1.00) |
0.0327 (1.00) |
0.582 (1.00) |
0.24 (1.00) |
0.408 (1.00) |
0.189 (1.00) |
0.811 (1.00) |
| BRS3 | 7 (4%) | 165 |
0.495 (1.00) |
0.188 (1.00) |
0.309 (1.00) |
0.101 (1.00) |
0.24 (1.00) |
1 (1.00) |
1 (1.00) |
0.995 (1.00) |
1 (1.00) |
0.14 (1.00) |
0.638 (1.00) |
0.473 (1.00) |
|
| LPPR4 | 14 (8%) | 158 |
0.711 (1.00) |
0.753 (1.00) |
0.598 (1.00) |
0.906 (1.00) |
0.531 (1.00) |
1 (1.00) |
0.578 (1.00) |
0.347 (1.00) |
1 (1.00) |
0.541 (1.00) |
0.447 (1.00) |
1 (1.00) |
|
| KDM5C | 8 (5%) | 164 |
0.575 (1.00) |
0.754 (1.00) |
0.105 (1.00) |
0.0984 (1.00) |
0.792 (1.00) |
0.0982 (1.00) |
1 (1.00) |
0.646 (1.00) |
1 (1.00) |
0.136 (1.00) |
0.167 (1.00) |
0.244 (1.00) |
|
| TRPC1 | 7 (4%) | 165 |
0.27 (1.00) |
0.814 (1.00) |
0.702 (1.00) |
0.142 (1.00) |
0.653 (1.00) |
1 (1.00) |
0.704 (1.00) |
0.723 (1.00) |
0.478 (1.00) |
0.172 (1.00) |
0.756 (1.00) |
0.15 (1.00) |
|
| AOAH | 8 (5%) | 164 |
0.852 (1.00) |
0.688 (1.00) |
0.0327 (1.00) |
0.0584 (1.00) |
0.424 (1.00) |
1 (1.00) |
0.291 (1.00) |
0.115 (1.00) |
1 (1.00) |
0.715 (1.00) |
0.297 (1.00) |
0.287 (1.00) |
|
| C9ORF64 | 3 (2%) | 169 |
0.563 (1.00) |
0.0499 (1.00) |
0.818 (1.00) |
1 (1.00) |
0.578 (1.00) |
1 (1.00) |
0.0949 (1.00) |
0.999 (1.00) |
1 (1.00) |
0.623 (1.00) |
|||
| ZNF260 | 4 (2%) | 168 |
0.969 (1.00) |
0.915 (1.00) |
0.978 (1.00) |
0.178 (1.00) |
0.651 (1.00) |
1 (1.00) |
1 (1.00) |
0.997 (1.00) |
0.308 (1.00) |
0.532 (1.00) |
0.552 (1.00) |
0.0526 (1.00) |
|
| OLAH | 7 (4%) | 165 |
0.52 (1.00) |
0.257 (1.00) |
0.013 (1.00) |
0.0684 (1.00) |
0.653 (1.00) |
1 (1.00) |
0.704 (1.00) |
0.201 (1.00) |
0.478 (1.00) |
0.0403 (1.00) |
0.715 (1.00) |
1 (1.00) |
|
| GRK5 | 7 (4%) | 165 |
0.124 (1.00) |
0.302 (1.00) |
0.082 (1.00) |
0.391 (1.00) |
0.339 (1.00) |
1 (1.00) |
0.704 (1.00) |
0.306 (1.00) |
1 (1.00) |
0.109 (1.00) |
0.301 (1.00) |
0.608 (1.00) |
|
| ATM | 15 (9%) | 157 |
0.934 (1.00) |
0.327 (1.00) |
0.677 (1.00) |
0.299 (1.00) |
0.816 (1.00) |
0.602 (1.00) |
0.596 (1.00) |
0.821 (1.00) |
1 (1.00) |
0.471 (1.00) |
0.187 (1.00) |
1 (1.00) |
|
| C1ORF49 | 6 (3%) | 166 |
0.64 (1.00) |
0.754 (1.00) |
0.728 (1.00) |
0.361 (1.00) |
0.733 (1.00) |
0.155 (1.00) |
0.415 (1.00) |
0.255 (1.00) |
0.196 (1.00) |
1 (1.00) |
0.124 (1.00) |
0.952 (1.00) |
0.289 (1.00) |
| MFSD8 | 5 (3%) | 167 |
0.886 (1.00) |
0.0903 (1.00) |
0.205 (1.00) |
0.395 (1.00) |
0.824 (1.00) |
1 (1.00) |
0.662 (1.00) |
1 (1.00) |
1 (1.00) |
0.528 (1.00) |
0.186 (1.00) |
1 (1.00) |
|
| LELP1 | 3 (2%) | 169 |
0.729 (1.00) |
0.794 (1.00) |
0.177 (1.00) |
0.578 (1.00) |
1 (1.00) |
0.0949 (1.00) |
1 (1.00) |
1 (1.00) |
0.304 (1.00) |
0.881 (1.00) |
|||
| OR4A15 | 14 (8%) | 158 |
0.698 (1.00) |
0.286 (1.00) |
0.896 (1.00) |
0.272 (1.00) |
0.511 (1.00) |
0.845 (1.00) |
0.414 (1.00) |
0.801 (1.00) |
1 (1.00) |
0.332 (1.00) |
0.782 (1.00) |
0.848 (1.00) |
|
| FCRL4 | 7 (4%) | 165 |
0.202 (1.00) |
0.937 (1.00) |
0.043 (1.00) |
0.0623 (1.00) |
0.567 (1.00) |
1 (1.00) |
0.25 (1.00) |
1 (1.00) |
1 (1.00) |
0.344 (1.00) |
0.711 (1.00) |
0.608 (1.00) |
|
| NT5DC3 | 5 (3%) | 167 |
0.884 (1.00) |
0.709 (1.00) |
0.486 (1.00) |
0.134 (1.00) |
0.401 (1.00) |
1 (1.00) |
0.662 (1.00) |
0.0928 (1.00) |
0.37 (1.00) |
0.802 (1.00) |
0.173 (1.00) |
||
| PTH | 3 (2%) | 169 |
0.0105 (1.00) |
0.805 (1.00) |
0.112 (1.00) |
0.177 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.91 (1.00) |
0.841 (1.00) |
1 (1.00) |
||
| NAALAD2 | 9 (5%) | 163 |
0.156 (1.00) |
0.524 (1.00) |
0.273 (1.00) |
0.508 (1.00) |
0.33 (1.00) |
0.757 (1.00) |
0.734 (1.00) |
0.382 (1.00) |
0.569 (1.00) |
0.824 (1.00) |
0.484 (1.00) |
0.473 (1.00) |
|
| C8ORF37 | 6 (3%) | 166 |
0.988 (1.00) |
0.095 (1.00) |
0.438 (1.00) |
0.24 (1.00) |
0.544 (1.00) |
1 (1.00) |
1 (1.00) |
0.982 (1.00) |
0.427 (1.00) |
0.979 (1.00) |
0.954 (1.00) |
0.54 (1.00) |
|
| IL18RAP | 5 (3%) | 167 |
0.0341 (1.00) |
0.271 (1.00) |
0.267 (1.00) |
0.56 (1.00) |
0.135 (1.00) |
0.179 (1.00) |
0.662 (1.00) |
1 (1.00) |
1 (1.00) |
0.862 (1.00) |
1 (1.00) |
||
| STAC3 | 3 (2%) | 169 |
0.0405 (1.00) |
0.956 (1.00) |
0.0269 (1.00) |
0.00689 (1.00) |
0.235 (1.00) |
0.066 (1.00) |
1 (1.00) |
0.969 (1.00) |
1 (1.00) |
0.359 (1.00) |
|||
| TAAR5 | 7 (4%) | 165 |
0.163 (1.00) |
0.46 (1.00) |
0.718 (1.00) |
0.603 (1.00) |
0.446 (1.00) |
0.441 (1.00) |
1 (1.00) |
0.0327 (1.00) |
0.999 (1.00) |
0.116 (1.00) |
0.049 (1.00) |
0.25 (1.00) |
1 (1.00) |
| C2ORF39 | 8 (5%) | 164 |
0.73 (1.00) |
0.408 (1.00) |
0.446 (1.00) |
0.588 (1.00) |
0.331 (1.00) |
0.743 (1.00) |
0.728 (1.00) |
0.924 (1.00) |
1 (1.00) |
0.88 (1.00) |
0.403 (1.00) |
0.401 (1.00) |
|
| OR2G2 | 12 (7%) | 160 |
0.846 (1.00) |
0.373 (1.00) |
0.74 (1.00) |
0.548 (1.00) |
0.758 (1.00) |
0.81 (1.00) |
0.774 (1.00) |
0.659 (1.00) |
0.281 (1.00) |
0.62 (1.00) |
0.782 (1.00) |
1 (1.00) |
|
| BTK | 7 (4%) | 165 |
0.14 (1.00) |
0.487 (1.00) |
0.623 (1.00) |
0.603 (1.00) |
0.339 (1.00) |
1 (1.00) |
0.455 (1.00) |
0.995 (1.00) |
1 (1.00) |
0.93 (1.00) |
0.229 (1.00) |
1 (1.00) |
|
| ADAMTS2 | 14 (8%) | 158 |
0.866 (1.00) |
0.939 (1.00) |
0.459 (1.00) |
0.457 (1.00) |
0.673 (1.00) |
0.313 (1.00) |
0.414 (1.00) |
0.919 (1.00) |
1 (1.00) |
0.765 (1.00) |
0.783 (1.00) |
0.848 (1.00) |
|
| REG1B | 13 (8%) | 159 |
0.499 (1.00) |
0.0653 (1.00) |
0.497 (1.00) |
0.589 (1.00) |
0.264 (1.00) |
0.455 (1.00) |
1 (1.00) |
0.129 (1.00) |
0.725 (1.00) |
0.0902 (1.00) |
0.729 (1.00) |
0.141 (1.00) |
1 (1.00) |
| COL5A2 | 18 (10%) | 154 |
0.672 (1.00) |
0.91 (1.00) |
0.045 (1.00) |
0.115 (1.00) |
0.702 (1.00) |
0.299 (1.00) |
0.214 (1.00) |
0.418 (1.00) |
0.0552 (1.00) |
0.447 (1.00) |
0.999 (1.00) |
0.848 (1.00) |
|
| NHEDC1 | 9 (5%) | 163 |
0.177 (1.00) |
0.0893 (1.00) |
0.488 (1.00) |
0.437 (1.00) |
0.299 (1.00) |
0.426 (1.00) |
0.734 (1.00) |
0.000682 (1.00) |
1 (1.00) |
0.0254 (1.00) |
0.859 (1.00) |
1 (1.00) |
|
| DGKB | 11 (6%) | 161 |
0.568 (1.00) |
0.243 (1.00) |
0.558 (1.00) |
0.445 (1.00) |
0.441 (1.00) |
0.512 (1.00) |
0.756 (1.00) |
0.667 (1.00) |
0.247 (1.00) |
0.134 (1.00) |
0.433 (1.00) |
0.608 (1.00) |
|
| IL1RAPL1 | 11 (6%) | 161 |
0.956 (1.00) |
0.442 (1.00) |
0.623 (1.00) |
0.875 (1.00) |
0.478 (1.00) |
0.652 (1.00) |
0.229 (1.00) |
0.976 (1.00) |
1 (1.00) |
0.924 (1.00) |
0.216 (1.00) |
0.796 (1.00) |
|
| SEMA5A | 15 (9%) | 157 |
0.337 (1.00) |
0.555 (1.00) |
0.609 (1.00) |
0.915 (1.00) |
0.4 (1.00) |
0.728 (1.00) |
0.596 (1.00) |
0.143 (1.00) |
1 (1.00) |
0.291 (1.00) |
0.155 (1.00) |
0.117 (1.00) |
|
| COL25A1 | 11 (6%) | 161 |
0.938 (1.00) |
0.995 (1.00) |
0.397 (1.00) |
0.421 (1.00) |
0.758 (1.00) |
0.792 (1.00) |
1 (1.00) |
0.0331 (1.00) |
0.602 (1.00) |
0.217 (1.00) |
0.952 (1.00) |
0.342 (1.00) |
|
| BHMT | 5 (3%) | 167 |
0.332 (1.00) |
0.456 (1.00) |
0.068 (1.00) |
0.189 (1.00) |
0.599 (1.00) |
0.314 (1.00) |
0.662 (1.00) |
1 (1.00) |
1 (1.00) |
0.462 (1.00) |
|||
| CXCL6 | 4 (2%) | 168 |
0.573 (1.00) |
0.978 (1.00) |
0.257 (1.00) |
0.281 (1.00) |
0.815 (1.00) |
0.503 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.869 (1.00) |
1 (1.00) |
||
| MUC16 | 78 (45%) | 94 |
0.433 (1.00) |
0.404 (1.00) |
0.559 (1.00) |
0.0567 (1.00) |
0.342 (1.00) |
0.771 (1.00) |
0.443 (1.00) |
0.671 (1.00) |
0.451 (1.00) |
0.789 (1.00) |
0.0399 (1.00) |
0.605 (1.00) |
0.319 (1.00) |
| DYDC2 | 3 (2%) | 169 |
0.781 (1.00) |
0.383 (1.00) |
0.818 (1.00) |
1 (1.00) |
0.578 (1.00) |
1 (1.00) |
0.0949 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|||
| CLEC12B | 3 (2%) | 169 |
0.3 (1.00) |
0.915 (1.00) |
0.904 (1.00) |
1 (1.00) |
0.407 (1.00) |
0.251 (1.00) |
0.999 (1.00) |
1 (1.00) |
1 (1.00) |
||||
| CMA1 | 4 (2%) | 168 |
0.0842 (1.00) |
0.152 (1.00) |
0.00954 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.479 (1.00) |
1 (1.00) |
0.151 (1.00) |
0.173 (1.00) |
|||
| AQP10 | 6 (3%) | 166 |
0.161 (1.00) |
0.00133 (1.00) |
0.728 (1.00) |
0.361 (1.00) |
0.733 (1.00) |
1 (1.00) |
0.688 (1.00) |
0.0384 (1.00) |
0.427 (1.00) |
0.96 (1.00) |
0.166 (1.00) |
0.287 (1.00) |
|
| P2RX7 | 4 (2%) | 168 |
0.815 (1.00) |
0.83 (1.00) |
0.246 (1.00) |
0.281 (1.00) |
1 (1.00) |
0.0701 (1.00) |
0.626 (1.00) |
0.0115 (1.00) |
1 (1.00) |
0.509 (1.00) |
|||
| POLR3B | 11 (6%) | 161 |
0.853 (1.00) |
0.161 (1.00) |
0.66 (1.00) |
0.134 (1.00) |
1 (1.00) |
0.38 (1.00) |
0.551 (1.00) |
0.994 (1.00) |
1 (1.00) |
0.41 (1.00) |
0.964 (1.00) |
1 (1.00) |
|
| NETO1 | 14 (8%) | 158 |
0.559 (1.00) |
0.154 (1.00) |
0.157 (1.00) |
0.272 (1.00) |
0.277 (1.00) |
0.313 (1.00) |
0.263 (1.00) |
0.919 (1.00) |
1 (1.00) |
0.349 (1.00) |
0.979 (1.00) |
1 (1.00) |
|
| MGAT4C | 7 (4%) | 165 |
0.351 (1.00) |
0.172 (1.00) |
0.38 (1.00) |
0.685 (1.00) |
0.764 (1.00) |
0.709 (1.00) |
0.455 (1.00) |
0.963 (1.00) |
0.478 (1.00) |
0.767 (1.00) |
0.00406 (1.00) |
0.23 (1.00) |
|
| SLC34A2 | 8 (5%) | 164 |
0.00218 (1.00) |
0.19 (1.00) |
0.741 (1.00) |
0.167 (1.00) |
0.792 (1.00) |
0.743 (1.00) |
1 (1.00) |
0.924 (1.00) |
1 (1.00) |
0.722 (1.00) |
0.0193 (1.00) |
0.401 (1.00) |
|
| NTRK1 | 7 (4%) | 165 |
0.6 (1.00) |
0.349 (1.00) |
0.00495 (1.00) |
0.00242 (1.00) |
0.0563 (1.00) |
0.709 (1.00) |
0.704 (1.00) |
0.201 (1.00) |
1 (1.00) |
0.876 (1.00) |
0.227 (1.00) |
0.416 (1.00) |
|
| DYTN | 10 (6%) | 162 |
0.393 (1.00) |
0.00184 (1.00) |
0.4 (1.00) |
0.566 (1.00) |
0.0961 (1.00) |
0.47 (1.00) |
1 (1.00) |
0.0626 (1.00) |
0.603 (1.00) |
0.659 (1.00) |
0.151 (1.00) |
1 (1.00) |
|
| VEGFC | 9 (5%) | 163 |
0.987 (1.00) |
0.51 (1.00) |
0.911 (1.00) |
0.372 (1.00) |
0.897 (1.00) |
1 (1.00) |
1 (1.00) |
0.999 (1.00) |
1 (1.00) |
0.337 (1.00) |
0.401 (1.00) |
||
| GC | 5 (3%) | 167 |
0.348 (1.00) |
0.74 (1.00) |
0.418 (1.00) |
0.227 (1.00) |
0.599 (1.00) |
1 (1.00) |
0.0191 (1.00) |
0.0653 (1.00) |
0.37 (1.00) |
0.736 (1.00) |
0.99 (1.00) |
1 (1.00) |
|
| EHHADH | 4 (2%) | 168 |
0.864 (1.00) |
0.606 (1.00) |
0.36 (1.00) |
0.281 (1.00) |
1 (1.00) |
1 (1.00) |
0.334 (1.00) |
0.997 (1.00) |
1 (1.00) |
0.283 (1.00) |
1 (1.00) |
||
| ABCG5 | 9 (5%) | 163 |
0.00716 (1.00) |
0.521 (1.00) |
0.391 (1.00) |
0.00216 (1.00) |
0.458 (1.00) |
0.757 (1.00) |
0.734 (1.00) |
0.999 (1.00) |
1 (1.00) |
0.69 (1.00) |
1 (1.00) |
P value = 6.16e-07 (Chi-square test), Q value = 0.0013
Table S1. Gene #10: 'SMARCA4 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
| nPatients | LUNG ACINAR ADENOCARCINOMA | LUNG ADENOCARCINOMA MIXED SUBTYPE | LUNG ADENOCARCINOMA- NOT OTHERWISE SPECIFIED (NOS) | LUNG BRONCHIOLOALVEOLAR CARCINOMA MUCINOUS | LUNG BRONCHIOLOALVEOLAR CARCINOMA NONMUCINOUS | LUNG CLEAR CELL ADENOCARCINOMA | LUNG MICROPAPILLARY ADENOCARCINOMA | LUNG MUCINOUS ADENOCARCINOMA | LUNG PAPILLARY ADENOCARCINOMA | LUNG SOLID PATTERN PREDOMINANT ADENOCARCINOMA | MUCINOUS (COLLOID) CARCINOMA |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ALL | 2 | 39 | 115 | 1 | 2 | 2 | 2 | 2 | 4 | 1 | 2 |
| SMARCA4 MUTATED | 0 | 1 | 9 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 2 |
| SMARCA4 WILD-TYPE | 2 | 38 | 106 | 0 | 2 | 2 | 2 | 2 | 4 | 0 | 0 |
Figure S1. Get High-res Image Gene #10: 'SMARCA4 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
P value = 1.19e-08 (Chi-square test), Q value = 2.5e-05
Table S2. Gene #16: 'U2AF1 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
| nPatients | LUNG ACINAR ADENOCARCINOMA | LUNG ADENOCARCINOMA MIXED SUBTYPE | LUNG ADENOCARCINOMA- NOT OTHERWISE SPECIFIED (NOS) | LUNG BRONCHIOLOALVEOLAR CARCINOMA MUCINOUS | LUNG BRONCHIOLOALVEOLAR CARCINOMA NONMUCINOUS | LUNG CLEAR CELL ADENOCARCINOMA | LUNG MICROPAPILLARY ADENOCARCINOMA | LUNG MUCINOUS ADENOCARCINOMA | LUNG PAPILLARY ADENOCARCINOMA | LUNG SOLID PATTERN PREDOMINANT ADENOCARCINOMA | MUCINOUS (COLLOID) CARCINOMA |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ALL | 2 | 39 | 115 | 1 | 2 | 2 | 2 | 2 | 4 | 1 | 2 |
| U2AF1 MUTATED | 0 | 2 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| U2AF1 WILD-TYPE | 2 | 37 | 113 | 1 | 2 | 0 | 2 | 2 | 4 | 1 | 2 |
Figure S2. Get High-res Image Gene #16: 'U2AF1 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'

P value = 8.52e-07 (Chi-square test), Q value = 0.0018
Table S3. Gene #29: 'GBA3 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
| nPatients | LUNG ACINAR ADENOCARCINOMA | LUNG ADENOCARCINOMA MIXED SUBTYPE | LUNG ADENOCARCINOMA- NOT OTHERWISE SPECIFIED (NOS) | LUNG BRONCHIOLOALVEOLAR CARCINOMA MUCINOUS | LUNG BRONCHIOLOALVEOLAR CARCINOMA NONMUCINOUS | LUNG CLEAR CELL ADENOCARCINOMA | LUNG MICROPAPILLARY ADENOCARCINOMA | LUNG MUCINOUS ADENOCARCINOMA | LUNG PAPILLARY ADENOCARCINOMA | LUNG SOLID PATTERN PREDOMINANT ADENOCARCINOMA | MUCINOUS (COLLOID) CARCINOMA |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ALL | 2 | 39 | 115 | 1 | 2 | 2 | 2 | 2 | 4 | 1 | 2 |
| GBA3 MUTATED | 0 | 0 | 5 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 0 |
| GBA3 WILD-TYPE | 2 | 39 | 110 | 1 | 0 | 2 | 2 | 2 | 3 | 1 | 2 |
Figure S3. Get High-res Image Gene #29: 'GBA3 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
P value = 7.17e-06 (Chi-square test), Q value = 0.015
Table S4. Gene #149: 'OR13G1 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
| nPatients | LUNG ACINAR ADENOCARCINOMA | LUNG ADENOCARCINOMA MIXED SUBTYPE | LUNG ADENOCARCINOMA- NOT OTHERWISE SPECIFIED (NOS) | LUNG BRONCHIOLOALVEOLAR CARCINOMA MUCINOUS | LUNG BRONCHIOLOALVEOLAR CARCINOMA NONMUCINOUS | LUNG CLEAR CELL ADENOCARCINOMA | LUNG MICROPAPILLARY ADENOCARCINOMA | LUNG MUCINOUS ADENOCARCINOMA | LUNG PAPILLARY ADENOCARCINOMA | LUNG SOLID PATTERN PREDOMINANT ADENOCARCINOMA | MUCINOUS (COLLOID) CARCINOMA |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ALL | 2 | 39 | 115 | 1 | 2 | 2 | 2 | 2 | 4 | 1 | 2 |
| OR13G1 MUTATED | 0 | 3 | 3 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 0 |
| OR13G1 WILD-TYPE | 2 | 36 | 112 | 1 | 0 | 2 | 2 | 2 | 3 | 1 | 2 |
Figure S4. Get High-res Image Gene #149: 'OR13G1 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'

P value = 9.35e-06 (Chi-square test), Q value = 0.02
Table S5. Gene #165: 'AKR1B10 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
| nPatients | LUNG ACINAR ADENOCARCINOMA | LUNG ADENOCARCINOMA MIXED SUBTYPE | LUNG ADENOCARCINOMA- NOT OTHERWISE SPECIFIED (NOS) | LUNG BRONCHIOLOALVEOLAR CARCINOMA MUCINOUS | LUNG BRONCHIOLOALVEOLAR CARCINOMA NONMUCINOUS | LUNG CLEAR CELL ADENOCARCINOMA | LUNG MICROPAPILLARY ADENOCARCINOMA | LUNG MUCINOUS ADENOCARCINOMA | LUNG PAPILLARY ADENOCARCINOMA | LUNG SOLID PATTERN PREDOMINANT ADENOCARCINOMA | MUCINOUS (COLLOID) CARCINOMA |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ALL | 2 | 39 | 115 | 1 | 2 | 2 | 2 | 2 | 4 | 1 | 2 |
| AKR1B10 MUTATED | 0 | 0 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| AKR1B10 WILD-TYPE | 2 | 39 | 113 | 1 | 1 | 1 | 2 | 2 | 4 | 1 | 2 |
Figure S5. Get High-res Image Gene #165: 'AKR1B10 MUTATION STATUS' versus Clinical Feature #9: 'HISTOLOGICAL.TYPE'
P value = 2.46e-14 (t-test), Q value = 5.2e-11
Table S6. Gene #167: 'STX2 MUTATION STATUS' versus Clinical Feature #11: 'NUMBERPACKYEARSSMOKED'
| nPatients | Mean (Std.Dev) | |
|---|---|---|
| ALL | 112 | 40.7 (25.4) |
| STX2 MUTATED | 3 | 19.7 (0.6) |
| STX2 WILD-TYPE | 109 | 41.3 (25.5) |
Figure S6. Get High-res Image Gene #167: 'STX2 MUTATION STATUS' versus Clinical Feature #11: 'NUMBERPACKYEARSSMOKED'
-
Mutation data file = transformed.cor.cli.txt
-
Clinical data file = LUAD-TP.merged_data.txt
-
Number of patients = 172
-
Number of significantly mutated genes = 179
-
Number of selected clinical features = 13
-
Exclude genes that fewer than K tumors have mutations, K = 3
For survival clinical features, the Kaplan-Meier survival curves of tumors with and without gene mutations were plotted and the statistical significance P values were estimated by logrank test (Bland and Altman 2004) using the 'survdiff' function in R
For continuous numerical clinical features, two-tailed Student's t test with unequal variance (Lehmann and Romano 2005) was applied to compare the clinical values between tumors with and without gene mutations using 't.test' function in R
For multi-class clinical features (nominal or ordinal), Chi-square tests (Greenwood and Nikulin 1996) were used to estimate the P values using the 'chisq.test' function in R
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
In addition to the links below, the full results of the analysis summarized in this report can also be downloaded programmatically using firehose_get, or interactively from either the Broad GDAC website or TCGA Data Coordination Center Portal.