This pipeline computes the correlation between significant copy number variation (cnv focal) genes and selected clinical features.
Testing the association between copy number variation 54 focal events and 14 clinical features across 281 patients, one significant finding detected with Q value < 0.25.
-
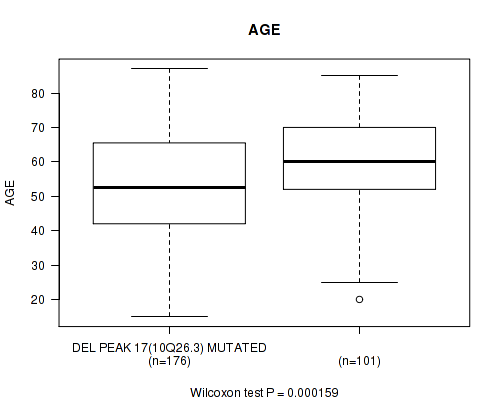
del_10q26.3 cnv correlated to 'AGE'.
Table 1. Get Full Table Overview of the association between significant copy number variation of 54 focal events and 14 clinical features. Shown in the table are P values (Q values). Thresholded by Q value < 0.25, one significant finding detected.
|
Clinical Features |
Time from Specimen Diagnosis to Death |
Time to Death |
AGE |
PRIMARY SITE OF DISEASE |
NEOPLASM DISEASESTAGE |
PATHOLOGY T STAGE |
PATHOLOGY N STAGE |
PATHOLOGY M STAGE |
MELANOMA ULCERATION |
MELANOMA PRIMARY KNOWN |
BRESLOW THICKNESS |
GENDER | RACE | ETHNICITY | ||
| nCNV (%) | nWild-Type | logrank test | logrank test | Wilcoxon-test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Wilcoxon-test | Fisher's exact test | Fisher's exact test | Fisher's exact test | |
| del 10q26 3 | 180 (64%) | 101 |
0.5 (1.00) |
0.275 (1.00) |
0.000159 (0.121) |
0.0213 (1.00) |
0.811 (1.00) |
0.327 (1.00) |
0.425 (1.00) |
0.124 (1.00) |
0.752 (1.00) |
0.849 (1.00) |
0.0621 (1.00) |
0.0552 (1.00) |
0.025 (1.00) |
0.286 (1.00) |
| amp 1p12 | 89 (32%) | 192 |
0.115 (1.00) |
0.0586 (1.00) |
0.803 (1.00) |
0.121 (1.00) |
0.468 (1.00) |
0.534 (1.00) |
0.99 (1.00) |
0.499 (1.00) |
0.417 (1.00) |
0.846 (1.00) |
0.534 (1.00) |
0.895 (1.00) |
0.763 (1.00) |
1 (1.00) |
| amp 1q21 3 | 148 (53%) | 133 |
0.789 (1.00) |
0.982 (1.00) |
0.893 (1.00) |
0.379 (1.00) |
0.711 (1.00) |
0.961 (1.00) |
0.661 (1.00) |
0.687 (1.00) |
0.647 (1.00) |
1 (1.00) |
0.855 (1.00) |
0.807 (1.00) |
0.824 (1.00) |
0.25 (1.00) |
| amp 1q44 | 152 (54%) | 129 |
0.489 (1.00) |
0.133 (1.00) |
0.821 (1.00) |
0.109 (1.00) |
0.441 (1.00) |
0.968 (1.00) |
0.895 (1.00) |
0.683 (1.00) |
0.445 (1.00) |
0.856 (1.00) |
0.901 (1.00) |
0.714 (1.00) |
0.818 (1.00) |
0.253 (1.00) |
| amp 3p13 | 76 (27%) | 205 |
0.014 (1.00) |
0.000814 (0.615) |
0.037 (1.00) |
0.735 (1.00) |
0.16 (1.00) |
0.176 (1.00) |
0.267 (1.00) |
0.69 (1.00) |
0.311 (1.00) |
0.417 (1.00) |
0.136 (1.00) |
0.0383 (1.00) |
0.719 (1.00) |
0.564 (1.00) |
| amp 4q12 | 52 (19%) | 229 |
0.375 (1.00) |
0.613 (1.00) |
0.515 (1.00) |
0.943 (1.00) |
0.254 (1.00) |
0.187 (1.00) |
0.827 (1.00) |
0.247 (1.00) |
0.696 (1.00) |
0.814 (1.00) |
0.918 (1.00) |
1 (1.00) |
1 (1.00) |
0.465 (1.00) |
| amp 5p15 33 | 89 (32%) | 192 |
0.622 (1.00) |
0.311 (1.00) |
0.363 (1.00) |
0.0263 (1.00) |
0.727 (1.00) |
0.409 (1.00) |
0.262 (1.00) |
0.275 (1.00) |
0.868 (1.00) |
0.432 (1.00) |
0.707 (1.00) |
0.895 (1.00) |
0.435 (1.00) |
1 (1.00) |
| amp 5q35 3 | 47 (17%) | 234 |
0.425 (1.00) |
0.42 (1.00) |
0.569 (1.00) |
0.0245 (1.00) |
0.936 (1.00) |
0.564 (1.00) |
0.605 (1.00) |
0.217 (1.00) |
0.303 (1.00) |
1 (1.00) |
0.152 (1.00) |
0.517 (1.00) |
0.665 (1.00) |
0.429 (1.00) |
| amp 6p24 3 | 159 (57%) | 122 |
0.00167 (1.00) |
0.121 (1.00) |
0.0762 (1.00) |
0.0689 (1.00) |
0.119 (1.00) |
0.23 (1.00) |
0.871 (1.00) |
0.338 (1.00) |
0.755 (1.00) |
1 (1.00) |
0.428 (1.00) |
0.711 (1.00) |
1 (1.00) |
1 (1.00) |
| amp 6q12 | 63 (22%) | 218 |
0.975 (1.00) |
0.995 (1.00) |
0.345 (1.00) |
0.0261 (1.00) |
0.0493 (1.00) |
0.461 (1.00) |
0.461 (1.00) |
0.836 (1.00) |
0.273 (1.00) |
0.829 (1.00) |
0.335 (1.00) |
0.241 (1.00) |
0.47 (1.00) |
1 (1.00) |
| amp 7p22 1 | 155 (55%) | 126 |
0.677 (1.00) |
0.712 (1.00) |
0.408 (1.00) |
0.718 (1.00) |
0.14 (1.00) |
0.222 (1.00) |
0.497 (1.00) |
0.559 (1.00) |
0.878 (1.00) |
0.142 (1.00) |
0.859 (1.00) |
0.902 (1.00) |
0.0402 (1.00) |
1 (1.00) |
| amp 7q34 | 169 (60%) | 112 |
0.73 (1.00) |
0.982 (1.00) |
0.301 (1.00) |
0.0153 (1.00) |
0.00628 (1.00) |
0.503 (1.00) |
0.224 (1.00) |
0.733 (1.00) |
0.434 (1.00) |
0.455 (1.00) |
0.994 (1.00) |
0.0812 (1.00) |
0.356 (1.00) |
0.566 (1.00) |
| amp 8q11 21 | 127 (45%) | 154 |
0.492 (1.00) |
0.184 (1.00) |
0.0151 (1.00) |
0.0267 (1.00) |
0.155 (1.00) |
0.314 (1.00) |
0.267 (1.00) |
0.442 (1.00) |
0.643 (1.00) |
0.1 (1.00) |
0.135 (1.00) |
0.538 (1.00) |
0.0187 (1.00) |
1 (1.00) |
| amp 8q24 21 | 146 (52%) | 135 |
0.919 (1.00) |
0.228 (1.00) |
0.011 (1.00) |
0.0769 (1.00) |
0.219 (1.00) |
0.2 (1.00) |
0.336 (1.00) |
0.755 (1.00) |
0.76 (1.00) |
0.273 (1.00) |
0.197 (1.00) |
0.464 (1.00) |
0.0435 (1.00) |
0.609 (1.00) |
| amp 11q13 3 | 54 (19%) | 227 |
0.533 (1.00) |
0.724 (1.00) |
0.321 (1.00) |
0.23 (1.00) |
0.0866 (1.00) |
0.058 (1.00) |
0.212 (1.00) |
0.845 (1.00) |
1 (1.00) |
0.818 (1.00) |
0.407 (1.00) |
0.756 (1.00) |
0.087 (1.00) |
1 (1.00) |
| amp 11q13 4 | 50 (18%) | 231 |
0.68 (1.00) |
0.641 (1.00) |
0.117 (1.00) |
0.0989 (1.00) |
0.56 (1.00) |
0.137 (1.00) |
0.0527 (1.00) |
0.553 (1.00) |
0.536 (1.00) |
1 (1.00) |
0.474 (1.00) |
1 (1.00) |
0.358 (1.00) |
1 (1.00) |
| amp 12q14 1 | 50 (18%) | 231 |
0.728 (1.00) |
0.307 (1.00) |
0.215 (1.00) |
0.609 (1.00) |
0.405 (1.00) |
0.435 (1.00) |
0.162 (1.00) |
0.708 (1.00) |
0.851 (1.00) |
0.0566 (1.00) |
0.277 (1.00) |
0.873 (1.00) |
0.359 (1.00) |
0.444 (1.00) |
| amp 12q15 | 48 (17%) | 233 |
0.526 (1.00) |
0.99 (1.00) |
0.205 (1.00) |
0.435 (1.00) |
0.655 (1.00) |
0.829 (1.00) |
0.145 (1.00) |
1 (1.00) |
0.695 (1.00) |
0.0863 (1.00) |
0.731 (1.00) |
1 (1.00) |
1 (1.00) |
0.429 (1.00) |
| amp 13q12 3 | 80 (28%) | 201 |
0.0407 (1.00) |
0.723 (1.00) |
0.433 (1.00) |
0.071 (1.00) |
0.305 (1.00) |
0.162 (1.00) |
0.0483 (1.00) |
0.522 (1.00) |
0.604 (1.00) |
1 (1.00) |
0.845 (1.00) |
1 (1.00) |
0.278 (1.00) |
0.561 (1.00) |
| amp 15q26 2 | 93 (33%) | 188 |
0.321 (1.00) |
0.124 (1.00) |
0.679 (1.00) |
0.434 (1.00) |
0.00365 (1.00) |
0.354 (1.00) |
0.646 (1.00) |
0.469 (1.00) |
0.174 (1.00) |
0.174 (1.00) |
0.316 (1.00) |
0.795 (1.00) |
1 (1.00) |
1 (1.00) |
| amp 17q25 3 | 96 (34%) | 185 |
0.6 (1.00) |
0.899 (1.00) |
0.029 (1.00) |
0.538 (1.00) |
0.525 (1.00) |
0.262 (1.00) |
0.96 (1.00) |
1 (1.00) |
0.417 (1.00) |
1 (1.00) |
0.785 (1.00) |
0.366 (1.00) |
0.566 (1.00) |
1 (1.00) |
| amp 19p13 2 | 53 (19%) | 228 |
0.857 (1.00) |
0.393 (1.00) |
0.565 (1.00) |
0.148 (1.00) |
0.0783 (1.00) |
0.623 (1.00) |
0.933 (1.00) |
0.00406 (1.00) |
0.444 (1.00) |
0.816 (1.00) |
0.343 (1.00) |
0.876 (1.00) |
1 (1.00) |
1 (1.00) |
| amp 20q13 33 | 147 (52%) | 134 |
0.611 (1.00) |
0.131 (1.00) |
0.0164 (1.00) |
0.718 (1.00) |
0.0542 (1.00) |
0.0573 (1.00) |
0.277 (1.00) |
0.0591 (1.00) |
0.128 (1.00) |
1 (1.00) |
0.0214 (1.00) |
0.715 (1.00) |
0.515 (1.00) |
0.607 (1.00) |
| amp 22q13 2 | 122 (43%) | 159 |
0.339 (1.00) |
0.501 (1.00) |
0.723 (1.00) |
0.139 (1.00) |
0.923 (1.00) |
0.168 (1.00) |
0.428 (1.00) |
0.969 (1.00) |
0.645 (1.00) |
1 (1.00) |
0.79 (1.00) |
0.46 (1.00) |
0.382 (1.00) |
0.261 (1.00) |
| del 1p36 31 | 68 (24%) | 213 |
0.27 (1.00) |
0.754 (1.00) |
0.218 (1.00) |
0.0803 (1.00) |
0.209 (1.00) |
0.471 (1.00) |
0.972 (1.00) |
0.199 (1.00) |
1 (1.00) |
0.831 (1.00) |
0.0848 (1.00) |
0.316 (1.00) |
1 (1.00) |
1 (1.00) |
| del 1p22 1 | 71 (25%) | 210 |
0.937 (1.00) |
0.6 (1.00) |
0.076 (1.00) |
0.654 (1.00) |
0.00452 (1.00) |
0.953 (1.00) |
0.126 (1.00) |
0.112 (1.00) |
0.0738 (1.00) |
0.836 (1.00) |
0.815 (1.00) |
0.324 (1.00) |
0.702 (1.00) |
0.574 (1.00) |
| del 2q37 3 | 69 (25%) | 212 |
0.101 (1.00) |
0.804 (1.00) |
0.638 (1.00) |
0.149 (1.00) |
0.957 (1.00) |
0.369 (1.00) |
0.351 (1.00) |
0.131 (1.00) |
0.292 (1.00) |
0.398 (1.00) |
0.337 (1.00) |
0.888 (1.00) |
1 (1.00) |
0.15 (1.00) |
| del 3p24 3 | 54 (19%) | 227 |
0.186 (1.00) |
0.126 (1.00) |
0.267 (1.00) |
0.381 (1.00) |
0.511 (1.00) |
0.04 (1.00) |
0.359 (1.00) |
1 (1.00) |
0.843 (1.00) |
0.818 (1.00) |
0.012 (1.00) |
0.214 (1.00) |
0.392 (1.00) |
1 (1.00) |
| del 3q23 | 46 (16%) | 235 |
0.586 (1.00) |
0.475 (1.00) |
0.395 (1.00) |
0.226 (1.00) |
0.286 (1.00) |
0.305 (1.00) |
0.385 (1.00) |
0.142 (1.00) |
0.843 (1.00) |
0.321 (1.00) |
0.463 (1.00) |
1 (1.00) |
0.324 (1.00) |
1 (1.00) |
| del 4q34 3 | 78 (28%) | 203 |
0.637 (1.00) |
0.83 (1.00) |
0.607 (1.00) |
0.254 (1.00) |
0.488 (1.00) |
0.736 (1.00) |
0.421 (1.00) |
0.266 (1.00) |
0.866 (1.00) |
0.31 (1.00) |
0.301 (1.00) |
0.0395 (1.00) |
0.725 (1.00) |
0.563 (1.00) |
| del 5p15 31 | 59 (21%) | 222 |
0.468 (1.00) |
0.753 (1.00) |
0.583 (1.00) |
0.772 (1.00) |
0.519 (1.00) |
0.0872 (1.00) |
0.856 (1.00) |
1 (1.00) |
0.00855 (1.00) |
0.184 (1.00) |
0.381 (1.00) |
0.229 (1.00) |
0.0188 (1.00) |
1 (1.00) |
| del 5q11 2 | 77 (27%) | 204 |
0.451 (1.00) |
0.449 (1.00) |
0.0885 (1.00) |
0.807 (1.00) |
0.413 (1.00) |
0.23 (1.00) |
0.638 (1.00) |
0.125 (1.00) |
0.165 (1.00) |
0.416 (1.00) |
0.883 (1.00) |
1 (1.00) |
1 (1.00) |
0.565 (1.00) |
| del 5q31 3 | 106 (38%) | 175 |
0.899 (1.00) |
0.768 (1.00) |
0.703 (1.00) |
0.907 (1.00) |
0.749 (1.00) |
0.143 (1.00) |
0.593 (1.00) |
0.134 (1.00) |
0.755 (1.00) |
0.0888 (1.00) |
0.525 (1.00) |
0.801 (1.00) |
1 (1.00) |
0.294 (1.00) |
| del 6q22 31 | 169 (60%) | 112 |
0.216 (1.00) |
0.846 (1.00) |
0.994 (1.00) |
0.357 (1.00) |
0.905 (1.00) |
0.484 (1.00) |
0.974 (1.00) |
0.972 (1.00) |
0.161 (1.00) |
0.709 (1.00) |
0.983 (1.00) |
0.456 (1.00) |
0.637 (1.00) |
0.566 (1.00) |
| del 6q26 | 174 (62%) | 107 |
0.217 (1.00) |
0.697 (1.00) |
0.9 (1.00) |
0.324 (1.00) |
0.654 (1.00) |
0.476 (1.00) |
0.931 (1.00) |
0.972 (1.00) |
0.344 (1.00) |
0.347 (1.00) |
0.534 (1.00) |
0.209 (1.00) |
0.106 (1.00) |
1 (1.00) |
| del 8p23 3 | 65 (23%) | 216 |
0.261 (1.00) |
0.998 (1.00) |
0.1 (1.00) |
0.34 (1.00) |
0.41 (1.00) |
0.0277 (1.00) |
0.318 (1.00) |
0.317 (1.00) |
0.466 (1.00) |
0.386 (1.00) |
0.0363 (1.00) |
0.663 (1.00) |
1 (1.00) |
1 (1.00) |
| del 9p23 | 191 (68%) | 90 |
0.0749 (1.00) |
0.607 (1.00) |
0.338 (1.00) |
0.606 (1.00) |
0.94 (1.00) |
0.712 (1.00) |
0.573 (1.00) |
0.0922 (1.00) |
1 (1.00) |
0.697 (1.00) |
0.769 (1.00) |
0.151 (1.00) |
0.769 (1.00) |
0.0324 (1.00) |
| del 9p21 3 | 214 (76%) | 67 |
0.0569 (1.00) |
0.995 (1.00) |
0.244 (1.00) |
0.0558 (1.00) |
0.869 (1.00) |
0.456 (1.00) |
0.723 (1.00) |
0.615 (1.00) |
0.86 (1.00) |
0.206 (1.00) |
0.404 (1.00) |
0.0614 (1.00) |
1 (1.00) |
0.0137 (1.00) |
| del 10p15 3 | 154 (55%) | 127 |
0.0912 (1.00) |
0.834 (1.00) |
0.0256 (1.00) |
0.0684 (1.00) |
0.94 (1.00) |
0.0833 (1.00) |
0.743 (1.00) |
0.841 (1.00) |
0.538 (1.00) |
0.856 (1.00) |
0.421 (1.00) |
0.268 (1.00) |
0.116 (1.00) |
0.589 (1.00) |
| del 10q23 31 | 175 (62%) | 106 |
0.522 (1.00) |
0.548 (1.00) |
0.0195 (1.00) |
0.198 (1.00) |
0.885 (1.00) |
0.192 (1.00) |
0.178 (1.00) |
0.289 (1.00) |
0.432 (1.00) |
0.574 (1.00) |
0.432 (1.00) |
0.0316 (1.00) |
0.24 (1.00) |
0.558 (1.00) |
| del 11p11 2 | 91 (32%) | 190 |
0.722 (1.00) |
0.636 (1.00) |
0.0203 (1.00) |
0.592 (1.00) |
0.35 (1.00) |
0.143 (1.00) |
0.812 (1.00) |
0.498 (1.00) |
0.332 (1.00) |
0.328 (1.00) |
0.397 (1.00) |
0.00575 (1.00) |
0.772 (1.00) |
0.553 (1.00) |
| del 11q23 3 | 154 (55%) | 127 |
0.644 (1.00) |
0.433 (1.00) |
0.331 (1.00) |
0.0833 (1.00) |
0.527 (1.00) |
0.605 (1.00) |
0.567 (1.00) |
0.879 (1.00) |
1 (1.00) |
0.856 (1.00) |
0.804 (1.00) |
0.176 (1.00) |
1 (1.00) |
0.593 (1.00) |
| del 12q23 3 | 68 (24%) | 213 |
0.88 (1.00) |
0.372 (1.00) |
0.721 (1.00) |
0.258 (1.00) |
0.935 (1.00) |
0.887 (1.00) |
0.602 (1.00) |
0.342 (1.00) |
1 (1.00) |
0.675 (1.00) |
0.752 (1.00) |
0.197 (1.00) |
0.501 (1.00) |
1 (1.00) |
| del 13q12 11 | 65 (23%) | 216 |
0.35 (1.00) |
0.576 (1.00) |
0.612 (1.00) |
0.743 (1.00) |
0.896 (1.00) |
0.114 (1.00) |
0.76 (1.00) |
0.0243 (1.00) |
0.862 (1.00) |
0.519 (1.00) |
0.132 (1.00) |
0.19 (1.00) |
0.69 (1.00) |
0.547 (1.00) |
| del 13q34 | 62 (22%) | 219 |
0.363 (1.00) |
0.48 (1.00) |
0.687 (1.00) |
0.267 (1.00) |
0.693 (1.00) |
0.559 (1.00) |
0.604 (1.00) |
0.0202 (1.00) |
0.602 (1.00) |
1 (1.00) |
0.679 (1.00) |
0.883 (1.00) |
0.68 (1.00) |
1 (1.00) |
| del 14q23 3 | 96 (34%) | 185 |
0.713 (1.00) |
0.45 (1.00) |
0.568 (1.00) |
0.099 (1.00) |
0.61 (1.00) |
0.6 (1.00) |
0.368 (1.00) |
0.294 (1.00) |
0.75 (1.00) |
1 (1.00) |
0.433 (1.00) |
0.44 (1.00) |
1 (1.00) |
0.553 (1.00) |
| del 14q32 2 | 95 (34%) | 186 |
0.687 (1.00) |
0.591 (1.00) |
0.551 (1.00) |
0.364 (1.00) |
0.299 (1.00) |
0.468 (1.00) |
0.26 (1.00) |
0.295 (1.00) |
0.747 (1.00) |
1 (1.00) |
0.347 (1.00) |
0.898 (1.00) |
1 (1.00) |
0.553 (1.00) |
| del 15q13 3 | 69 (25%) | 212 |
0.0789 (1.00) |
0.213 (1.00) |
0.451 (1.00) |
0.946 (1.00) |
0.58 (1.00) |
0.157 (1.00) |
0.654 (1.00) |
0.901 (1.00) |
0.862 (1.00) |
0.398 (1.00) |
0.697 (1.00) |
1 (1.00) |
0.696 (1.00) |
0.576 (1.00) |
| del 15q14 | 70 (25%) | 211 |
0.027 (1.00) |
0.172 (1.00) |
0.797 (1.00) |
0.964 (1.00) |
0.102 (1.00) |
0.206 (1.00) |
0.504 (1.00) |
0.568 (1.00) |
0.86 (1.00) |
0.398 (1.00) |
0.422 (1.00) |
0.778 (1.00) |
0.224 (1.00) |
1 (1.00) |
| del 15q15 2 | 73 (26%) | 208 |
0.102 (1.00) |
0.454 (1.00) |
0.666 (1.00) |
0.991 (1.00) |
0.211 (1.00) |
0.401 (1.00) |
0.497 (1.00) |
0.601 (1.00) |
0.726 (1.00) |
0.299 (1.00) |
0.367 (1.00) |
0.675 (1.00) |
0.242 (1.00) |
1 (1.00) |
| del 16p13 3 | 53 (19%) | 228 |
0.357 (1.00) |
0.657 (1.00) |
0.111 (1.00) |
0.383 (1.00) |
0.828 (1.00) |
0.549 (1.00) |
0.291 (1.00) |
0.731 (1.00) |
0.851 (1.00) |
0.0369 (1.00) |
0.71 (1.00) |
0.16 (1.00) |
0.228 (1.00) |
0.0948 (1.00) |
| del 16q12 1 | 92 (33%) | 189 |
0.769 (1.00) |
0.75 (1.00) |
0.417 (1.00) |
0.671 (1.00) |
0.416 (1.00) |
0.791 (1.00) |
0.358 (1.00) |
0.498 (1.00) |
0.873 (1.00) |
0.443 (1.00) |
0.507 (1.00) |
0.152 (1.00) |
0.553 (1.00) |
1 (1.00) |
| del 16q24 3 | 101 (36%) | 180 |
0.583 (1.00) |
0.645 (1.00) |
0.197 (1.00) |
0.523 (1.00) |
0.0756 (1.00) |
0.704 (1.00) |
0.276 (1.00) |
0.608 (1.00) |
0.752 (1.00) |
0.45 (1.00) |
0.488 (1.00) |
0.202 (1.00) |
0.341 (1.00) |
1 (1.00) |
| del 19p13 3 | 81 (29%) | 200 |
0.345 (1.00) |
0.228 (1.00) |
0.968 (1.00) |
0.532 (1.00) |
0.608 (1.00) |
0.621 (1.00) |
0.398 (1.00) |
0.226 (1.00) |
0.503 (1.00) |
0.842 (1.00) |
0.299 (1.00) |
0.223 (1.00) |
1 (1.00) |
0.559 (1.00) |
P value = 0.000159 (Wilcoxon-test), Q value = 0.12
Table S1. Gene #40: 'del_10q26.3' versus Clinical Feature #3: 'AGE'
| nPatients | Mean (Std.Dev) | |
|---|---|---|
| ALL | 277 | 55.8 (15.5) |
| DEL PEAK 17(10Q26.3) MUTATED | 176 | 53.1 (15.8) |
| DEL PEAK 17(10Q26.3) WILD-TYPE | 101 | 60.4 (13.8) |
Figure S1. Get High-res Image Gene #40: 'del_10q26.3' versus Clinical Feature #3: 'AGE'
-
Copy number data file = transformed.cor.cli.txt
-
Clinical data file = SKCM-TM.merged_data.txt
-
Number of patients = 281
-
Number of significantly focal cnvs = 54
-
Number of selected clinical features = 14
-
Exclude genes that fewer than K tumors have mutations, K = 3
For survival clinical features, the Kaplan-Meier survival curves of tumors with and without gene mutations were plotted and the statistical significance P values were estimated by logrank test (Bland and Altman 2004) using the 'survdiff' function in R
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
In addition to the links below, the full results of the analysis summarized in this report can also be downloaded programmatically using firehose_get, or interactively from either the Broad GDAC website or TCGA Data Coordination Center Portal.