This pipeline computes the correlation between significantly recurrent gene mutations and molecular subtypes.
Testing the association between mutation status of 129 genes and 8 molecular subtypes across 225 patients, 2 significant findings detected with P value < 0.05 and Q value < 0.25.
-
BRAF mutation correlated to 'CN_CNMF' and 'MRNASEQ_CHIERARCHICAL'.
Table 1. Get Full Table Overview of the association between mutation status of 129 genes and 8 molecular subtypes. Shown in the table are P values (Q values). Thresholded by P value < 0.05 and Q value < 0.25, 2 significant findings detected.
|
Clinical Features |
CN CNMF |
METHLYATION CNMF |
RPPA CNMF |
RPPA CHIERARCHICAL |
MRNASEQ CNMF |
MRNASEQ CHIERARCHICAL |
MIRSEQ CNMF |
MIRSEQ CHIERARCHICAL |
||
| nMutated (%) | nWild-Type | Fisher's exact test | Fisher's exact test | Fisher's exact test | Chi-square test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | |
| BRAF | 115 (51%) | 110 |
9.59e-08 (9.84e-05) |
0.0572 (1.00) |
0.0174 (1.00) |
0.00731 (1.00) |
0.00747 (1.00) |
1.94e-05 (0.0199) |
0.195 (1.00) |
0.0864 (1.00) |
| TP53 | 34 (15%) | 191 |
0.168 (1.00) |
0.0269 (1.00) |
0.356 (1.00) |
0.159 (1.00) |
0.181 (1.00) |
0.512 (1.00) |
0.283 (1.00) |
0.0584 (1.00) |
| C15ORF23 | 14 (6%) | 211 |
0.104 (1.00) |
0.215 (1.00) |
0.838 (1.00) |
0.815 (1.00) |
0.787 (1.00) |
1 (1.00) |
0.731 (1.00) |
0.719 (1.00) |
| CDKN2A | 31 (14%) | 194 |
0.153 (1.00) |
0.275 (1.00) |
0.417 (1.00) |
0.77 (1.00) |
0.652 (1.00) |
0.364 (1.00) |
0.753 (1.00) |
0.814 (1.00) |
| NRAS | 65 (29%) | 160 |
0.00053 (0.542) |
0.0383 (1.00) |
0.0899 (1.00) |
0.321 (1.00) |
0.898 (1.00) |
0.839 (1.00) |
0.863 (1.00) |
0.682 (1.00) |
| OXA1L | 6 (3%) | 219 |
0.288 (1.00) |
0.582 (1.00) |
0.617 (1.00) |
0.475 (1.00) |
0.679 (1.00) |
0.222 (1.00) |
0.24 (1.00) |
0.247 (1.00) |
| STK19 | 10 (4%) | 215 |
0.921 (1.00) |
0.133 (1.00) |
0.238 (1.00) |
0.999 (1.00) |
0.78 (1.00) |
0.491 (1.00) |
0.601 (1.00) |
0.718 (1.00) |
| PTEN | 18 (8%) | 207 |
0.674 (1.00) |
0.243 (1.00) |
0.149 (1.00) |
0.135 (1.00) |
0.952 (1.00) |
0.0688 (1.00) |
0.816 (1.00) |
0.346 (1.00) |
| RAC1 | 16 (7%) | 209 |
0.763 (1.00) |
0.946 (1.00) |
0.94 (1.00) |
0.45 (1.00) |
0.463 (1.00) |
0.942 (1.00) |
0.405 (1.00) |
0.224 (1.00) |
| ACSM2B | 36 (16%) | 189 |
0.454 (1.00) |
0.436 (1.00) |
0.815 (1.00) |
0.401 (1.00) |
0.441 (1.00) |
0.57 (1.00) |
0.153 (1.00) |
0.621 (1.00) |
| CADM2 | 22 (10%) | 203 |
0.66 (1.00) |
0.175 (1.00) |
0.153 (1.00) |
0.353 (1.00) |
0.816 (1.00) |
0.583 (1.00) |
0.345 (1.00) |
0.017 (1.00) |
| OR51S1 | 26 (12%) | 199 |
0.366 (1.00) |
0.649 (1.00) |
0.898 (1.00) |
0.113 (1.00) |
0.306 (1.00) |
0.795 (1.00) |
0.536 (1.00) |
0.79 (1.00) |
| LCE1B | 13 (6%) | 212 |
0.158 (1.00) |
0.626 (1.00) |
0.221 (1.00) |
0.892 (1.00) |
0.52 (1.00) |
0.707 (1.00) |
1 (1.00) |
1 (1.00) |
| IDH1 | 11 (5%) | 214 |
0.367 (1.00) |
0.18 (1.00) |
0.104 (1.00) |
0.281 (1.00) |
0.433 (1.00) |
0.715 (1.00) |
0.212 (1.00) |
0.107 (1.00) |
| TAF1A | 14 (6%) | 211 |
0.449 (1.00) |
0.692 (1.00) |
0.446 (1.00) |
0.055 (1.00) |
0.647 (1.00) |
0.243 (1.00) |
0.382 (1.00) |
0.719 (1.00) |
| NAP1L2 | 21 (9%) | 204 |
0.601 (1.00) |
0.74 (1.00) |
0.79 (1.00) |
0.809 (1.00) |
0.809 (1.00) |
1 (1.00) |
0.673 (1.00) |
1 (1.00) |
| MUC7 | 19 (8%) | 206 |
1 (1.00) |
0.468 (1.00) |
0.143 (1.00) |
0.706 (1.00) |
0.503 (1.00) |
0.0393 (1.00) |
0.0929 (1.00) |
0.0511 (1.00) |
| PPP6C | 17 (8%) | 208 |
0.508 (1.00) |
0.371 (1.00) |
0.0952 (1.00) |
0.78 (1.00) |
0.413 (1.00) |
0.542 (1.00) |
0.287 (1.00) |
0.151 (1.00) |
| FRG2B | 17 (8%) | 208 |
1 (1.00) |
0.773 (1.00) |
0.975 (1.00) |
0.997 (1.00) |
0.487 (1.00) |
0.945 (1.00) |
0.81 (1.00) |
0.326 (1.00) |
| USP29 | 35 (16%) | 190 |
0.92 (1.00) |
0.945 (1.00) |
0.883 (1.00) |
0.719 (1.00) |
0.918 (1.00) |
1 (1.00) |
0.306 (1.00) |
0.743 (1.00) |
| PRB4 | 18 (8%) | 207 |
0.385 (1.00) |
0.643 (1.00) |
0.137 (1.00) |
0.191 (1.00) |
0.0119 (1.00) |
0.191 (1.00) |
0.439 (1.00) |
0.394 (1.00) |
| HIST1H2AA | 10 (4%) | 215 |
0.363 (1.00) |
0.712 (1.00) |
0.655 (1.00) |
0.721 (1.00) |
0.335 (1.00) |
0.919 (1.00) |
0.343 (1.00) |
0.231 (1.00) |
| RPTN | 38 (17%) | 187 |
0.332 (1.00) |
0.167 (1.00) |
0.38 (1.00) |
0.367 (1.00) |
0.0182 (1.00) |
0.402 (1.00) |
0.471 (1.00) |
0.479 (1.00) |
| ZNF479 | 22 (10%) | 203 |
0.721 (1.00) |
1 (1.00) |
0.741 (1.00) |
0.112 (1.00) |
0.681 (1.00) |
0.546 (1.00) |
0.0685 (1.00) |
0.488 (1.00) |
| C8A | 26 (12%) | 199 |
0.701 (1.00) |
0.0835 (1.00) |
0.302 (1.00) |
0.394 (1.00) |
0.0971 (1.00) |
0.795 (1.00) |
0.284 (1.00) |
0.115 (1.00) |
| FUT9 | 22 (10%) | 203 |
0.288 (1.00) |
0.0387 (1.00) |
0.487 (1.00) |
0.85 (1.00) |
0.518 (1.00) |
0.109 (1.00) |
0.157 (1.00) |
0.61 (1.00) |
| ZNF679 | 18 (8%) | 207 |
0.558 (1.00) |
0.78 (1.00) |
0.632 (1.00) |
0.673 (1.00) |
0.235 (1.00) |
0.404 (1.00) |
0.347 (1.00) |
0.419 (1.00) |
| NRK | 28 (12%) | 197 |
0.904 (1.00) |
0.904 (1.00) |
0.632 (1.00) |
0.303 (1.00) |
0.289 (1.00) |
0.836 (1.00) |
0.758 (1.00) |
0.608 (1.00) |
| CDH9 | 32 (14%) | 193 |
0.888 (1.00) |
0.141 (1.00) |
0.251 (1.00) |
0.577 (1.00) |
0.888 (1.00) |
0.539 (1.00) |
0.00123 (1.00) |
0.288 (1.00) |
| PRAMEF11 | 21 (9%) | 204 |
0.808 (1.00) |
0.0585 (1.00) |
0.937 (1.00) |
0.96 (1.00) |
0.183 (1.00) |
0.498 (1.00) |
0.877 (1.00) |
0.711 (1.00) |
| DDX3X | 18 (8%) | 207 |
0.586 (1.00) |
0.953 (1.00) |
0.229 (1.00) |
0.174 (1.00) |
0.676 (1.00) |
0.95 (1.00) |
0.86 (1.00) |
0.211 (1.00) |
| GRXCR1 | 18 (8%) | 207 |
0.909 (1.00) |
0.067 (1.00) |
0.416 (1.00) |
0.95 (1.00) |
0.0351 (1.00) |
1 (1.00) |
0.671 (1.00) |
0.211 (1.00) |
| PRB2 | 29 (13%) | 196 |
0.555 (1.00) |
0.572 (1.00) |
0.773 (1.00) |
0.741 (1.00) |
0.00736 (1.00) |
0.0712 (1.00) |
0.171 (1.00) |
0.111 (1.00) |
| ELF5 | 7 (3%) | 218 |
0.711 (1.00) |
1 (1.00) |
0.933 (1.00) |
0.521 (1.00) |
0.344 (1.00) |
1 (1.00) |
0.0653 (1.00) |
0.0684 (1.00) |
| IL32 | 9 (4%) | 216 |
0.392 (1.00) |
0.114 (1.00) |
0.69 (1.00) |
0.149 (1.00) |
0.58 (1.00) |
1 (1.00) |
0.626 (1.00) |
1 (1.00) |
| LUZP2 | 19 (8%) | 206 |
1 (1.00) |
0.468 (1.00) |
0.205 (1.00) |
0.758 (1.00) |
1 (1.00) |
0.671 (1.00) |
0.0377 (1.00) |
0.466 (1.00) |
| PDE1A | 30 (13%) | 195 |
0.498 (1.00) |
0.909 (1.00) |
0.523 (1.00) |
0.384 (1.00) |
0.0374 (1.00) |
0.31 (1.00) |
0.559 (1.00) |
0.177 (1.00) |
| RBM11 | 12 (5%) | 213 |
0.186 (1.00) |
0.0599 (1.00) |
0.793 (1.00) |
0.872 (1.00) |
0.572 (1.00) |
0.0738 (1.00) |
0.308 (1.00) |
0.571 (1.00) |
| GLRB | 22 (10%) | 203 |
0.255 (1.00) |
0.513 (1.00) |
0.0532 (1.00) |
0.033 (1.00) |
0.46 (1.00) |
0.232 (1.00) |
0.41 (1.00) |
0.387 (1.00) |
| PRB1 | 21 (9%) | 204 |
0.503 (1.00) |
0.623 (1.00) |
0.127 (1.00) |
0.0897 (1.00) |
0.299 (1.00) |
0.602 (1.00) |
0.472 (1.00) |
0.208 (1.00) |
| KIAA1257 | 14 (6%) | 211 |
0.42 (1.00) |
1 (1.00) |
0.637 (1.00) |
0.775 (1.00) |
0.787 (1.00) |
0.765 (1.00) |
0.533 (1.00) |
0.48 (1.00) |
| USP17L2 | 18 (8%) | 207 |
0.261 (1.00) |
0.11 (1.00) |
0.185 (1.00) |
0.26 (1.00) |
0.127 (1.00) |
0.445 (1.00) |
0.383 (1.00) |
0.676 (1.00) |
| SPAG16 | 16 (7%) | 209 |
0.487 (1.00) |
1 (1.00) |
0.132 (1.00) |
0.289 (1.00) |
0.047 (1.00) |
0.044 (1.00) |
0.0604 (1.00) |
0.108 (1.00) |
| OR4M2 | 22 (10%) | 203 |
1 (1.00) |
0.96 (1.00) |
0.0678 (1.00) |
0.455 (1.00) |
0.298 (1.00) |
0.703 (1.00) |
0.132 (1.00) |
0.365 (1.00) |
| GFRAL | 28 (12%) | 197 |
0.34 (1.00) |
0.305 (1.00) |
0.681 (1.00) |
0.764 (1.00) |
0.905 (1.00) |
0.836 (1.00) |
0.873 (1.00) |
0.956 (1.00) |
| UNC119B | 6 (3%) | 219 |
0.448 (1.00) |
0.398 (1.00) |
0.215 (1.00) |
0.915 (1.00) |
0.592 (1.00) |
0.551 (1.00) |
1 (1.00) |
0.468 (1.00) |
| PHGDH | 8 (4%) | 217 |
0.668 (1.00) |
1 (1.00) |
1 (1.00) |
0.425 (1.00) |
0.903 (1.00) |
0.894 (1.00) |
0.731 (1.00) |
0.425 (1.00) |
| STXBP5L | 42 (19%) | 183 |
0.37 (1.00) |
0.93 (1.00) |
0.837 (1.00) |
0.617 (1.00) |
0.0957 (1.00) |
0.333 (1.00) |
0.462 (1.00) |
0.559 (1.00) |
| TRAT1 | 12 (5%) | 213 |
0.932 (1.00) |
0.365 (1.00) |
0.732 (1.00) |
0.679 (1.00) |
0.433 (1.00) |
1 (1.00) |
0.219 (1.00) |
0.466 (1.00) |
| ZNF844 | 9 (4%) | 216 |
1 (1.00) |
1 (1.00) |
0.655 (1.00) |
0.915 (1.00) |
0.821 (1.00) |
0.724 (1.00) |
0.564 (1.00) |
0.78 (1.00) |
| TTN | 171 (76%) | 54 |
0.403 (1.00) |
0.0198 (1.00) |
0.415 (1.00) |
0.0912 (1.00) |
0.125 (1.00) |
0.181 (1.00) |
0.156 (1.00) |
0.276 (1.00) |
| COPG2 | 9 (4%) | 216 |
0.319 (1.00) |
0.914 (1.00) |
0.287 (1.00) |
0.251 (1.00) |
0.693 (1.00) |
0.618 (1.00) |
0.908 (1.00) |
0.78 (1.00) |
| DSG3 | 41 (18%) | 184 |
0.54 (1.00) |
0.976 (1.00) |
0.782 (1.00) |
0.302 (1.00) |
0.597 (1.00) |
0.299 (1.00) |
0.345 (1.00) |
0.654 (1.00) |
| GML | 9 (4%) | 216 |
0.176 (1.00) |
0.387 (1.00) |
0.94 (1.00) |
0.418 (1.00) |
0.835 (1.00) |
0.903 (1.00) |
0.181 (1.00) |
0.78 (1.00) |
| HHLA2 | 14 (6%) | 211 |
0.104 (1.00) |
0.47 (1.00) |
0.434 (1.00) |
0.0038 (1.00) |
1 (1.00) |
0.108 (1.00) |
0.111 (1.00) |
0.66 (1.00) |
| LILRB4 | 27 (12%) | 198 |
0.214 (1.00) |
1 (1.00) |
0.224 (1.00) |
0.668 (1.00) |
0.556 (1.00) |
0.188 (1.00) |
0.528 (1.00) |
0.543 (1.00) |
| SLC38A4 | 25 (11%) | 200 |
0.00845 (1.00) |
0.772 (1.00) |
0.983 (1.00) |
0.962 (1.00) |
0.0403 (1.00) |
0.725 (1.00) |
0.713 (1.00) |
0.709 (1.00) |
| OR2L3 | 14 (6%) | 211 |
0.609 (1.00) |
0.0148 (1.00) |
0.745 (1.00) |
0.468 (1.00) |
0.739 (1.00) |
0.32 (1.00) |
0.775 (1.00) |
0.517 (1.00) |
| HBD | 9 (4%) | 216 |
0.58 (1.00) |
1 (1.00) |
0.832 (1.00) |
0.241 (1.00) |
0.054 (1.00) |
0.253 (1.00) |
0.507 (1.00) |
0.889 (1.00) |
| TBC1D3B | 5 (2%) | 220 |
0.226 (1.00) |
0.745 (1.00) |
0.0606 (1.00) |
0.258 (1.00) |
0.86 (1.00) |
1 (1.00) |
0.856 (1.00) |
0.645 (1.00) |
| TCEB3C | 26 (12%) | 199 |
0.524 (1.00) |
0.313 (1.00) |
0.343 (1.00) |
0.543 (1.00) |
0.246 (1.00) |
0.0572 (1.00) |
0.694 (1.00) |
0.242 (1.00) |
| CYLC2 | 19 (8%) | 206 |
0.354 (1.00) |
0.569 (1.00) |
0.0934 (1.00) |
0.121 (1.00) |
0.207 (1.00) |
0.122 (1.00) |
0.297 (1.00) |
0.339 (1.00) |
| HBG2 | 7 (3%) | 218 |
0.34 (1.00) |
0.558 (1.00) |
0.234 (1.00) |
0.339 (1.00) |
0.298 (1.00) |
1 (1.00) |
0.229 (1.00) |
0.302 (1.00) |
| LOC649330 | 22 (10%) | 203 |
0.245 (1.00) |
0.472 (1.00) |
0.791 (1.00) |
0.756 (1.00) |
0.0756 (1.00) |
0.767 (1.00) |
0.714 (1.00) |
0.434 (1.00) |
| FIGLA | 3 (1%) | 222 |
0.632 (1.00) |
0.628 (1.00) |
0.512 (1.00) |
0.432 (1.00) |
0.612 (1.00) |
0.691 (1.00) |
||
| PARM1 | 17 (8%) | 208 |
0.856 (1.00) |
1 (1.00) |
0.726 (1.00) |
0.131 (1.00) |
0.775 (1.00) |
0.674 (1.00) |
0.195 (1.00) |
0.46 (1.00) |
| CCK | 6 (3%) | 219 |
0.584 (1.00) |
0.662 (1.00) |
0.322 (1.00) |
0.0724 (1.00) |
0.447 (1.00) |
0.879 (1.00) |
0.577 (1.00) |
0.714 (1.00) |
| ZFP106 | 10 (4%) | 215 |
0.85 (1.00) |
0.777 (1.00) |
0.264 (1.00) |
0.264 (1.00) |
0.512 (1.00) |
0.919 (1.00) |
0.552 (1.00) |
0.449 (1.00) |
| UGT2B15 | 19 (8%) | 206 |
0.336 (1.00) |
0.382 (1.00) |
0.767 (1.00) |
0.734 (1.00) |
0.431 (1.00) |
1 (1.00) |
0.357 (1.00) |
0.466 (1.00) |
| FGF16 | 7 (3%) | 218 |
1 (1.00) |
0.897 (1.00) |
0.00479 (1.00) |
0.0142 (1.00) |
1 (1.00) |
0.154 (1.00) |
0.0929 (1.00) |
0.528 (1.00) |
| NUDT4 | 5 (2%) | 220 |
1 (1.00) |
0.375 (1.00) |
0.361 (1.00) |
0.145 (1.00) |
1 (1.00) |
0.839 (1.00) |
0.612 (1.00) |
0.503 (1.00) |
| DSG1 | 37 (16%) | 188 |
0.604 (1.00) |
0.524 (1.00) |
0.0841 (1.00) |
0.0575 (1.00) |
0.333 (1.00) |
0.771 (1.00) |
0.315 (1.00) |
0.546 (1.00) |
| ARL16 | 7 (3%) | 218 |
0.18 (1.00) |
0.344 (1.00) |
0.14 (1.00) |
0.649 (1.00) |
0.395 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| VEGFC | 16 (7%) | 209 |
0.649 (1.00) |
0.143 (1.00) |
0.349 (1.00) |
0.0255 (1.00) |
0.165 (1.00) |
0.319 (1.00) |
0.00821 (1.00) |
0.0406 (1.00) |
| PSG4 | 23 (10%) | 202 |
0.489 (1.00) |
0.961 (1.00) |
0.426 (1.00) |
0.387 (1.00) |
0.649 (1.00) |
0.713 (1.00) |
0.268 (1.00) |
0.119 (1.00) |
| DEFB119 | 6 (3%) | 219 |
1 (1.00) |
0.771 (1.00) |
0.493 (1.00) |
0.885 (1.00) |
0.876 (1.00) |
0.879 (1.00) |
0.437 (1.00) |
1 (1.00) |
| KLHL4 | 20 (9%) | 205 |
0.799 (1.00) |
0.532 (1.00) |
0.762 (1.00) |
0.863 (1.00) |
0.213 (1.00) |
0.208 (1.00) |
0.955 (1.00) |
0.482 (1.00) |
| EIF3D | 4 (2%) | 221 |
0.832 (1.00) |
0.837 (1.00) |
0.246 (1.00) |
0.436 (1.00) |
1 (1.00) |
0.367 (1.00) |
0.469 (1.00) |
0.43 (1.00) |
| OR2W1 | 16 (7%) | 209 |
0.157 (1.00) |
0.719 (1.00) |
0.244 (1.00) |
0.0888 (1.00) |
0.633 (1.00) |
1 (1.00) |
0.515 (1.00) |
1 (1.00) |
| OR4N2 | 21 (9%) | 204 |
0.34 (1.00) |
0.144 (1.00) |
0.64 (1.00) |
0.364 (1.00) |
0.0282 (1.00) |
0.242 (1.00) |
0.43 (1.00) |
0.0559 (1.00) |
| CCDC11 | 17 (8%) | 208 |
0.903 (1.00) |
0.0644 (1.00) |
0.265 (1.00) |
0.212 (1.00) |
0.0802 (1.00) |
0.126 (1.00) |
0.0273 (1.00) |
1 (1.00) |
| LIN7A | 10 (4%) | 215 |
0.921 (1.00) |
0.267 (1.00) |
0.556 (1.00) |
0.648 (1.00) |
0.176 (1.00) |
0.693 (1.00) |
0.0225 (1.00) |
0.395 (1.00) |
| RAPGEF5 | 11 (5%) | 214 |
1 (1.00) |
0.459 (1.00) |
0.604 (1.00) |
0.494 (1.00) |
0.311 (1.00) |
0.607 (1.00) |
0.274 (1.00) |
0.0629 (1.00) |
| CD2 | 16 (7%) | 209 |
0.395 (1.00) |
0.579 (1.00) |
0.776 (1.00) |
0.516 (1.00) |
1 (1.00) |
0.839 (1.00) |
0.671 (1.00) |
0.514 (1.00) |
| C2ORF40 | 7 (3%) | 218 |
0.236 (1.00) |
0.798 (1.00) |
0.94 (1.00) |
0.725 (1.00) |
0.894 (1.00) |
0.788 (1.00) |
0.429 (1.00) |
0.629 (1.00) |
| SGCZ | 19 (8%) | 206 |
0.626 (1.00) |
0.625 (1.00) |
0.866 (1.00) |
0.7 (1.00) |
0.278 (1.00) |
0.0419 (1.00) |
0.953 (1.00) |
0.784 (1.00) |
| SPANXN2 | 14 (6%) | 211 |
0.283 (1.00) |
0.692 (1.00) |
0.584 (1.00) |
0.171 (1.00) |
0.302 (1.00) |
0.674 (1.00) |
0.607 (1.00) |
0.106 (1.00) |
| TLL1 | 42 (19%) | 183 |
0.976 (1.00) |
0.0183 (1.00) |
0.618 (1.00) |
0.0421 (1.00) |
0.084 (1.00) |
0.149 (1.00) |
0.821 (1.00) |
0.403 (1.00) |
| C9ORF119 | 7 (3%) | 218 |
1 (1.00) |
0.178 (1.00) |
0.476 (1.00) |
0.563 (1.00) |
0.344 (1.00) |
0.154 (1.00) |
0.379 (1.00) |
0.528 (1.00) |
| CCDC54 | 13 (6%) | 212 |
0.0812 (1.00) |
0.334 (1.00) |
0.294 (1.00) |
0.863 (1.00) |
0.11 (1.00) |
0.0875 (1.00) |
0.0879 (1.00) |
0.772 (1.00) |
| DEFB118 | 8 (4%) | 217 |
0.197 (1.00) |
0.316 (1.00) |
0.933 (1.00) |
0.269 (1.00) |
0.667 (1.00) |
0.724 (1.00) |
0.38 (1.00) |
0.668 (1.00) |
| HTR3B | 16 (7%) | 209 |
0.0169 (1.00) |
0.135 (1.00) |
0.0261 (1.00) |
0.0598 (1.00) |
0.000541 (0.554) |
0.0202 (1.00) |
0.283 (1.00) |
0.224 (1.00) |
| MARCH11 | 10 (4%) | 215 |
0.85 (1.00) |
1 (1.00) |
0.378 (1.00) |
0.721 (1.00) |
0.176 (1.00) |
0.166 (1.00) |
1 (1.00) |
0.891 (1.00) |
| MKX | 14 (6%) | 211 |
0.139 (1.00) |
0.2 (1.00) |
0.583 (1.00) |
0.971 (1.00) |
0.0691 (1.00) |
0.382 (1.00) |
0.196 (1.00) |
0.208 (1.00) |
| OR5H2 | 14 (6%) | 211 |
0.783 (1.00) |
1 (1.00) |
0.458 (1.00) |
0.518 (1.00) |
0.739 (1.00) |
0.674 (1.00) |
0.318 (1.00) |
0.208 (1.00) |
| SIGLEC14 | 11 (5%) | 214 |
0.367 (1.00) |
0.338 (1.00) |
0.0358 (1.00) |
0.464 (1.00) |
0.581 (1.00) |
0.715 (1.00) |
0.625 (1.00) |
0.81 (1.00) |
| TUBB8 | 12 (5%) | 213 |
0.704 (1.00) |
0.867 (1.00) |
0.214 (1.00) |
0.138 (1.00) |
0.542 (1.00) |
0.302 (1.00) |
0.284 (1.00) |
0.25 (1.00) |
| ZIM3 | 23 (10%) | 202 |
0.194 (1.00) |
0.14 (1.00) |
0.675 (1.00) |
0.431 (1.00) |
0.208 (1.00) |
0.879 (1.00) |
0.64 (1.00) |
0.69 (1.00) |
| PRC1 | 13 (6%) | 212 |
0.938 (1.00) |
0.42 (1.00) |
0.0564 (1.00) |
0.716 (1.00) |
0.279 (1.00) |
0.56 (1.00) |
0.291 (1.00) |
0.458 (1.00) |
| MUM1L1 | 25 (11%) | 200 |
0.555 (1.00) |
0.802 (1.00) |
0.738 (1.00) |
0.0918 (1.00) |
0.597 (1.00) |
0.133 (1.00) |
0.589 (1.00) |
0.781 (1.00) |
| TRIM58 | 15 (7%) | 210 |
0.561 (1.00) |
0.891 (1.00) |
0.815 (1.00) |
0.861 (1.00) |
0.562 (1.00) |
0.687 (1.00) |
0.432 (1.00) |
0.861 (1.00) |
| ANKRD20A4 | 4 (2%) | 221 |
0.385 (1.00) |
0.454 (1.00) |
0.322 (1.00) |
0.135 (1.00) |
0.106 (1.00) |
0.809 (1.00) |
0.469 (1.00) |
1 (1.00) |
| OR5J2 | 13 (6%) | 212 |
0.633 (1.00) |
0.55 (1.00) |
0.312 (1.00) |
0.5 (1.00) |
0.938 (1.00) |
1 (1.00) |
0.107 (1.00) |
0.458 (1.00) |
| CLEC14A | 18 (8%) | 207 |
0.0425 (1.00) |
0.495 (1.00) |
0.143 (1.00) |
0.483 (1.00) |
0.135 (1.00) |
0.587 (1.00) |
0.439 (1.00) |
1 (1.00) |
| FAM19A1 | 8 (4%) | 217 |
0.156 (1.00) |
0.386 (1.00) |
0.458 (1.00) |
0.112 (1.00) |
0.485 (1.00) |
1 (1.00) |
0.904 (1.00) |
0.877 (1.00) |
| GK2 | 28 (12%) | 197 |
0.792 (1.00) |
0.603 (1.00) |
0.328 (1.00) |
0.187 (1.00) |
0.587 (1.00) |
0.581 (1.00) |
0.152 (1.00) |
0.803 (1.00) |
| LONRF2 | 16 (7%) | 209 |
0.898 (1.00) |
0.343 (1.00) |
0.945 (1.00) |
0.988 (1.00) |
0.948 (1.00) |
0.839 (1.00) |
0.486 (1.00) |
0.691 (1.00) |
| HAPLN1 | 11 (5%) | 214 |
1 (1.00) |
0.0959 (1.00) |
0.772 (1.00) |
0.882 (1.00) |
0.8 (1.00) |
1 (1.00) |
0.857 (1.00) |
0.608 (1.00) |
| NAP1L4 | 9 (4%) | 216 |
0.0205 (1.00) |
0.046 (1.00) |
0.0358 (1.00) |
0.6 (1.00) |
0.634 (1.00) |
0.0611 (1.00) |
0.626 (1.00) |
1 (1.00) |
| CLCC1 | 13 (6%) | 212 |
0.938 (1.00) |
0.361 (1.00) |
0.533 (1.00) |
0.144 (1.00) |
0.15 (1.00) |
0.388 (1.00) |
0.0193 (1.00) |
0.542 (1.00) |
| OR52J3 | 13 (6%) | 212 |
0.301 (1.00) |
0.278 (1.00) |
0.367 (1.00) |
0.464 (1.00) |
0.52 (1.00) |
0.604 (1.00) |
0.717 (1.00) |
0.277 (1.00) |
| TMCO5A | 8 (4%) | 217 |
0.00885 (1.00) |
0.47 (1.00) |
0.164 (1.00) |
0.0956 (1.00) |
0.0235 (1.00) |
0.291 (1.00) |
0.904 (1.00) |
1 (1.00) |
| C4ORF22 | 11 (5%) | 214 |
0.633 (1.00) |
0.925 (1.00) |
0.593 (1.00) |
0.281 (1.00) |
0.926 (1.00) |
0.852 (1.00) |
0.15 (1.00) |
0.273 (1.00) |
| MAP2K1 | 11 (5%) | 214 |
0.157 (1.00) |
0.0658 (1.00) |
0.0558 (1.00) |
0.124 (1.00) |
0.0316 (1.00) |
0.0475 (1.00) |
0.857 (1.00) |
0.437 (1.00) |
| OR7D2 | 17 (8%) | 208 |
0.535 (1.00) |
0.35 (1.00) |
0.778 (1.00) |
0.838 (1.00) |
0.0294 (1.00) |
0.847 (1.00) |
0.452 (1.00) |
0.533 (1.00) |
| CXCR2 | 11 (5%) | 214 |
0.86 (1.00) |
0.495 (1.00) |
0.866 (1.00) |
0.952 (1.00) |
0.465 (1.00) |
0.778 (1.00) |
0.79 (1.00) |
0.81 (1.00) |
| RBM22 | 7 (3%) | 218 |
0.442 (1.00) |
0.178 (1.00) |
0.344 (1.00) |
0.461 (1.00) |
1 (1.00) |
0.528 (1.00) |
||
| CX3CL1 | 4 (2%) | 221 |
0.143 (1.00) |
0.139 (1.00) |
1 (1.00) |
0.555 (1.00) |
0.124 (1.00) |
0.43 (1.00) |
||
| IGF2BP3 | 6 (3%) | 219 |
0.879 (1.00) |
1 (1.00) |
0.868 (1.00) |
0.646 (1.00) |
0.516 (1.00) |
1 (1.00) |
0.24 (1.00) |
0.589 (1.00) |
| KLRC3 | 10 (4%) | 215 |
1 (1.00) |
0.558 (1.00) |
0.517 (1.00) |
0.0113 (1.00) |
0.61 (1.00) |
0.101 (1.00) |
0.291 (1.00) |
1 (1.00) |
| LRRIQ4 | 19 (8%) | 206 |
0.2 (1.00) |
0.0957 (1.00) |
0.111 (1.00) |
0.357 (1.00) |
0.711 (1.00) |
0.729 (1.00) |
0.08 (1.00) |
0.00244 (1.00) |
| NR1H4 | 11 (5%) | 214 |
0.263 (1.00) |
0.338 (1.00) |
0.374 (1.00) |
0.362 (1.00) |
0.8 (1.00) |
0.778 (1.00) |
1 (1.00) |
0.437 (1.00) |
| OR4A15 | 21 (9%) | 204 |
0.437 (1.00) |
0.321 (1.00) |
1 (1.00) |
0.202 (1.00) |
0.0203 (1.00) |
0.339 (1.00) |
0.643 (1.00) |
0.419 (1.00) |
| MAGEA4 | 10 (4%) | 215 |
0.398 (1.00) |
0.558 (1.00) |
0.0143 (1.00) |
0.353 (1.00) |
0.144 (1.00) |
0.693 (1.00) |
0.42 (1.00) |
0.512 (1.00) |
| DGAT2L6 | 10 (4%) | 215 |
0.508 (1.00) |
0.712 (1.00) |
0.853 (1.00) |
0.11 (1.00) |
0.335 (1.00) |
0.693 (1.00) |
0.654 (1.00) |
0.395 (1.00) |
| RGS18 | 8 (4%) | 217 |
0.35 (1.00) |
0.899 (1.00) |
0.122 (1.00) |
0.403 (1.00) |
0.226 (1.00) |
0.724 (1.00) |
0.218 (1.00) |
0.771 (1.00) |
| TSGA13 | 9 (4%) | 216 |
0.53 (1.00) |
0.579 (1.00) |
0.118 (1.00) |
0.333 (1.00) |
0.58 (1.00) |
0.745 (1.00) |
0.762 (1.00) |
0.78 (1.00) |
| OPN5 | 7 (3%) | 218 |
0.208 (1.00) |
0.897 (1.00) |
1 (1.00) |
0.343 (1.00) |
0.395 (1.00) |
0.599 (1.00) |
1 (1.00) |
1 (1.00) |
| SPINK13 | 7 (3%) | 218 |
0.299 (1.00) |
0.233 (1.00) |
0.892 (1.00) |
0.457 (1.00) |
0.566 (1.00) |
0.683 (1.00) |
0.329 (1.00) |
0.739 (1.00) |
P value = 9.59e-08 (Fisher's exact test), Q value = 9.8e-05
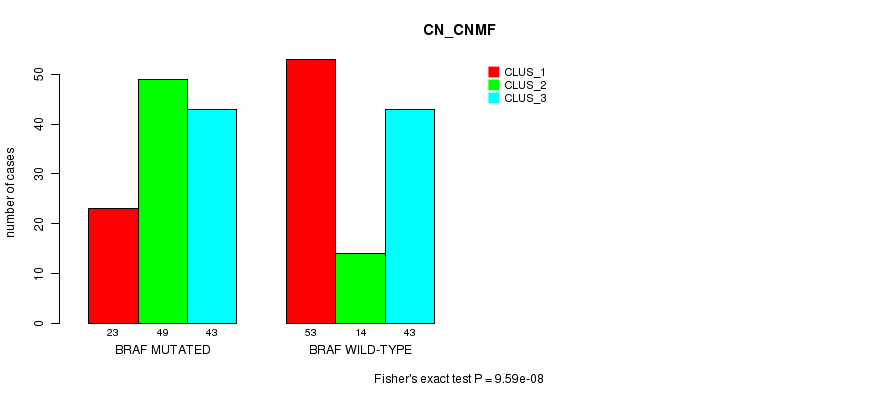
Table S1. Gene #2: 'BRAF MUTATION STATUS' versus Clinical Feature #1: 'CN_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 |
|---|---|---|---|
| ALL | 76 | 63 | 86 |
| BRAF MUTATED | 23 | 49 | 43 |
| BRAF WILD-TYPE | 53 | 14 | 43 |
Figure S1. Get High-res Image Gene #2: 'BRAF MUTATION STATUS' versus Clinical Feature #1: 'CN_CNMF'
P value = 1.94e-05 (Fisher's exact test), Q value = 0.02
Table S2. Gene #2: 'BRAF MUTATION STATUS' versus Clinical Feature #6: 'MRNASEQ_CHIERARCHICAL'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 |
|---|---|---|---|
| ALL | 55 | 55 | 112 |
| BRAF MUTATED | 14 | 37 | 63 |
| BRAF WILD-TYPE | 41 | 18 | 49 |
Figure S2. Get High-res Image Gene #2: 'BRAF MUTATION STATUS' versus Clinical Feature #6: 'MRNASEQ_CHIERARCHICAL'

-
Mutation data file = SKCM-TM.mutsig.cluster.txt
-
Molecular subtypes file = SKCM-TM.transferedmergedcluster.txt
-
Number of patients = 225
-
Number of significantly mutated genes = 129
-
Number of Molecular subtypes = 8
-
Exclude genes that fewer than K tumors have mutations, K = 3
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multi-class clinical features (nominal or ordinal), Chi-square tests (Greenwood and Nikulin 1996) were used to estimate the P values using the 'chisq.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
This is an experimental feature. The full results of the analysis summarized in this report can be downloaded from the TCGA Data Coordination Center.