This pipeline computes the correlation between significant copy number variation (cnv focal) genes and molecular subtypes.
Testing the association between copy number variation 62 focal events and 8 molecular subtypes across 56 patients, 2 significant findings detected with P value < 0.05 and Q value < 0.25.
-
amp_19q12 cnv correlated to 'METHLYATION_CNMF'.
-
del_13q14.2 cnv correlated to 'METHLYATION_CNMF'.
Table 1. Get Full Table Overview of the association between significant copy number variation of 62 focal events and 8 molecular subtypes. Shown in the table are P values (Q values). Thresholded by P value < 0.05 and Q value < 0.25, 2 significant findings detected.
|
Clinical Features |
CN CNMF |
METHLYATION CNMF |
MRNASEQ CNMF |
MRNASEQ CHIERARCHICAL |
MIRSEQ CNMF |
MIRSEQ CHIERARCHICAL |
MIRSEQ MATURE CNMF |
MIRSEQ MATURE CHIERARCHICAL |
||
| nCNV (%) | nWild-Type | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | |
| amp 19q12 | 45 (80%) | 11 |
0.00058 (0.287) |
8e-05 (0.0397) |
0.196 (1.00) |
0.0196 (1.00) |
0.645 (1.00) |
0.5 (1.00) |
0.44 (1.00) |
0.582 (1.00) |
| del 13q14 2 | 32 (57%) | 24 |
0.599 (1.00) |
0.00045 (0.223) |
0.0133 (1.00) |
0.0209 (1.00) |
0.479 (1.00) |
0.156 (1.00) |
0.14 (1.00) |
0.304 (1.00) |
| amp 1q22 | 42 (75%) | 14 |
0.922 (1.00) |
0.225 (1.00) |
0.476 (1.00) |
0.296 (1.00) |
0.183 (1.00) |
0.187 (1.00) |
0.241 (1.00) |
0.49 (1.00) |
| amp 2p14 | 28 (50%) | 28 |
0.499 (1.00) |
0.0262 (1.00) |
0.00696 (1.00) |
0.0799 (1.00) |
0.488 (1.00) |
0.247 (1.00) |
0.382 (1.00) |
0.0839 (1.00) |
| amp 2q13 | 29 (52%) | 27 |
0.386 (1.00) |
0.0162 (1.00) |
0.0465 (1.00) |
0.319 (1.00) |
1 (1.00) |
0.664 (1.00) |
0.841 (1.00) |
0.336 (1.00) |
| amp 3p25 1 | 20 (36%) | 36 |
0.0295 (1.00) |
0.512 (1.00) |
0.299 (1.00) |
0.113 (1.00) |
0.231 (1.00) |
0.0656 (1.00) |
0.465 (1.00) |
0.179 (1.00) |
| amp 3q26 2 | 37 (66%) | 19 |
0.00151 (0.741) |
0.00516 (1.00) |
0.09 (1.00) |
0.156 (1.00) |
0.349 (1.00) |
0.242 (1.00) |
0.731 (1.00) |
0.107 (1.00) |
| amp 4p16 3 | 25 (45%) | 31 |
0.03 (1.00) |
0.131 (1.00) |
0.386 (1.00) |
0.19 (1.00) |
0.976 (1.00) |
0.29 (1.00) |
0.877 (1.00) |
0.666 (1.00) |
| amp 5p13 2 | 26 (46%) | 30 |
0.0574 (1.00) |
0.572 (1.00) |
0.245 (1.00) |
0.0561 (1.00) |
0.929 (1.00) |
0.0456 (1.00) |
0.976 (1.00) |
0.141 (1.00) |
| amp 6p24 2 | 37 (66%) | 19 |
0.109 (1.00) |
0.971 (1.00) |
0.688 (1.00) |
0.36 (1.00) |
0.874 (1.00) |
0.5 (1.00) |
0.861 (1.00) |
0.576 (1.00) |
| amp 8p11 21 | 33 (59%) | 23 |
0.374 (1.00) |
0.627 (1.00) |
0.68 (1.00) |
0.557 (1.00) |
0.622 (1.00) |
1 (1.00) |
0.791 (1.00) |
0.955 (1.00) |
| amp 8q11 23 | 38 (68%) | 18 |
0.147 (1.00) |
0.226 (1.00) |
0.2 (1.00) |
0.164 (1.00) |
0.771 (1.00) |
0.922 (1.00) |
0.729 (1.00) |
0.958 (1.00) |
| amp 8q24 21 | 42 (75%) | 14 |
0.0438 (1.00) |
0.757 (1.00) |
0.189 (1.00) |
0.34 (1.00) |
0.0383 (1.00) |
0.0317 (1.00) |
0.0168 (1.00) |
0.0627 (1.00) |
| amp 8q24 21 | 40 (71%) | 16 |
0.00741 (1.00) |
0.357 (1.00) |
0.0802 (1.00) |
0.117 (1.00) |
0.0485 (1.00) |
0.0629 (1.00) |
0.018 (1.00) |
0.106 (1.00) |
| amp 10q22 2 | 26 (46%) | 30 |
0.246 (1.00) |
0.492 (1.00) |
0.965 (1.00) |
0.923 (1.00) |
0.726 (1.00) |
0.74 (1.00) |
0.599 (1.00) |
0.72 (1.00) |
| amp 11q13 1 | 19 (34%) | 37 |
0.461 (1.00) |
0.648 (1.00) |
0.327 (1.00) |
0.302 (1.00) |
0.76 (1.00) |
1 (1.00) |
0.67 (1.00) |
0.869 (1.00) |
| amp 12q12 | 22 (39%) | 34 |
0.162 (1.00) |
0.107 (1.00) |
0.253 (1.00) |
0.403 (1.00) |
0.928 (1.00) |
1 (1.00) |
0.689 (1.00) |
0.795 (1.00) |
| amp 12q15 | 17 (30%) | 39 |
0.469 (1.00) |
0.378 (1.00) |
0.252 (1.00) |
0.702 (1.00) |
0.799 (1.00) |
0.166 (1.00) |
0.556 (1.00) |
0.572 (1.00) |
| amp 13q31 3 | 25 (45%) | 31 |
0.407 (1.00) |
0.443 (1.00) |
0.139 (1.00) |
0.117 (1.00) |
0.0865 (1.00) |
0.0374 (1.00) |
0.0308 (1.00) |
0.0407 (1.00) |
| amp 16p11 2 | 20 (36%) | 36 |
0.00455 (1.00) |
0.28 (1.00) |
0.655 (1.00) |
0.0729 (1.00) |
0.76 (1.00) |
0.411 (1.00) |
0.402 (1.00) |
0.141 (1.00) |
| amp 17q12 | 27 (48%) | 29 |
0.939 (1.00) |
0.189 (1.00) |
0.334 (1.00) |
0.458 (1.00) |
0.343 (1.00) |
0.785 (1.00) |
0.639 (1.00) |
1 (1.00) |
| amp 17q25 1 | 32 (57%) | 24 |
0.64 (1.00) |
0.0122 (1.00) |
0.632 (1.00) |
0.307 (1.00) |
0.687 (1.00) |
0.721 (1.00) |
0.504 (1.00) |
0.756 (1.00) |
| amp 19p13 2 | 19 (34%) | 37 |
0.394 (1.00) |
0.271 (1.00) |
0.797 (1.00) |
0.323 (1.00) |
0.346 (1.00) |
0.275 (1.00) |
0.215 (1.00) |
0.665 (1.00) |
| amp 20q11 21 | 48 (86%) | 8 |
0.0141 (1.00) |
0.167 (1.00) |
0.544 (1.00) |
0.423 (1.00) |
0.161 (1.00) |
0.144 (1.00) |
0.128 (1.00) |
0.192 (1.00) |
| amp 20q11 21 | 48 (86%) | 8 |
0.155 (1.00) |
0.255 (1.00) |
0.193 (1.00) |
0.316 (1.00) |
0.178 (1.00) |
0.352 (1.00) |
0.147 (1.00) |
0.572 (1.00) |
| amp xp11 21 | 28 (50%) | 28 |
0.268 (1.00) |
0.566 (1.00) |
0.0488 (1.00) |
0.3 (1.00) |
0.0285 (1.00) |
0.0997 (1.00) |
0.0813 (1.00) |
0.088 (1.00) |
| del 1p36 21 | 19 (34%) | 37 |
0.083 (1.00) |
0.378 (1.00) |
0.348 (1.00) |
0.0816 (1.00) |
0.135 (1.00) |
0.0243 (1.00) |
0.121 (1.00) |
0.102 (1.00) |
| del 2q22 1 | 11 (20%) | 45 |
0.502 (1.00) |
0.545 (1.00) |
0.526 (1.00) |
0.171 (1.00) |
0.551 (1.00) |
0.555 (1.00) |
0.284 (1.00) |
0.162 (1.00) |
| del 3p21 1 | 37 (66%) | 19 |
0.428 (1.00) |
0.46 (1.00) |
0.466 (1.00) |
0.433 (1.00) |
0.519 (1.00) |
0.768 (1.00) |
0.49 (1.00) |
0.255 (1.00) |
| del 3p14 1 | 30 (54%) | 26 |
1 (1.00) |
0.651 (1.00) |
0.172 (1.00) |
0.619 (1.00) |
0.0874 (1.00) |
0.537 (1.00) |
0.205 (1.00) |
0.133 (1.00) |
| del 3q13 31 | 19 (34%) | 37 |
0.117 (1.00) |
0.0989 (1.00) |
0.923 (1.00) |
0.159 (1.00) |
0.972 (1.00) |
0.655 (1.00) |
0.707 (1.00) |
0.617 (1.00) |
| del 4q22 1 | 37 (66%) | 19 |
0.0643 (1.00) |
0.0555 (1.00) |
0.0245 (1.00) |
0.154 (1.00) |
0.0124 (1.00) |
0.125 (1.00) |
0.0245 (1.00) |
0.14 (1.00) |
| del 4q34 3 | 37 (66%) | 19 |
0.00425 (1.00) |
0.0198 (1.00) |
0.011 (1.00) |
0.0404 (1.00) |
0.00298 (1.00) |
0.201 (1.00) |
0.00818 (1.00) |
0.101 (1.00) |
| del 5q11 2 | 26 (46%) | 30 |
0.153 (1.00) |
0.0922 (1.00) |
0.585 (1.00) |
0.45 (1.00) |
0.549 (1.00) |
0.295 (1.00) |
0.0779 (1.00) |
0.12 (1.00) |
| del 6q26 | 10 (18%) | 46 |
0.528 (1.00) |
0.543 (1.00) |
0.121 (1.00) |
0.701 (1.00) |
0.33 (1.00) |
0.3 (1.00) |
0.174 (1.00) |
0.719 (1.00) |
| del 7q11 22 | 12 (21%) | 44 |
0.265 (1.00) |
0.509 (1.00) |
0.761 (1.00) |
0.834 (1.00) |
0.861 (1.00) |
0.533 (1.00) |
0.808 (1.00) |
0.876 (1.00) |
| del 7q36 2 | 20 (36%) | 36 |
0.169 (1.00) |
0.169 (1.00) |
0.494 (1.00) |
0.273 (1.00) |
0.401 (1.00) |
0.0458 (1.00) |
0.548 (1.00) |
0.265 (1.00) |
| del 8p21 3 | 32 (57%) | 24 |
0.00064 (0.316) |
0.45 (1.00) |
0.267 (1.00) |
0.327 (1.00) |
0.812 (1.00) |
0.287 (1.00) |
0.443 (1.00) |
0.707 (1.00) |
| del 9p23 | 34 (61%) | 22 |
0.0948 (1.00) |
0.122 (1.00) |
0.636 (1.00) |
0.325 (1.00) |
0.503 (1.00) |
0.425 (1.00) |
0.889 (1.00) |
0.25 (1.00) |
| del 9q21 13 | 39 (70%) | 17 |
0.108 (1.00) |
0.324 (1.00) |
0.212 (1.00) |
0.254 (1.00) |
0.479 (1.00) |
0.32 (1.00) |
0.678 (1.00) |
0.653 (1.00) |
| del 9q33 3 | 38 (68%) | 18 |
0.448 (1.00) |
0.0663 (1.00) |
0.087 (1.00) |
0.169 (1.00) |
1 (1.00) |
0.103 (1.00) |
0.856 (1.00) |
0.447 (1.00) |
| del 10q21 1 | 18 (32%) | 38 |
0.561 (1.00) |
0.397 (1.00) |
0.603 (1.00) |
0.318 (1.00) |
0.47 (1.00) |
0.293 (1.00) |
0.312 (1.00) |
0.554 (1.00) |
| del 10q23 31 | 20 (36%) | 36 |
0.142 (1.00) |
0.103 (1.00) |
0.123 (1.00) |
0.246 (1.00) |
0.76 (1.00) |
0.853 (1.00) |
0.591 (1.00) |
0.906 (1.00) |
| del 11p15 5 | 25 (45%) | 31 |
0.19 (1.00) |
0.766 (1.00) |
0.899 (1.00) |
0.895 (1.00) |
0.726 (1.00) |
0.897 (1.00) |
0.239 (1.00) |
0.831 (1.00) |
| del 11q14 1 | 26 (46%) | 30 |
0.648 (1.00) |
0.216 (1.00) |
1 (1.00) |
0.922 (1.00) |
0.268 (1.00) |
0.525 (1.00) |
0.261 (1.00) |
0.293 (1.00) |
| del 11q24 2 | 27 (48%) | 29 |
0.565 (1.00) |
0.0315 (1.00) |
0.139 (1.00) |
0.784 (1.00) |
0.248 (1.00) |
0.621 (1.00) |
0.253 (1.00) |
0.281 (1.00) |
| del 12q23 1 | 20 (36%) | 36 |
0.661 (1.00) |
0.898 (1.00) |
0.655 (1.00) |
0.75 (1.00) |
0.95 (1.00) |
0.34 (1.00) |
0.929 (1.00) |
0.65 (1.00) |
| del 12q24 31 | 20 (36%) | 36 |
0.813 (1.00) |
0.825 (1.00) |
0.155 (1.00) |
0.259 (1.00) |
0.562 (1.00) |
0.887 (1.00) |
0.15 (1.00) |
0.914 (1.00) |
| del 13q12 11 | 30 (54%) | 26 |
0.468 (1.00) |
0.0198 (1.00) |
0.373 (1.00) |
0.208 (1.00) |
0.723 (1.00) |
0.0256 (1.00) |
0.487 (1.00) |
0.0876 (1.00) |
| del 14q21 1 | 28 (50%) | 28 |
0.732 (1.00) |
0.0499 (1.00) |
0.0632 (1.00) |
0.0561 (1.00) |
0.38 (1.00) |
0.379 (1.00) |
0.355 (1.00) |
0.663 (1.00) |
| del 15q15 1 | 43 (77%) | 13 |
0.0399 (1.00) |
0.0312 (1.00) |
0.348 (1.00) |
0.768 (1.00) |
0.117 (1.00) |
0.345 (1.00) |
0.0493 (1.00) |
0.243 (1.00) |
| del 16p13 3 | 33 (59%) | 23 |
0.163 (1.00) |
0.551 (1.00) |
0.569 (1.00) |
0.228 (1.00) |
0.347 (1.00) |
0.39 (1.00) |
0.604 (1.00) |
0.173 (1.00) |
| del 16q23 1 | 39 (70%) | 17 |
0.161 (1.00) |
0.422 (1.00) |
0.398 (1.00) |
0.393 (1.00) |
0.867 (1.00) |
0.812 (1.00) |
0.479 (1.00) |
0.98 (1.00) |
| del 17p13 1 | 42 (75%) | 14 |
0.0678 (1.00) |
0.0554 (1.00) |
0.0581 (1.00) |
0.425 (1.00) |
0.132 (1.00) |
0.182 (1.00) |
0.0625 (1.00) |
0.123 (1.00) |
| del 17q21 32 | 19 (34%) | 37 |
0.87 (1.00) |
0.763 (1.00) |
0.175 (1.00) |
0.0663 (1.00) |
0.223 (1.00) |
0.209 (1.00) |
0.186 (1.00) |
0.297 (1.00) |
| del 18q22 2 | 26 (46%) | 30 |
0.134 (1.00) |
0.342 (1.00) |
0.505 (1.00) |
0.362 (1.00) |
0.0224 (1.00) |
0.0795 (1.00) |
0.0896 (1.00) |
0.231 (1.00) |
| del 19p13 3 | 47 (84%) | 9 |
0.142 (1.00) |
0.02 (1.00) |
0.0981 (1.00) |
0.38 (1.00) |
0.0148 (1.00) |
0.0849 (1.00) |
0.00856 (1.00) |
0.16 (1.00) |
| del 19q13 33 | 17 (30%) | 39 |
0.0381 (1.00) |
0.00896 (1.00) |
0.00064 (0.316) |
0.0358 (1.00) |
0.222 (1.00) |
0.0379 (1.00) |
0.558 (1.00) |
0.0162 (1.00) |
| del 20p12 1 | 9 (16%) | 47 |
1 (1.00) |
0.699 (1.00) |
0.389 (1.00) |
0.53 (1.00) |
0.221 (1.00) |
0.339 (1.00) |
0.318 (1.00) |
0.595 (1.00) |
| del 22q13 31 | 40 (71%) | 16 |
0.584 (1.00) |
0.408 (1.00) |
0.892 (1.00) |
0.456 (1.00) |
0.576 (1.00) |
0.351 (1.00) |
0.485 (1.00) |
0.727 (1.00) |
| del xp21 1 | 18 (32%) | 38 |
0.6 (1.00) |
0.421 (1.00) |
0.821 (1.00) |
0.87 (1.00) |
0.361 (1.00) |
1 (1.00) |
0.781 (1.00) |
0.656 (1.00) |
| del xq25 | 23 (41%) | 33 |
0.554 (1.00) |
0.311 (1.00) |
0.112 (1.00) |
0.899 (1.00) |
0.731 (1.00) |
0.566 (1.00) |
0.566 (1.00) |
0.506 (1.00) |
P value = 8e-05 (Fisher's exact test), Q value = 0.04
Table S1. Gene #22: 'amp_19q12' versus Molecular Subtype #2: 'METHLYATION_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 | CLUS_5 |
|---|---|---|---|---|---|
| ALL | 11 | 9 | 10 | 13 | 13 |
| AMP PEAK 22(19Q12) MUTATED | 11 | 7 | 3 | 11 | 13 |
| AMP PEAK 22(19Q12) WILD-TYPE | 0 | 2 | 7 | 2 | 0 |
Figure S1. Get High-res Image Gene #22: 'amp_19q12' versus Molecular Subtype #2: 'METHLYATION_CNMF'
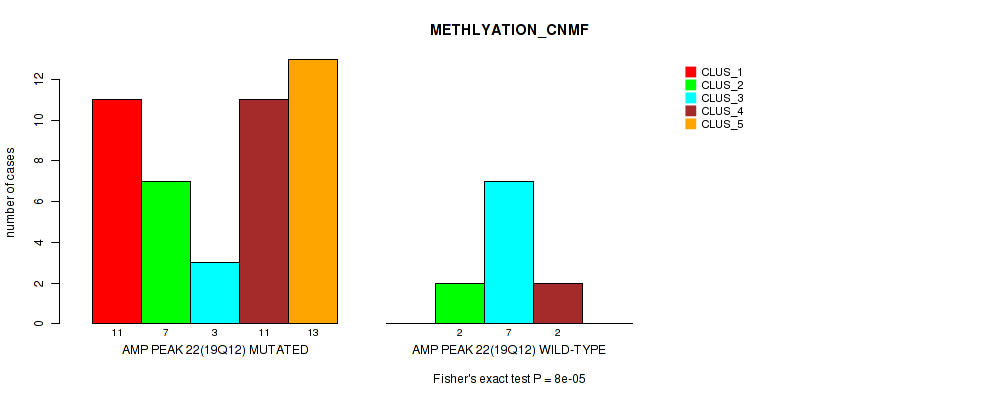
P value = 0.00045 (Fisher's exact test), Q value = 0.22
Table S2. Gene #49: 'del_13q14.2' versus Molecular Subtype #2: 'METHLYATION_CNMF'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 | CLUS_5 |
|---|---|---|---|---|---|
| ALL | 11 | 9 | 10 | 13 | 13 |
| DEL PEAK 24(13Q14.2) MUTATED | 3 | 7 | 7 | 12 | 3 |
| DEL PEAK 24(13Q14.2) WILD-TYPE | 8 | 2 | 3 | 1 | 10 |
Figure S2. Get High-res Image Gene #49: 'del_13q14.2' versus Molecular Subtype #2: 'METHLYATION_CNMF'
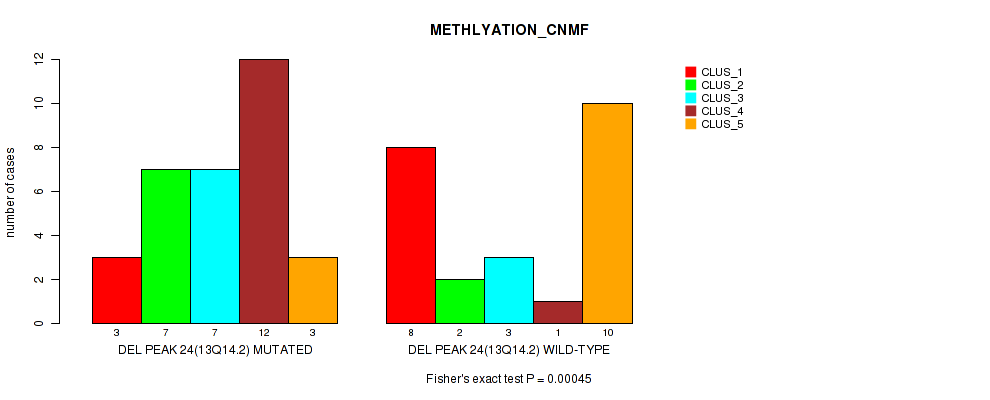
-
Copy number data file = transformed.cor.cli.txt
-
Molecular subtype file = UCS-TP.transferedmergedcluster.txt
-
Number of patients = 56
-
Number of significantly focal cnvs = 62
-
Number of molecular subtypes = 8
-
Exclude genes that fewer than K tumors have alterations, K = 3
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
In addition to the links below, the full results of the analysis summarized in this report can also be downloaded programmatically using firehose_get, or interactively from either the Broad GDAC website or TCGA Data Coordination Center Portal.