This pipeline computes the correlation between significantly recurrent gene mutations and selected clinical features.
Testing the association between mutation status of 119 genes and 7 clinical features across 62 patients, one significant finding detected with Q value < 0.25.
-
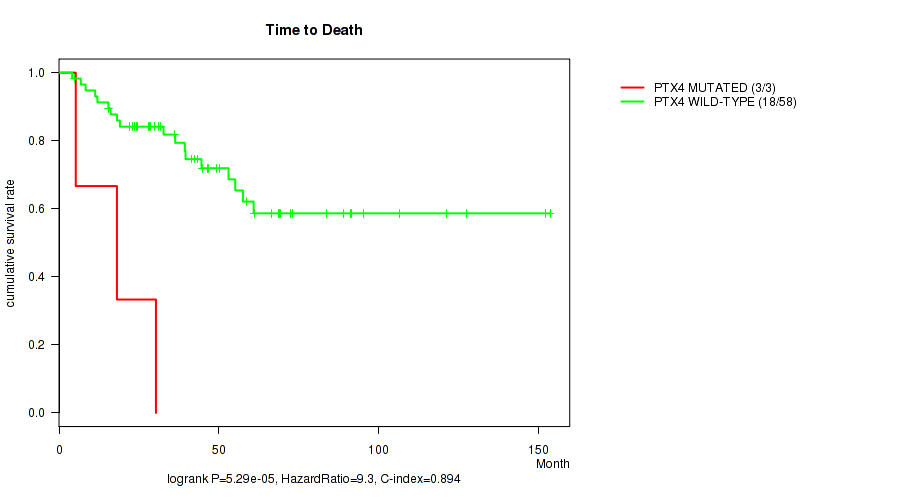
PTX4 mutation correlated to 'Time to Death'.
Table 1. Get Full Table Overview of the association between mutation status of 119 genes and 7 clinical features. Shown in the table are P values (Q values). Thresholded by Q value < 0.25, one significant finding detected.
|
Clinical Features |
Time to Death |
AGE |
NEOPLASM DISEASESTAGE |
PATHOLOGY T STAGE |
PATHOLOGY N STAGE |
GENDER | ETHNICITY | ||
| nMutated (%) | nWild-Type | logrank test | Wilcoxon-test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | |
| PTX4 | 3 (5%) | 59 |
5.29e-05 (0.0436) |
0.301 (1.00) |
0.0109 (1.00) |
0.0354 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| ZFPM1 | 24 (39%) | 38 |
0.347 (1.00) |
0.534 (1.00) |
0.311 (1.00) |
0.975 (1.00) |
0.669 (1.00) |
1 (1.00) |
0.677 (1.00) |
| LACTB | 19 (31%) | 43 |
0.722 (1.00) |
0.945 (1.00) |
0.201 (1.00) |
0.0921 (1.00) |
1 (1.00) |
0.568 (1.00) |
0.656 (1.00) |
| CCDC102A | 17 (27%) | 45 |
0.204 (1.00) |
0.477 (1.00) |
0.154 (1.00) |
0.207 (1.00) |
0.168 (1.00) |
1 (1.00) |
1 (1.00) |
| ZNF517 | 13 (21%) | 49 |
0.745 (1.00) |
0.0421 (1.00) |
0.737 (1.00) |
0.753 (1.00) |
1 (1.00) |
0.756 (1.00) |
0.306 (1.00) |
| MAL2 | 11 (18%) | 51 |
0.933 (1.00) |
0.0708 (1.00) |
0.395 (1.00) |
0.0426 (1.00) |
0.31 (1.00) |
0.0789 (1.00) |
0.3 (1.00) |
| CLDN23 | 10 (16%) | 52 |
0.867 (1.00) |
0.716 (1.00) |
0.625 (1.00) |
0.785 (1.00) |
0.266 (1.00) |
0.472 (1.00) |
0.559 (1.00) |
| TOR3A | 12 (19%) | 50 |
0.607 (1.00) |
0.533 (1.00) |
0.191 (1.00) |
0.138 (1.00) |
0.582 (1.00) |
0.74 (1.00) |
1 (1.00) |
| USP42 | 15 (24%) | 47 |
0.52 (1.00) |
0.681 (1.00) |
0.588 (1.00) |
0.821 (1.00) |
0.638 (1.00) |
0.541 (1.00) |
1 (1.00) |
| TP53 | 12 (19%) | 50 |
0.0232 (1.00) |
0.782 (1.00) |
0.106 (1.00) |
0.317 (1.00) |
1 (1.00) |
0.74 (1.00) |
0.58 (1.00) |
| APOE | 7 (11%) | 55 |
0.909 (1.00) |
0.133 (1.00) |
0.927 (1.00) |
0.923 (1.00) |
1 (1.00) |
0.0861 (1.00) |
1 (1.00) |
| ZAR1 | 11 (18%) | 51 |
0.509 (1.00) |
0.672 (1.00) |
0.619 (1.00) |
0.337 (1.00) |
0.31 (1.00) |
0.0789 (1.00) |
0.559 (1.00) |
| ASPDH | 8 (13%) | 54 |
0.264 (1.00) |
0.983 (1.00) |
0.00348 (1.00) |
0.346 (1.00) |
0.108 (1.00) |
0.7 (1.00) |
1 (1.00) |
| KCNK17 | 9 (15%) | 53 |
0.293 (1.00) |
0.719 (1.00) |
0.898 (1.00) |
0.435 (1.00) |
0.583 (1.00) |
0.709 (1.00) |
0.18 (1.00) |
| LZTR1 | 6 (10%) | 56 |
0.456 (1.00) |
0.46 (1.00) |
1 (1.00) |
1 (1.00) |
0.49 (1.00) |
1 (1.00) |
1 (1.00) |
| ERCC2 | 10 (16%) | 52 |
0.729 (1.00) |
0.833 (1.00) |
0.607 (1.00) |
0.613 (1.00) |
0.266 (1.00) |
1 (1.00) |
0.511 (1.00) |
| RINL | 8 (13%) | 54 |
0.324 (1.00) |
0.622 (1.00) |
0.622 (1.00) |
0.523 (1.00) |
1 (1.00) |
0.7 (1.00) |
0.268 (1.00) |
| SYT8 | 8 (13%) | 54 |
0.19 (1.00) |
0.407 (1.00) |
0.202 (1.00) |
0.695 (1.00) |
0.583 (1.00) |
1 (1.00) |
0.559 (1.00) |
| LRIG1 | 16 (26%) | 46 |
0.579 (1.00) |
0.459 (1.00) |
0.942 (1.00) |
0.725 (1.00) |
0.165 (1.00) |
0.375 (1.00) |
0.644 (1.00) |
| C1ORF106 | 9 (15%) | 53 |
0.166 (1.00) |
0.384 (1.00) |
0.48 (1.00) |
0.282 (1.00) |
0.224 (1.00) |
0.14 (1.00) |
0.559 (1.00) |
| GPRIN2 | 8 (13%) | 54 |
0.927 (1.00) |
0.401 (1.00) |
0.899 (1.00) |
0.555 (1.00) |
0.583 (1.00) |
1 (1.00) |
1 (1.00) |
| CTNNB1 | 7 (11%) | 55 |
0.237 (1.00) |
0.266 (1.00) |
0.731 (1.00) |
0.934 (1.00) |
0.144 (1.00) |
0.0443 (1.00) |
0.559 (1.00) |
| C16ORF3 | 5 (8%) | 57 |
0.449 (1.00) |
0.756 (1.00) |
0.269 (1.00) |
0.565 (1.00) |
0.427 (1.00) |
0.151 (1.00) |
1 (1.00) |
| CCDC105 | 6 (10%) | 56 |
0.416 (1.00) |
0.432 (1.00) |
0.29 (1.00) |
0.109 (1.00) |
0.49 (1.00) |
0.0808 (1.00) |
0.511 (1.00) |
| RGS9BP | 8 (13%) | 54 |
0.365 (1.00) |
0.983 (1.00) |
0.941 (1.00) |
0.0381 (1.00) |
0.548 (1.00) |
0.438 (1.00) |
0.511 (1.00) |
| HHIPL1 | 6 (10%) | 56 |
0.731 (1.00) |
0.6 (1.00) |
0.735 (1.00) |
0.713 (1.00) |
0.49 (1.00) |
0.657 (1.00) |
1 (1.00) |
| C10ORF95 | 6 (10%) | 56 |
0.97 (1.00) |
0.536 (1.00) |
0.524 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| TSC22D2 | 8 (13%) | 54 |
0.108 (1.00) |
0.629 (1.00) |
0.317 (1.00) |
0.13 (1.00) |
1 (1.00) |
0.7 (1.00) |
0.511 (1.00) |
| NOXA1 | 5 (8%) | 57 |
0.715 (1.00) |
0.99 (1.00) |
0.901 (1.00) |
0.802 (1.00) |
0.427 (1.00) |
0.151 (1.00) |
1 (1.00) |
| THEM4 | 5 (8%) | 57 |
0.388 (1.00) |
0.313 (1.00) |
0.43 (1.00) |
0.394 (1.00) |
0.357 (1.00) |
0.647 (1.00) |
1 (1.00) |
| ATXN1 | 10 (16%) | 52 |
0.847 (1.00) |
0.263 (1.00) |
0.78 (1.00) |
0.429 (1.00) |
1 (1.00) |
0.082 (1.00) |
1 (1.00) |
| PLIN5 | 5 (8%) | 57 |
0.497 (1.00) |
0.587 (1.00) |
1 (1.00) |
0.899 (1.00) |
0.427 (1.00) |
0.647 (1.00) |
0.374 (1.00) |
| OPRD1 | 12 (19%) | 50 |
0.814 (1.00) |
0.139 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.512 (1.00) |
1 (1.00) |
| KRTAP4-5 | 5 (8%) | 57 |
0.146 (1.00) |
0.477 (1.00) |
0.579 (1.00) |
0.362 (1.00) |
1 (1.00) |
0.647 (1.00) |
1 (1.00) |
| C19ORF10 | 7 (11%) | 55 |
0.989 (1.00) |
0.533 (1.00) |
0.144 (1.00) |
0.453 (1.00) |
0.548 (1.00) |
0.405 (1.00) |
1 (1.00) |
| AATK | 6 (10%) | 56 |
0.343 (1.00) |
0.034 (1.00) |
0.646 (1.00) |
0.646 (1.00) |
0.49 (1.00) |
0.409 (1.00) |
1 (1.00) |
| SARM1 | 6 (10%) | 56 |
0.203 (1.00) |
0.739 (1.00) |
0.694 (1.00) |
0.241 (1.00) |
1 (1.00) |
0.657 (1.00) |
0.101 (1.00) |
| TRIOBP | 10 (16%) | 52 |
0.831 (1.00) |
0.767 (1.00) |
0.953 (1.00) |
1 (1.00) |
0.0548 (1.00) |
0.082 (1.00) |
1 (1.00) |
| ZNF598 | 12 (19%) | 50 |
0.634 (1.00) |
0.021 (1.00) |
0.516 (1.00) |
0.802 (1.00) |
0.582 (1.00) |
0.0186 (1.00) |
0.58 (1.00) |
| WDR34 | 5 (8%) | 57 |
0.601 (1.00) |
0.679 (1.00) |
1 (1.00) |
0.741 (1.00) |
1 (1.00) |
0.0493 (1.00) |
1 (1.00) |
| ZNF628 | 7 (11%) | 55 |
0.108 (1.00) |
0.755 (1.00) |
0.463 (1.00) |
0.422 (1.00) |
1 (1.00) |
1 (1.00) |
0.511 (1.00) |
| BTBD11 | 6 (10%) | 56 |
0.662 (1.00) |
0.868 (1.00) |
0.0402 (1.00) |
0.149 (1.00) |
0.49 (1.00) |
0.409 (1.00) |
1 (1.00) |
| IRX3 | 5 (8%) | 57 |
0.924 (1.00) |
0.313 (1.00) |
0.244 (1.00) |
0.508 (1.00) |
1 (1.00) |
0.647 (1.00) |
1 (1.00) |
| FPGS | 5 (8%) | 57 |
0.404 (1.00) |
0.319 (1.00) |
0.268 (1.00) |
0.565 (1.00) |
0.427 (1.00) |
1 (1.00) |
1 (1.00) |
| MEN1 | 5 (8%) | 57 |
0.0634 (1.00) |
0.688 (1.00) |
0.0362 (1.00) |
0.159 (1.00) |
0.357 (1.00) |
1 (1.00) |
0.511 (1.00) |
| BHLHE22 | 5 (8%) | 57 |
0.179 (1.00) |
0.928 (1.00) |
0.325 (1.00) |
0.898 (1.00) |
0.0759 (1.00) |
0.647 (1.00) |
0.18 (1.00) |
| DMKN | 3 (5%) | 59 |
0.201 (1.00) |
0.384 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
||
| OBSCN | 18 (29%) | 44 |
0.65 (1.00) |
0.0453 (1.00) |
0.506 (1.00) |
0.861 (1.00) |
0.341 (1.00) |
0.561 (1.00) |
1 (1.00) |
| PANK2 | 5 (8%) | 57 |
0.0662 (1.00) |
0.166 (1.00) |
0.9 (1.00) |
0.632 (1.00) |
1 (1.00) |
0.0493 (1.00) |
1 (1.00) |
| RREB1 | 5 (8%) | 57 |
0.805 (1.00) |
0.938 (1.00) |
0.693 (1.00) |
0.242 (1.00) |
0.0759 (1.00) |
1 (1.00) |
0.374 (1.00) |
| RNF149 | 4 (6%) | 58 |
0.193 (1.00) |
0.0709 (1.00) |
0.236 (1.00) |
0.396 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| GLTPD2 | 6 (10%) | 56 |
0.361 (1.00) |
0.1 (1.00) |
0.693 (1.00) |
0.63 (1.00) |
1 (1.00) |
0.657 (1.00) |
0.206 (1.00) |
| SNED1 | 5 (8%) | 57 |
0.433 (1.00) |
0.737 (1.00) |
0.137 (1.00) |
0.187 (1.00) |
0.0759 (1.00) |
0.647 (1.00) |
1 (1.00) |
| TPO | 12 (19%) | 50 |
0.621 (1.00) |
0.852 (1.00) |
0.866 (1.00) |
0.917 (1.00) |
0.0919 (1.00) |
1 (1.00) |
1 (1.00) |
| PTPLA | 4 (6%) | 58 |
0.689 (1.00) |
0.72 (1.00) |
0.617 (1.00) |
0.642 (1.00) |
1 (1.00) |
0.61 (1.00) |
0.511 (1.00) |
| PRSS27 | 3 (5%) | 59 |
0.0881 (1.00) |
0.412 (1.00) |
0.476 (1.00) |
0.528 (1.00) |
1 (1.00) |
0.285 (1.00) |
0.0374 (1.00) |
| CD320 | 4 (6%) | 58 |
0.291 (1.00) |
0.282 (1.00) |
0.161 (1.00) |
0.286 (1.00) |
1 (1.00) |
1 (1.00) |
0.101 (1.00) |
| SEMA5B | 8 (13%) | 54 |
0.803 (1.00) |
0.644 (1.00) |
0.898 (1.00) |
1 (1.00) |
1 (1.00) |
0.438 (1.00) |
1 (1.00) |
| CCDC150 | 4 (6%) | 58 |
0.0759 (1.00) |
0.456 (1.00) |
0.0366 (1.00) |
0.109 (1.00) |
0.357 (1.00) |
1 (1.00) |
0.101 (1.00) |
| TAF5 | 4 (6%) | 58 |
0.346 (1.00) |
0.596 (1.00) |
0.327 (1.00) |
0.848 (1.00) |
0.357 (1.00) |
1 (1.00) |
0.374 (1.00) |
| GARS | 19 (31%) | 43 |
0.00873 (1.00) |
0.225 (1.00) |
0.00656 (1.00) |
0.00598 (1.00) |
0.656 (1.00) |
1 (1.00) |
0.644 (1.00) |
| MUC5B | 22 (35%) | 40 |
0.866 (1.00) |
0.581 (1.00) |
0.776 (1.00) |
0.876 (1.00) |
0.653 (1.00) |
1 (1.00) |
1 (1.00) |
| KIAA1984 | 4 (6%) | 58 |
0.898 (1.00) |
0.657 (1.00) |
0.834 (1.00) |
0.526 (1.00) |
1 (1.00) |
0.124 (1.00) |
0.511 (1.00) |
| SRPX | 3 (5%) | 59 |
0.953 (1.00) |
0.922 (1.00) |
0.837 (1.00) |
0.637 (1.00) |
0.279 (1.00) |
0.546 (1.00) |
0.511 (1.00) |
| TNIP2 | 5 (8%) | 57 |
0.173 (1.00) |
0.605 (1.00) |
0.117 (1.00) |
0.641 (1.00) |
0.0478 (1.00) |
1 (1.00) |
1 (1.00) |
| IDUA | 8 (13%) | 54 |
0.157 (1.00) |
0.204 (1.00) |
0.1 (1.00) |
0.223 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| RNF39 | 5 (8%) | 57 |
0.632 (1.00) |
0.776 (1.00) |
0.371 (1.00) |
0.847 (1.00) |
0.357 (1.00) |
1 (1.00) |
0.374 (1.00) |
| PABPC1 | 4 (6%) | 58 |
0.666 (1.00) |
0.398 (1.00) |
1 (1.00) |
1 (1.00) |
0.357 (1.00) |
0.61 (1.00) |
0.559 (1.00) |
| SCRT1 | 3 (5%) | 59 |
0.0287 (1.00) |
0.225 (1.00) |
0.837 (1.00) |
0.337 (1.00) |
0.279 (1.00) |
0.546 (1.00) |
1 (1.00) |
| MAP1S | 5 (8%) | 57 |
0.647 (1.00) |
0.401 (1.00) |
0.395 (1.00) |
1 (1.00) |
0.427 (1.00) |
0.337 (1.00) |
1 (1.00) |
| NF1 | 7 (11%) | 55 |
0.213 (1.00) |
0.681 (1.00) |
0.928 (1.00) |
1 (1.00) |
1 (1.00) |
0.00642 (1.00) |
0.511 (1.00) |
| AKAP2 | 4 (6%) | 58 |
0.707 (1.00) |
0.586 (1.00) |
0.326 (1.00) |
0.741 (1.00) |
1 (1.00) |
0.124 (1.00) |
|
| NOTCH2 | 5 (8%) | 57 |
0.489 (1.00) |
0.766 (1.00) |
0.693 (1.00) |
0.47 (1.00) |
0.357 (1.00) |
1 (1.00) |
1 (1.00) |
| FANK1 | 4 (6%) | 58 |
0.718 (1.00) |
0.218 (1.00) |
0.066 (1.00) |
0.09 (1.00) |
0.357 (1.00) |
0.287 (1.00) |
0.559 (1.00) |
| NEFH | 6 (10%) | 56 |
0.151 (1.00) |
0.536 (1.00) |
1 (1.00) |
0.411 (1.00) |
0.49 (1.00) |
1 (1.00) |
0.18 (1.00) |
| NPTX1 | 3 (5%) | 59 |
0.997 (1.00) |
0.896 (1.00) |
0.256 (1.00) |
0.249 (1.00) |
1 (1.00) |
0.285 (1.00) |
1 (1.00) |
| KBTBD13 | 9 (15%) | 53 |
0.477 (1.00) |
0.497 (1.00) |
0.0962 (1.00) |
0.122 (1.00) |
0.224 (1.00) |
0.14 (1.00) |
1 (1.00) |
| RASIP1 | 6 (10%) | 56 |
0.0484 (1.00) |
0.99 (1.00) |
0.68 (1.00) |
0.485 (1.00) |
0.49 (1.00) |
0.409 (1.00) |
1 (1.00) |
| AR | 4 (6%) | 58 |
0.125 (1.00) |
0.785 (1.00) |
0.373 (1.00) |
0.159 (1.00) |
0.357 (1.00) |
1 (1.00) |
1 (1.00) |
| KNDC1 | 9 (15%) | 53 |
0.214 (1.00) |
0.912 (1.00) |
0.11 (1.00) |
0.0627 (1.00) |
1 (1.00) |
0.14 (1.00) |
1 (1.00) |
| SEZ6L2 | 8 (13%) | 54 |
0.64 (1.00) |
0.883 (1.00) |
0.899 (1.00) |
0.661 (1.00) |
1 (1.00) |
1 (1.00) |
0.306 (1.00) |
| VARS | 6 (10%) | 56 |
0.991 (1.00) |
0.84 (1.00) |
0.298 (1.00) |
0.241 (1.00) |
1 (1.00) |
0.174 (1.00) |
0.0211 (1.00) |
| IER5 | 3 (5%) | 59 |
0.588 (1.00) |
0.0919 (1.00) |
0.0346 (1.00) |
0.279 (1.00) |
0.546 (1.00) |
1 (1.00) |
|
| NMU | 5 (8%) | 57 |
0.247 (1.00) |
0.272 (1.00) |
1 (1.00) |
0.899 (1.00) |
0.427 (1.00) |
0.151 (1.00) |
0.511 (1.00) |
| TMEM189 | 3 (5%) | 59 |
0.441 (1.00) |
1 (1.00) |
1 (1.00) |
0.374 (1.00) |
|||
| ADAD2 | 4 (6%) | 58 |
0.971 (1.00) |
0.626 (1.00) |
0.703 (1.00) |
1 (1.00) |
1 (1.00) |
0.124 (1.00) |
1 (1.00) |
| PLEC | 13 (21%) | 49 |
0.451 (1.00) |
0.568 (1.00) |
0.847 (1.00) |
0.825 (1.00) |
1 (1.00) |
0.112 (1.00) |
0.644 (1.00) |
| RGMB | 6 (10%) | 56 |
0.371 (1.00) |
0.73 (1.00) |
0.928 (1.00) |
0.647 (1.00) |
0.49 (1.00) |
1 (1.00) |
0.306 (1.00) |
| CLIC6 | 5 (8%) | 57 |
0.343 (1.00) |
0.345 (1.00) |
0.476 (1.00) |
0.712 (1.00) |
1 (1.00) |
0.647 (1.00) |
1 (1.00) |
| ZNF814 | 3 (5%) | 59 |
0.743 (1.00) |
0.0279 (1.00) |
0.0665 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| DSPP | 9 (15%) | 53 |
0.98 (1.00) |
0.412 (1.00) |
0.758 (1.00) |
1 (1.00) |
0.224 (1.00) |
0.471 (1.00) |
1 (1.00) |
| AMDHD1 | 10 (16%) | 52 |
0.848 (1.00) |
0.0434 (1.00) |
0.826 (1.00) |
1 (1.00) |
0.577 (1.00) |
0.307 (1.00) |
0.511 (1.00) |
| CRIPAK | 10 (16%) | 52 |
0.689 (1.00) |
0.116 (1.00) |
0.0923 (1.00) |
0.306 (1.00) |
0.577 (1.00) |
1 (1.00) |
1 (1.00) |
| NOM1 | 6 (10%) | 56 |
0.895 (1.00) |
0.625 (1.00) |
0.215 (1.00) |
0.266 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| MAP7 | 3 (5%) | 59 |
0.97 (1.00) |
0.73 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| LRP11 | 6 (10%) | 56 |
0.607 (1.00) |
0.497 (1.00) |
0.636 (1.00) |
1 (1.00) |
1 (1.00) |
0.657 (1.00) |
0.559 (1.00) |
| PDCD6 | 3 (5%) | 59 |
0.919 (1.00) |
0.0879 (1.00) |
0.0106 (1.00) |
0.00491 (1.00) |
0.279 (1.00) |
1 (1.00) |
0.206 (1.00) |
| SHOX2 | 3 (5%) | 59 |
0.226 (1.00) |
0.896 (1.00) |
1 (1.00) |
0.787 (1.00) |
0.279 (1.00) |
1 (1.00) |
1 (1.00) |
| UQCRFS1 | 5 (8%) | 57 |
0.182 (1.00) |
0.0493 (1.00) |
0.0389 (1.00) |
0.0223 (1.00) |
0.427 (1.00) |
1 (1.00) |
0.18 (1.00) |
| CTBP2 | 3 (5%) | 59 |
0.942 (1.00) |
0.244 (1.00) |
0.397 (1.00) |
0.169 (1.00) |
1 (1.00) |
1 (1.00) |
0.511 (1.00) |
| PRKAR1A | 6 (10%) | 56 |
0.807 (1.00) |
0.83 (1.00) |
0.0701 (1.00) |
0.0681 (1.00) |
0.49 (1.00) |
0.0808 (1.00) |
0.374 (1.00) |
| JMJD4 | 3 (5%) | 59 |
0.757 (1.00) |
0.844 (1.00) |
1 (1.00) |
0.632 (1.00) |
1 (1.00) |
0.285 (1.00) |
0.374 (1.00) |
| HSD17B1 | 4 (6%) | 58 |
0.638 (1.00) |
0.596 (1.00) |
0.192 (1.00) |
0.239 (1.00) |
1 (1.00) |
1 (1.00) |
0.374 (1.00) |
| COQ2 | 5 (8%) | 57 |
0.962 (1.00) |
0.124 (1.00) |
0.479 (1.00) |
0.711 (1.00) |
1 (1.00) |
0.647 (1.00) |
1 (1.00) |
| FEZ2 | 3 (5%) | 59 |
0.7 (1.00) |
0.491 (1.00) |
0.702 (1.00) |
0.787 (1.00) |
0.279 (1.00) |
1 (1.00) |
1 (1.00) |
| DLEU7 | 3 (5%) | 59 |
0.611 (1.00) |
0.705 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
|
| RNF135 | 3 (5%) | 59 |
0.226 (1.00) |
0.831 (1.00) |
0.577 (1.00) |
0.784 (1.00) |
1 (1.00) |
1 (1.00) |
0.511 (1.00) |
| SPIRE2 | 3 (5%) | 59 |
0.936 (1.00) |
0.0637 (1.00) |
0.397 (1.00) |
0.167 (1.00) |
1 (1.00) |
0.546 (1.00) |
0.206 (1.00) |
| KRTAP5-5 | 3 (5%) | 59 |
0.314 (1.00) |
0.0661 (1.00) |
0.706 (1.00) |
1 (1.00) |
1 (1.00) |
0.285 (1.00) |
1 (1.00) |
| SGK223 | 3 (5%) | 59 |
0.747 (1.00) |
0.46 (1.00) |
0.477 (1.00) |
0.527 (1.00) |
1 (1.00) |
0.546 (1.00) |
0.374 (1.00) |
| C9ORF66 | 5 (8%) | 57 |
0.13 (1.00) |
0.359 (1.00) |
0.437 (1.00) |
0.563 (1.00) |
1 (1.00) |
0.337 (1.00) |
0.0374 (1.00) |
| AQP7 | 3 (5%) | 59 |
0.747 (1.00) |
0.0435 (1.00) |
0.576 (1.00) |
0.787 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| ADAMTS7 | 4 (6%) | 58 |
0.172 (1.00) |
0.129 (1.00) |
0.237 (1.00) |
0.396 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| PCDHB13 | 5 (8%) | 57 |
0.669 (1.00) |
0.127 (1.00) |
0.36 (1.00) |
0.214 (1.00) |
1 (1.00) |
1 (1.00) |
0.101 (1.00) |
| CSGALNACT2 | 3 (5%) | 59 |
0.835 (1.00) |
0.706 (1.00) |
0.704 (1.00) |
1 (1.00) |
1 (1.00) |
0.546 (1.00) |
1 (1.00) |
| HLA-A | 3 (5%) | 59 |
0.354 (1.00) |
0.225 (1.00) |
1 (1.00) |
0.633 (1.00) |
1 (1.00) |
0.285 (1.00) |
0.206 (1.00) |
| MTFMT | 3 (5%) | 59 |
0.777 (1.00) |
0.577 (1.00) |
0.705 (1.00) |
1 (1.00) |
1 (1.00) |
0.0407 (1.00) |
1 (1.00) |
| NLRP5 | 4 (6%) | 58 |
0.924 (1.00) |
0.053 (1.00) |
0.487 (1.00) |
0.196 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
| KCNJ11 | 4 (6%) | 58 |
0.459 (1.00) |
0.414 (1.00) |
0.0376 (1.00) |
0.74 (1.00) |
0.0478 (1.00) |
0.61 (1.00) |
1 (1.00) |
P value = 5.29e-05 (logrank test), Q value = 0.044
Table S1. Gene #95: 'PTX4 MUTATION STATUS' versus Clinical Feature #1: 'Time to Death'
| nPatients | nDeath | Duration Range (Median), Month | |
|---|---|---|---|
| ALL | 61 | 21 | 4.1 - 153.6 (41.4) |
| PTX4 MUTATED | 3 | 3 | 5.2 - 30.3 (18.1) |
| PTX4 WILD-TYPE | 58 | 18 | 4.1 - 153.6 (42.9) |
Figure S1. Get High-res Image Gene #95: 'PTX4 MUTATION STATUS' versus Clinical Feature #1: 'Time to Death'
-
Mutation data file = transformed.cor.cli.txt
-
Clinical data file = ACC-TP.merged_data.txt
-
Number of patients = 62
-
Number of significantly mutated genes = 119
-
Number of selected clinical features = 7
-
Exclude genes that fewer than K tumors have mutations, K = 3
For survival clinical features, the Kaplan-Meier survival curves of tumors with and without gene mutations were plotted and the statistical significance P values were estimated by logrank test (Bland and Altman 2004) using the 'survdiff' function in R
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
In addition to the links below, the full results of the analysis summarized in this report can also be downloaded programmatically using firehose_get, or interactively from either the Broad GDAC website or TCGA Data Coordination Center Portal.