(NRAS_Hotspot_Mutants cohort)
This pipeline computes the correlation between significantly recurrent gene mutations and molecular subtypes.
Testing the association between mutation status of 106 genes and 8 molecular subtypes across 62 patients, one significant finding detected with P value < 0.05 and Q value < 0.25.
-
HHLA2 mutation correlated to 'RPPA_CHIERARCHICAL'.
Table 1. Get Full Table Overview of the association between mutation status of 106 genes and 8 molecular subtypes. Shown in the table are P values (Q values). Thresholded by P value < 0.05 and Q value < 0.25, one significant finding detected.
|
Clinical Features |
CN CNMF |
METHLYATION CNMF |
RPPA CNMF |
RPPA CHIERARCHICAL |
MRNASEQ CNMF |
MRNASEQ CHIERARCHICAL |
MIRSEQ CNMF |
MIRSEQ CHIERARCHICAL |
||
| nMutated (%) | nWild-Type | Fisher's exact test | Fisher's exact test | Fisher's exact test | Chi-square test | Fisher's exact test | Fisher's exact test | Fisher's exact test | Fisher's exact test | |
| HHLA2 | 6 (10%) | 56 |
0.556 (1.00) |
0.0818 (1.00) |
0.0471 (1.00) |
0.000135 (0.114) |
0.485 (1.00) |
0.211 (1.00) |
0.00182 (1.00) |
0.0409 (1.00) |
| CDKN2A | 12 (19%) | 50 |
0.0101 (1.00) |
0.849 (1.00) |
0.695 (1.00) |
0.323 (1.00) |
0.266 (1.00) |
0.507 (1.00) |
0.888 (1.00) |
0.887 (1.00) |
| TP53 | 13 (21%) | 49 |
0.368 (1.00) |
0.0276 (1.00) |
1 (1.00) |
0.0752 (1.00) |
0.927 (1.00) |
0.481 (1.00) |
0.533 (1.00) |
0.0619 (1.00) |
| MUM1L1 | 13 (21%) | 49 |
0.737 (1.00) |
0.629 (1.00) |
0.235 (1.00) |
0.322 (1.00) |
0.113 (1.00) |
0.0517 (1.00) |
0.266 (1.00) |
0.805 (1.00) |
| CD163L1 | 21 (34%) | 41 |
0.837 (1.00) |
0.0213 (1.00) |
1 (1.00) |
0.209 (1.00) |
0.792 (1.00) |
0.655 (1.00) |
0.289 (1.00) |
0.847 (1.00) |
| PSG4 | 12 (19%) | 50 |
0.11 (1.00) |
1 (1.00) |
1 (1.00) |
0.37 (1.00) |
0.778 (1.00) |
0.772 (1.00) |
0.243 (1.00) |
0.389 (1.00) |
| PPP6C | 10 (16%) | 52 |
0.822 (1.00) |
0.688 (1.00) |
0.605 (1.00) |
0.22 (1.00) |
0.624 (1.00) |
0.613 (1.00) |
0.469 (1.00) |
0.436 (1.00) |
| ZNF99 | 16 (26%) | 46 |
0.201 (1.00) |
0.817 (1.00) |
0.32 (1.00) |
0.196 (1.00) |
0.0577 (1.00) |
0.702 (1.00) |
0.675 (1.00) |
0.671 (1.00) |
| KHDRBS2 | 10 (16%) | 52 |
0.287 (1.00) |
0.234 (1.00) |
0.127 (1.00) |
0.699 (1.00) |
0.15 (1.00) |
0.0596 (1.00) |
0.456 (1.00) |
0.592 (1.00) |
| POTEG | 10 (16%) | 52 |
0.179 (1.00) |
0.624 (1.00) |
0.695 (1.00) |
0.0969 (1.00) |
0.624 (1.00) |
0.496 (1.00) |
0.173 (1.00) |
0.673 (1.00) |
| MARCO | 13 (21%) | 49 |
0.107 (1.00) |
0.425 (1.00) |
0.451 (1.00) |
0.997 (1.00) |
0.104 (1.00) |
0.0606 (1.00) |
0.332 (1.00) |
0.294 (1.00) |
| SGCZ | 9 (15%) | 53 |
0.804 (1.00) |
0.354 (1.00) |
0.0202 (1.00) |
0.578 (1.00) |
1 (1.00) |
0.656 (1.00) |
0.741 (1.00) |
1 (1.00) |
| GLRB | 10 (16%) | 52 |
0.775 (1.00) |
0.111 (1.00) |
0.407 (1.00) |
0.141 (1.00) |
0.428 (1.00) |
0.904 (1.00) |
0.619 (1.00) |
0.512 (1.00) |
| OR2W1 | 8 (13%) | 54 |
0.0495 (1.00) |
0.079 (1.00) |
1 (1.00) |
0.655 (1.00) |
0.89 (1.00) |
0.89 (1.00) |
0.126 (1.00) |
1 (1.00) |
| C4ORF22 | 7 (11%) | 55 |
0.712 (1.00) |
1 (1.00) |
0.106 (1.00) |
0.261 (1.00) |
0.185 (1.00) |
0.883 (1.00) |
0.946 (1.00) |
0.581 (1.00) |
| UGT2B28 | 11 (18%) | 51 |
0.792 (1.00) |
0.177 (1.00) |
0.695 (1.00) |
0.456 (1.00) |
0.778 (1.00) |
0.487 (1.00) |
0.209 (1.00) |
0.768 (1.00) |
| ZNF676 | 11 (18%) | 51 |
0.102 (1.00) |
0.648 (1.00) |
0.695 (1.00) |
0.559 (1.00) |
0.778 (1.00) |
1 (1.00) |
0.909 (1.00) |
0.299 (1.00) |
| LIPI | 9 (15%) | 53 |
0.833 (1.00) |
1 (1.00) |
1 (1.00) |
0.589 (1.00) |
0.185 (1.00) |
1 (1.00) |
0.565 (1.00) |
0.859 (1.00) |
| CADM2 | 8 (13%) | 54 |
0.214 (1.00) |
0.358 (1.00) |
1 (1.00) |
0.731 (1.00) |
0.726 (1.00) |
1 (1.00) |
0.33 (1.00) |
0.0173 (1.00) |
| CDH9 | 11 (18%) | 51 |
0.938 (1.00) |
0.111 (1.00) |
0.695 (1.00) |
0.37 (1.00) |
0.415 (1.00) |
0.487 (1.00) |
0.7 (1.00) |
0.683 (1.00) |
| OR52E2 | 7 (11%) | 55 |
0.0374 (1.00) |
0.78 (1.00) |
0.661 (1.00) |
0.815 (1.00) |
1 (1.00) |
0.677 (1.00) |
1 (1.00) |
0.712 (1.00) |
| CLEC5A | 5 (8%) | 57 |
0.233 (1.00) |
0.509 (1.00) |
0.106 (1.00) |
0.144 (1.00) |
0.246 (1.00) |
0.155 (1.00) |
0.174 (1.00) |
1 (1.00) |
| HIST1H2AA | 5 (8%) | 57 |
1 (1.00) |
0.612 (1.00) |
1 (1.00) |
0.769 (1.00) |
0.729 (1.00) |
0.439 (1.00) |
0.277 (1.00) |
0.117 (1.00) |
| KIAA1257 | 8 (13%) | 54 |
0.75 (1.00) |
0.451 (1.00) |
0.0202 (1.00) |
0.578 (1.00) |
0.726 (1.00) |
1 (1.00) |
0.615 (1.00) |
0.728 (1.00) |
| PCDH18 | 16 (26%) | 46 |
0.477 (1.00) |
0.936 (1.00) |
0.48 (1.00) |
0.372 (1.00) |
0.154 (1.00) |
0.618 (1.00) |
0.955 (1.00) |
0.3 (1.00) |
| SPOCK3 | 9 (15%) | 53 |
0.101 (1.00) |
0.146 (1.00) |
0.407 (1.00) |
0.281 (1.00) |
0.73 (1.00) |
0.537 (1.00) |
0.186 (1.00) |
0.338 (1.00) |
| OR2G6 | 10 (16%) | 52 |
0.799 (1.00) |
0.0163 (1.00) |
1 (1.00) |
0.256 (1.00) |
0.75 (1.00) |
0.613 (1.00) |
0.964 (1.00) |
0.322 (1.00) |
| STXBP5L | 14 (23%) | 48 |
0.472 (1.00) |
0.303 (1.00) |
0.48 (1.00) |
0.913 (1.00) |
0.513 (1.00) |
0.296 (1.00) |
0.323 (1.00) |
0.902 (1.00) |
| CNGB3 | 15 (24%) | 47 |
0.659 (1.00) |
0.754 (1.00) |
0.00836 (1.00) |
0.198 (1.00) |
0.454 (1.00) |
0.188 (1.00) |
0.93 (1.00) |
0.231 (1.00) |
| HHIPL2 | 4 (6%) | 58 |
0.354 (1.00) |
0.139 (1.00) |
1 (1.00) |
0.545 (1.00) |
0.668 (1.00) |
0.909 (1.00) |
1 (1.00) |
|
| OR10G8 | 6 (10%) | 56 |
0.0137 (1.00) |
0.145 (1.00) |
1 (1.00) |
0.839 (1.00) |
0.0149 (1.00) |
0.132 (1.00) |
0.621 (1.00) |
0.668 (1.00) |
| UGT2B4 | 11 (18%) | 51 |
0.91 (1.00) |
0.493 (1.00) |
1 (1.00) |
0.677 (1.00) |
0.59 (1.00) |
0.224 (1.00) |
0.0717 (1.00) |
1 (1.00) |
| ZBBX | 11 (18%) | 51 |
0.507 (1.00) |
1 (1.00) |
1 (1.00) |
0.524 (1.00) |
0.529 (1.00) |
0.185 (1.00) |
0.232 (1.00) |
1 (1.00) |
| C1QTNF9 | 6 (10%) | 56 |
0.213 (1.00) |
0.759 (1.00) |
0.487 (1.00) |
0.871 (1.00) |
1 (1.00) |
0.933 (1.00) |
0.249 (1.00) |
|
| HCRTR2 | 9 (15%) | 53 |
0.53 (1.00) |
0.0395 (1.00) |
1 (1.00) |
0.642 (1.00) |
0.73 (1.00) |
0.537 (1.00) |
0.0606 (1.00) |
0.338 (1.00) |
| KRT78 | 7 (11%) | 55 |
0.903 (1.00) |
0.102 (1.00) |
1 (1.00) |
0.769 (1.00) |
0.469 (1.00) |
0.883 (1.00) |
0.646 (1.00) |
0.079 (1.00) |
| BCLAF1 | 14 (23%) | 48 |
0.766 (1.00) |
0.329 (1.00) |
0.716 (1.00) |
0.914 (1.00) |
0.865 (1.00) |
0.393 (1.00) |
0.318 (1.00) |
0.513 (1.00) |
| LPAR1 | 6 (10%) | 56 |
0.458 (1.00) |
0.194 (1.00) |
1 (1.00) |
0.655 (1.00) |
0.157 (1.00) |
0.0991 (1.00) |
0.148 (1.00) |
0.408 (1.00) |
| STK31 | 10 (16%) | 52 |
0.139 (1.00) |
0.234 (1.00) |
0.605 (1.00) |
0.175 (1.00) |
0.167 (1.00) |
0.496 (1.00) |
0.863 (1.00) |
1 (1.00) |
| HBG2 | 4 (6%) | 58 |
0.423 (1.00) |
1 (1.00) |
1 (1.00) |
0.0153 (1.00) |
0.83 (1.00) |
1 (1.00) |
0.909 (1.00) |
1 (1.00) |
| BAGE2 | 4 (6%) | 58 |
0.541 (1.00) |
0.829 (1.00) |
0.487 (1.00) |
0.0881 (1.00) |
0.0331 (1.00) |
0.384 (1.00) |
0.407 (1.00) |
|
| ADH1B | 6 (10%) | 56 |
0.938 (1.00) |
0.421 (1.00) |
0.605 (1.00) |
0.636 (1.00) |
0.871 (1.00) |
1 (1.00) |
0.325 (1.00) |
0.668 (1.00) |
| CNTN4 | 12 (19%) | 50 |
0.438 (1.00) |
0.781 (1.00) |
0.716 (1.00) |
0.565 (1.00) |
0.923 (1.00) |
1 (1.00) |
1 (1.00) |
0.144 (1.00) |
| DEFA4 | 5 (8%) | 57 |
1 (1.00) |
0.363 (1.00) |
1 (1.00) |
0.607 (1.00) |
0.209 (1.00) |
0.155 (1.00) |
0.742 (1.00) |
1 (1.00) |
| FAM117B | 6 (10%) | 56 |
0.746 (1.00) |
0.868 (1.00) |
0.605 (1.00) |
0.162 (1.00) |
0.485 (1.00) |
0.633 (1.00) |
0.933 (1.00) |
1 (1.00) |
| GFRAL | 10 (16%) | 52 |
0.424 (1.00) |
0.624 (1.00) |
0.342 (1.00) |
0.105 (1.00) |
0.0312 (1.00) |
0.0325 (1.00) |
0.602 (1.00) |
0.867 (1.00) |
| ST8SIA3 | 8 (13%) | 54 |
0.0416 (1.00) |
0.403 (1.00) |
1 (1.00) |
0.169 (1.00) |
1 (1.00) |
0.712 (1.00) |
0.249 (1.00) |
1 (1.00) |
| DSPP | 9 (15%) | 53 |
0.288 (1.00) |
0.165 (1.00) |
1 (1.00) |
0.366 (1.00) |
0.73 (1.00) |
0.537 (1.00) |
0.17 (1.00) |
0.859 (1.00) |
| OR10K2 | 8 (13%) | 54 |
0.334 (1.00) |
0.893 (1.00) |
0.661 (1.00) |
0.729 (1.00) |
0.359 (1.00) |
0.193 (1.00) |
0.377 (1.00) |
0.607 (1.00) |
| CLEC17A | 5 (8%) | 57 |
0.328 (1.00) |
0.212 (1.00) |
1 (1.00) |
0.435 (1.00) |
0.176 (1.00) |
0.439 (1.00) |
1 (1.00) |
0.47 (1.00) |
| POU6F2 | 9 (15%) | 53 |
0.127 (1.00) |
0.283 (1.00) |
1 (1.00) |
0.497 (1.00) |
1 (1.00) |
1 (1.00) |
0.922 (1.00) |
0.13 (1.00) |
| LRRIQ3 | 10 (16%) | 52 |
0.498 (1.00) |
0.83 (1.00) |
1 (1.00) |
0.534 (1.00) |
0.0672 (1.00) |
0.023 (1.00) |
0.964 (1.00) |
1 (1.00) |
| ARID2 | 11 (18%) | 51 |
0.814 (1.00) |
1 (1.00) |
1 (1.00) |
0.304 (1.00) |
0.529 (1.00) |
0.224 (1.00) |
0.665 (1.00) |
0.355 (1.00) |
| GAS2 | 7 (11%) | 55 |
0.819 (1.00) |
0.78 (1.00) |
1 (1.00) |
0.814 (1.00) |
0.604 (1.00) |
1 (1.00) |
0.743 (1.00) |
0.111 (1.00) |
| SIRPB1 | 6 (10%) | 56 |
0.121 (1.00) |
0.759 (1.00) |
1 (1.00) |
0.731 (1.00) |
0.65 (1.00) |
1 (1.00) |
0.325 (1.00) |
0.249 (1.00) |
| ACTC1 | 6 (10%) | 56 |
0.788 (1.00) |
0.224 (1.00) |
0.605 (1.00) |
0.191 (1.00) |
0.365 (1.00) |
0.633 (1.00) |
0.531 (1.00) |
0.408 (1.00) |
| PRB2 | 9 (15%) | 53 |
0.32 (1.00) |
1 (1.00) |
1 (1.00) |
0.0612 (1.00) |
0.545 (1.00) |
0.537 (1.00) |
0.859 (1.00) |
0.859 (1.00) |
| C15ORF23 | 4 (6%) | 58 |
0.259 (1.00) |
0.194 (1.00) |
1 (1.00) |
0.769 (1.00) |
1 (1.00) |
1 (1.00) |
0.102 (1.00) |
0.407 (1.00) |
| FASLG | 5 (8%) | 57 |
0.0507 (1.00) |
0.612 (1.00) |
1 (1.00) |
0.655 (1.00) |
1 (1.00) |
1 (1.00) |
0.094 (1.00) |
0.163 (1.00) |
| PAK7 | 12 (19%) | 50 |
0.577 (1.00) |
0.52 (1.00) |
0.407 (1.00) |
0.361 (1.00) |
0.565 (1.00) |
0.294 (1.00) |
0.0101 (1.00) |
0.254 (1.00) |
| SIGLEC14 | 6 (10%) | 56 |
0.192 (1.00) |
0.357 (1.00) |
1 (1.00) |
0.564 (1.00) |
0.746 (1.00) |
0.274 (1.00) |
0.408 (1.00) |
|
| TPTE2 | 8 (13%) | 54 |
0.601 (1.00) |
0.804 (1.00) |
0.661 (1.00) |
0.585 (1.00) |
0.439 (1.00) |
0.554 (1.00) |
0.418 (1.00) |
0.728 (1.00) |
| APBB1IP | 6 (10%) | 56 |
0.25 (1.00) |
1 (1.00) |
0.487 (1.00) |
1 (1.00) |
1 (1.00) |
0.56 (1.00) |
0.249 (1.00) |
|
| ANGPT1 | 8 (13%) | 54 |
0.956 (1.00) |
0.57 (1.00) |
0.605 (1.00) |
0.731 (1.00) |
0.253 (1.00) |
0.0642 (1.00) |
0.207 (1.00) |
1 (1.00) |
| ARL16 | 3 (5%) | 59 |
0.111 (1.00) |
1 (1.00) |
1 (1.00) |
0.839 (1.00) |
1 (1.00) |
0.787 (1.00) |
0.18 (1.00) |
0.263 (1.00) |
| ARMC4 | 15 (24%) | 47 |
0.808 (1.00) |
0.869 (1.00) |
1 (1.00) |
0.471 (1.00) |
0.259 (1.00) |
0.159 (1.00) |
0.377 (1.00) |
0.658 (1.00) |
| CD2 | 3 (5%) | 59 |
0.882 (1.00) |
0.282 (1.00) |
1 (1.00) |
0.625 (1.00) |
0.787 (1.00) |
0.271 (1.00) |
1 (1.00) |
|
| FRG2B | 5 (8%) | 57 |
0.158 (1.00) |
0.305 (1.00) |
1 (1.00) |
0.508 (1.00) |
0.176 (1.00) |
0.266 (1.00) |
0.859 (1.00) |
0.163 (1.00) |
| KRTAP12-1 | 3 (5%) | 59 |
0.78 (1.00) |
0.63 (1.00) |
0.487 (1.00) |
0.485 (1.00) |
0.117 (1.00) |
0.513 (1.00) |
1 (1.00) |
|
| CYP4X1 | 7 (11%) | 55 |
0.353 (1.00) |
0.418 (1.00) |
1 (1.00) |
0.0153 (1.00) |
0.779 (1.00) |
0.677 (1.00) |
0.743 (1.00) |
0.712 (1.00) |
| DSG1 | 14 (23%) | 48 |
0.952 (1.00) |
0.805 (1.00) |
0.695 (1.00) |
0.0749 (1.00) |
1 (1.00) |
1 (1.00) |
0.541 (1.00) |
0.902 (1.00) |
| ADAM7 | 13 (21%) | 49 |
0.292 (1.00) |
0.16 (1.00) |
0.182 (1.00) |
0.821 (1.00) |
0.0333 (1.00) |
0.172 (1.00) |
0.465 (1.00) |
0.558 (1.00) |
| KCNQ5 | 10 (16%) | 52 |
0.964 (1.00) |
0.0287 (1.00) |
1 (1.00) |
0.257 (1.00) |
0.312 (1.00) |
0.146 (1.00) |
0.508 (1.00) |
0.0317 (1.00) |
| POF1B | 10 (16%) | 52 |
0.901 (1.00) |
0.83 (1.00) |
1 (1.00) |
0.551 (1.00) |
1 (1.00) |
0.378 (1.00) |
0.678 (1.00) |
0.436 (1.00) |
| TCEB3C | 7 (11%) | 55 |
0.782 (1.00) |
0.206 (1.00) |
1 (1.00) |
0.169 (1.00) |
1 (1.00) |
0.883 (1.00) |
0.0122 (1.00) |
0.015 (1.00) |
| F2RL1 | 6 (10%) | 56 |
0.556 (1.00) |
1 (1.00) |
1 (1.00) |
0.602 (1.00) |
0.758 (1.00) |
1 (1.00) |
0.0289 (1.00) |
0.668 (1.00) |
| SNCAIP | 12 (19%) | 50 |
0.0659 (1.00) |
0.717 (1.00) |
1 (1.00) |
0.786 (1.00) |
0.287 (1.00) |
0.0735 (1.00) |
0.00343 (1.00) |
0.784 (1.00) |
| LOC649330 | 8 (13%) | 54 |
0.0738 (1.00) |
0.638 (1.00) |
1 (1.00) |
0.0417 (1.00) |
0.253 (1.00) |
0.712 (1.00) |
0.714 (1.00) |
1 (1.00) |
| UGT2A3 | 8 (13%) | 54 |
0.214 (1.00) |
0.507 (1.00) |
1 (1.00) |
0.701 (1.00) |
0.398 (1.00) |
0.0909 (1.00) |
0.0238 (1.00) |
0.102 (1.00) |
| C3ORF71 | 3 (5%) | 59 |
0.406 (1.00) |
0.382 (1.00) |
1 (1.00) |
0.342 (1.00) |
0.773 (1.00) |
0.787 (1.00) |
0.764 (1.00) |
0.157 (1.00) |
| NDUFA13 | 4 (6%) | 58 |
0.0885 (1.00) |
0.251 (1.00) |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.155 (1.00) |
0.407 (1.00) |
|
| ZIM3 | 8 (13%) | 54 |
0.0712 (1.00) |
0.02 (1.00) |
1 (1.00) |
0.0567 (1.00) |
0.359 (1.00) |
0.303 (1.00) |
0.714 (1.00) |
1 (1.00) |
| DDX3X | 7 (11%) | 55 |
0.903 (1.00) |
0.78 (1.00) |
0.106 (1.00) |
0.131 (1.00) |
0.211 (1.00) |
0.677 (1.00) |
0.438 (1.00) |
0.712 (1.00) |
| LILRB4 | 8 (13%) | 54 |
0.878 (1.00) |
1 (1.00) |
1 (1.00) |
0.783 (1.00) |
0.0764 (1.00) |
0.554 (1.00) |
0.0897 (1.00) |
0.859 (1.00) |
| TLR4 | 11 (18%) | 51 |
0.531 (1.00) |
0.288 (1.00) |
1 (1.00) |
0.642 (1.00) |
0.913 (1.00) |
0.402 (1.00) |
0.604 (1.00) |
0.417 (1.00) |
| ZNF479 | 5 (8%) | 57 |
0.486 (1.00) |
0.00447 (1.00) |
0.231 (1.00) |
0.0874 (1.00) |
0.618 (1.00) |
0.439 (1.00) |
0.0437 (1.00) |
0.0455 (1.00) |
| GABRG1 | 8 (13%) | 54 |
0.415 (1.00) |
0.57 (1.00) |
0.661 (1.00) |
0.357 (1.00) |
0.398 (1.00) |
0.303 (1.00) |
0.493 (1.00) |
0.728 (1.00) |
| KLRC3 | 4 (6%) | 58 |
0.702 (1.00) |
0.376 (1.00) |
0.605 (1.00) |
0.144 (1.00) |
1 (1.00) |
1 (1.00) |
0.137 (1.00) |
0.139 (1.00) |
| OR1N2 | 7 (11%) | 55 |
0.86 (1.00) |
0.18 (1.00) |
1 (1.00) |
0.201 (1.00) |
0.159 (1.00) |
0.141 (1.00) |
0.00455 (1.00) |
0.0573 (1.00) |
| SYCE1 | 6 (10%) | 56 |
0.343 (1.00) |
0.568 (1.00) |
1 (1.00) |
0.809 (1.00) |
0.0648 (1.00) |
0.489 (1.00) |
0.474 (1.00) |
0.518 (1.00) |
| RGPD3 | 12 (19%) | 50 |
0.371 (1.00) |
0.616 (1.00) |
0.182 (1.00) |
0.179 (1.00) |
0.435 (1.00) |
1 (1.00) |
0.481 (1.00) |
0.0647 (1.00) |
| CDH10 | 13 (21%) | 49 |
0.318 (1.00) |
0.859 (1.00) |
0.695 (1.00) |
0.253 (1.00) |
0.927 (1.00) |
1 (1.00) |
0.567 (1.00) |
0.805 (1.00) |
| C6ORF105 | 5 (8%) | 57 |
0.233 (1.00) |
1 (1.00) |
1 (1.00) |
0.359 (1.00) |
0.176 (1.00) |
0.266 (1.00) |
0.599 (1.00) |
0.354 (1.00) |
| FPR2 | 5 (8%) | 57 |
0.805 (1.00) |
0.727 (1.00) |
0.605 (1.00) |
0.58 (1.00) |
0.846 (1.00) |
0.625 (1.00) |
1 (1.00) |
0.163 (1.00) |
| UGT1A7 | 4 (6%) | 58 |
1 (1.00) |
1 (1.00) |
1 (1.00) |
0.658 (1.00) |
0.83 (1.00) |
0.668 (1.00) |
0.0607 (1.00) |
1 (1.00) |
| JAKMIP1 | 9 (15%) | 53 |
0.146 (1.00) |
0.491 (1.00) |
1 (1.00) |
0.377 (1.00) |
0.662 (1.00) |
1 (1.00) |
0.644 (1.00) |
0.649 (1.00) |
| CYLC2 | 6 (10%) | 56 |
0.343 (1.00) |
0.759 (1.00) |
1 (1.00) |
0.475 (1.00) |
0.365 (1.00) |
0.132 (1.00) |
0.00958 (1.00) |
0.135 (1.00) |
| DRGX | 6 (10%) | 56 |
0.529 (1.00) |
0.303 (1.00) |
1 (1.00) |
0.662 (1.00) |
0.65 (1.00) |
0.633 (1.00) |
1 (1.00) |
0.829 (1.00) |
| IDH1 | 5 (8%) | 57 |
0.307 (1.00) |
0.093 (1.00) |
1 (1.00) |
0.658 (1.00) |
0.0935 (1.00) |
1 (1.00) |
0.05 (1.00) |
0.0801 (1.00) |
| ASB5 | 6 (10%) | 56 |
0.613 (1.00) |
0.868 (1.00) |
1 (1.00) |
0.608 (1.00) |
0.424 (1.00) |
0.211 (1.00) |
0.157 (1.00) |
0.408 (1.00) |
| COL21A1 | 11 (18%) | 51 |
0.149 (1.00) |
0.843 (1.00) |
1 (1.00) |
0.921 (1.00) |
0.913 (1.00) |
0.402 (1.00) |
0.471 (1.00) |
0.615 (1.00) |
| OR4M2 | 7 (11%) | 55 |
0.603 (1.00) |
0.475 (1.00) |
1 (1.00) |
0.413 (1.00) |
0.779 (1.00) |
1 (1.00) |
0.946 (1.00) |
0.378 (1.00) |
| ANKRD45 | 7 (11%) | 55 |
0.0423 (1.00) |
0.537 (1.00) |
1 (1.00) |
0.601 (1.00) |
1 (1.00) |
0.453 (1.00) |
0.677 (1.00) |
0.198 (1.00) |
| LCE1B | 5 (8%) | 57 |
0.25 (1.00) |
0.852 (1.00) |
0.605 (1.00) |
0.692 (1.00) |
0.299 (1.00) |
1 (1.00) |
0.277 (1.00) |
0.354 (1.00) |
| MGAM | 21 (34%) | 41 |
0.837 (1.00) |
0.531 (1.00) |
1 (1.00) |
0.428 (1.00) |
0.29 (1.00) |
0.738 (1.00) |
0.805 (1.00) |
0.432 (1.00) |
| ACSM2B | 9 (15%) | 53 |
0.53 (1.00) |
0.545 (1.00) |
1 (1.00) |
0.913 (1.00) |
0.204 (1.00) |
0.326 (1.00) |
0.112 (1.00) |
0.75 (1.00) |
P value = 0.000135 (Chi-square test), Q value = 0.11
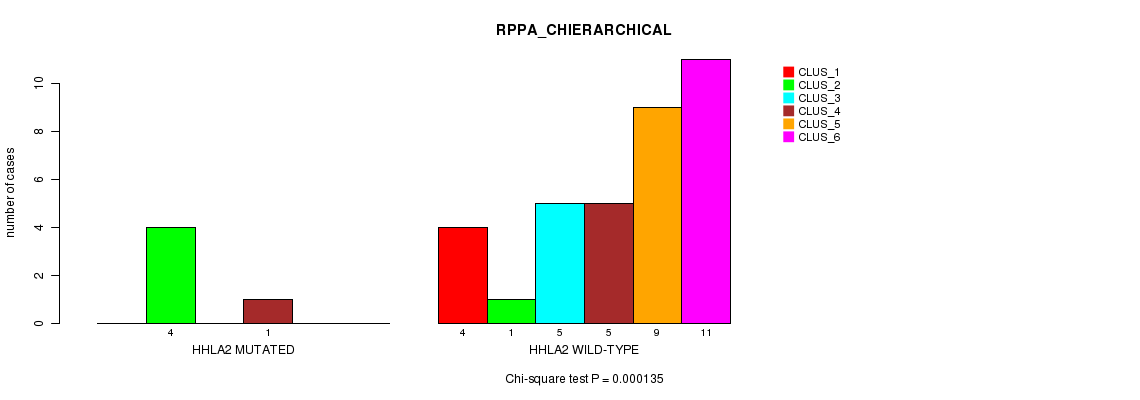
Table S1. Gene #89: 'HHLA2 MUTATION STATUS' versus Clinical Feature #4: 'RPPA_CHIERARCHICAL'
| nPatients | CLUS_1 | CLUS_2 | CLUS_3 | CLUS_4 | CLUS_5 | CLUS_6 |
|---|---|---|---|---|---|---|
| ALL | 4 | 5 | 5 | 6 | 9 | 11 |
| HHLA2 MUTATED | 0 | 4 | 0 | 1 | 0 | 0 |
| HHLA2 WILD-TYPE | 4 | 1 | 5 | 5 | 9 | 11 |
Figure S1. Get High-res Image Gene #89: 'HHLA2 MUTATION STATUS' versus Clinical Feature #4: 'RPPA_CHIERARCHICAL'
-
Mutation data file = SKCM-NRAS_Hotspot_Mutants.mutsig.cluster.txt
-
Molecular subtypes file = SKCM-NRAS_Hotspot_Mutants.transferedmergedcluster.txt
-
Number of patients = 62
-
Number of significantly mutated genes = 106
-
Number of Molecular subtypes = 8
-
Exclude genes that fewer than K tumors have mutations, K = 3
For binary or multi-class clinical features (nominal or ordinal), two-tailed Fisher's exact tests (Fisher 1922) were used to estimate the P values using the 'fisher.test' function in R
For multi-class clinical features (nominal or ordinal), Chi-square tests (Greenwood and Nikulin 1996) were used to estimate the P values using the 'chisq.test' function in R
For multiple hypothesis correction, Q value is the False Discovery Rate (FDR) analogue of the P value (Benjamini and Hochberg 1995), defined as the minimum FDR at which the test may be called significant. We used the 'Benjamini and Hochberg' method of 'p.adjust' function in R to convert P values into Q values.
This is an experimental feature. The full results of the analysis summarized in this report can be downloaded from the TCGA Data Coordination Center.